Практикум 12.
1. Сравните выравнивания одних и тех же последовательностей тремя разными программами MAFFT, MSAprobs, Tcoffee.
В качестве обьектов для выравнивания я выбрала белки из 9 практикума, так как уже делала с ними множественное выравнивание.(1.EX7L_JANMA 2.EX7L_ALKMQ 3.EX7L_BACCN 4.EX7L_STAA1 5.EX7L_BACP2).
Множественное выравнивание я делала с помощью программ: MSAprobs,Mafft,Tcoffee.
Для выполнения этого задания я пользовалась программой, написанной невероятной Ксенией Кирцовой. (Спасибо ей большое:3)
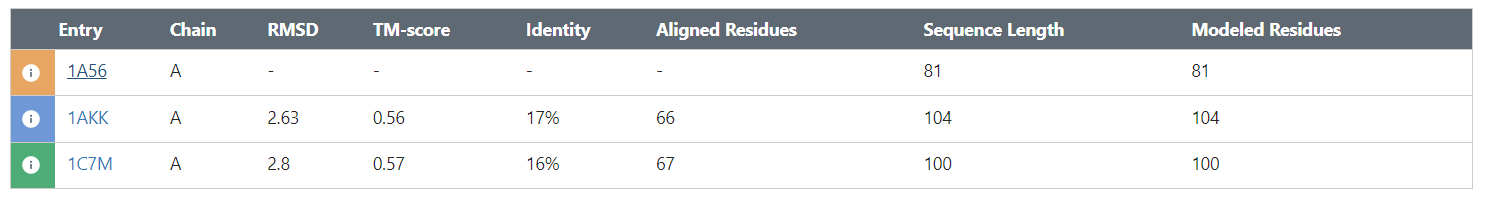
В качестве референсного выравнивания я выбралa MSAprobs, результаты сравнения которого с Tcoffee и MAFFT приведены в таблице 1 и 2.
| (s1,f1) | (s2,f2) | длина |
| (13-36) | (13-36) | 24 |
| (38) | (38) | 1 |
| (40-204) | (40-204) | 165 |
| (206-362) | (206-362) | 157 |
| (367-374) | (367-374) | 8 |
| (380-424) | (380-424) | 45 |
| (428-458) | (428-458) | 31 |
Таблица 1. Совпадающие блоки относительно MSAprobs и MAFFT:
| (s1,f1) | (s2,f2) | длина |
| (1) | (1) | 1 |
| (14-331) | (14-331) | 318 |
| (383-461) | (383-461) | 79 |
Таблица 2. Совпадающие блоки относительно MSAprobs и Tcoffee:
Длина выравнивания одинакова для всех: 471
Исходя из полученных данный мы можем сделать вывод, что алгоритмы работы программ MSAprobs и MAFFT более схожи, чем MSAprobs и Tcoffee, так как общая длина совпадающих колонок в выравнивании MSAprobs и MAFFT (431) больше чем общая длина совпадающих колонок MSAprobs и Tcoffee (398).
2.Построение выравнивания по совмещению структур и сравнение его с выравниванием программой MSA.
Для того, чтобы построить выравнивание 3D структур были выбраны 3 структуры из случайно выбранного семейства Cytochrome c (PF00034). Структуры:
С помощью Analyze ->Pairwise Structure Alignment на сайте PDB было получено парное выравнивание структур:
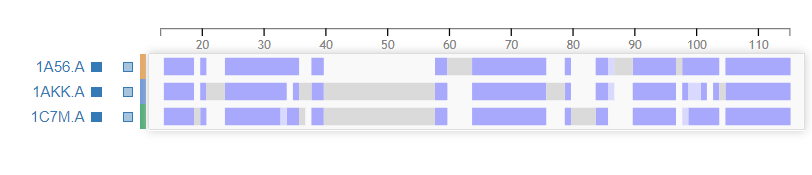
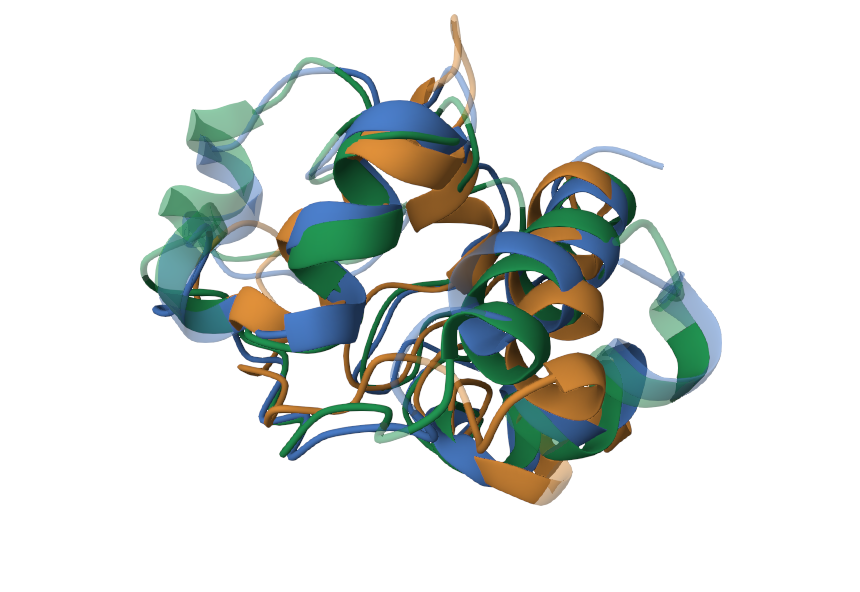
В качестве референсной структуры был 1a56.
Далее с помощью текстового редактора я сделала множественное выравнивание.
Далее я построила выравнивание в JalView (в проекте 2 выравнивания:jalView и PDB) этих трех структур с помощью программы MUSCLE with defaults. Далее посредством программы Ксении Кирцовой, можно увидеть совпадающие блоки относительно двух этих выравниваний:
| (s1,f1) | (s2,f2) | длина |
| (15-24) | (12-21) | 10 |
| (27-39) | (23-35) | 13 |
| (122-123) | (107-108) | 2 |
Таблица 3. Совпадающие блоки
Описание программы Mafft
MAFFT (Multiple Alignment using Fast Fourier Transform) - это программа с помощью которой можно осуществить множественное выравнивание последовательностей.
Основные этапы работы прогрпммы:
1.Предварительное выравнивание: В начале программа выравнивает все входные последовательности попарно с использованием оптимизированного алгоритма локального выравнивания!
2.Основное прогрессивное выравнивание: MAFFT использует модифицированный вариант классического алгоритма прогрессивного выравнивания. На каждом шаге две наиболее схожие группы последовательностей объединяются и выравниваются с использованием весовых матриц сумм пар (Weighted Sum-of-Pairs scoring matrices).
3.Итеративное уточнение выравнивания: Затем выравнивание уточняется засчет перемещения блоков в пределах выравнивания для максимизации суммарного веса. Это повторяется многократно.
4.Окончательная обработка: На финальном этапе MAFFT применяет дополнительные методы для дороботки выравнивания, например добавление пропусков и т д.