Практикум 10.
1. Консервативные мотивы в выравнивании.
Я выбрала домен Cellulase DNA gyrase B с идентификатором PF00204. В выравнивании seed этого домена содержится 46 последовательностей, Главная роль GyrB — АТФ-зависимое введение отрицательных супервитков в ДНК за счет гидролиза АТФ.
Далее я скачала это выравнивание и загрузила в JalView. После удалила последовательности, которые были на 90% идентичные, ничего не изменилось, то есть идентичные на 90% отсутствуют. Дальше покрасила Color Clustal и выставила Above identity threshold 100%. Снижала порог пока не выделился мотив, так как изначально были только изолированные колонки. Наиболее консервативным мне показался мотив на позициях 168-173 Паттерн мотива: [FY][EDTQASVN][GSN]Q[TVA][KQ]. Паттерн был найден во всех последовательностях 46/46.

Паттерн в формате Prosite: [FY]-[EDTQASVN]-[GSN]-Q-[TVA]-[KQ]. Далее я провела поиск по паттерну в формате Prosite в базе данных SwissProt, используя инструмент scanProsite(result) Found: 928 hits in 928 sequences. Судя по данным с UniProt данные белки выполняют разнве функции и принадлежат как прокариотам так и эукариотом, поэтому высока вероятность того, что находки случайны, и результат не имеет смысла.
Далее я сделала выравнивание с помощью Mafft. Оно получилось плохим, много пропуском, мотивы не выровнялись
2. Мотив, специфичный для одной клады филогенетического дерева.
Далее по выравниванию seed я построила дерево с помощью NJ в Jalview.
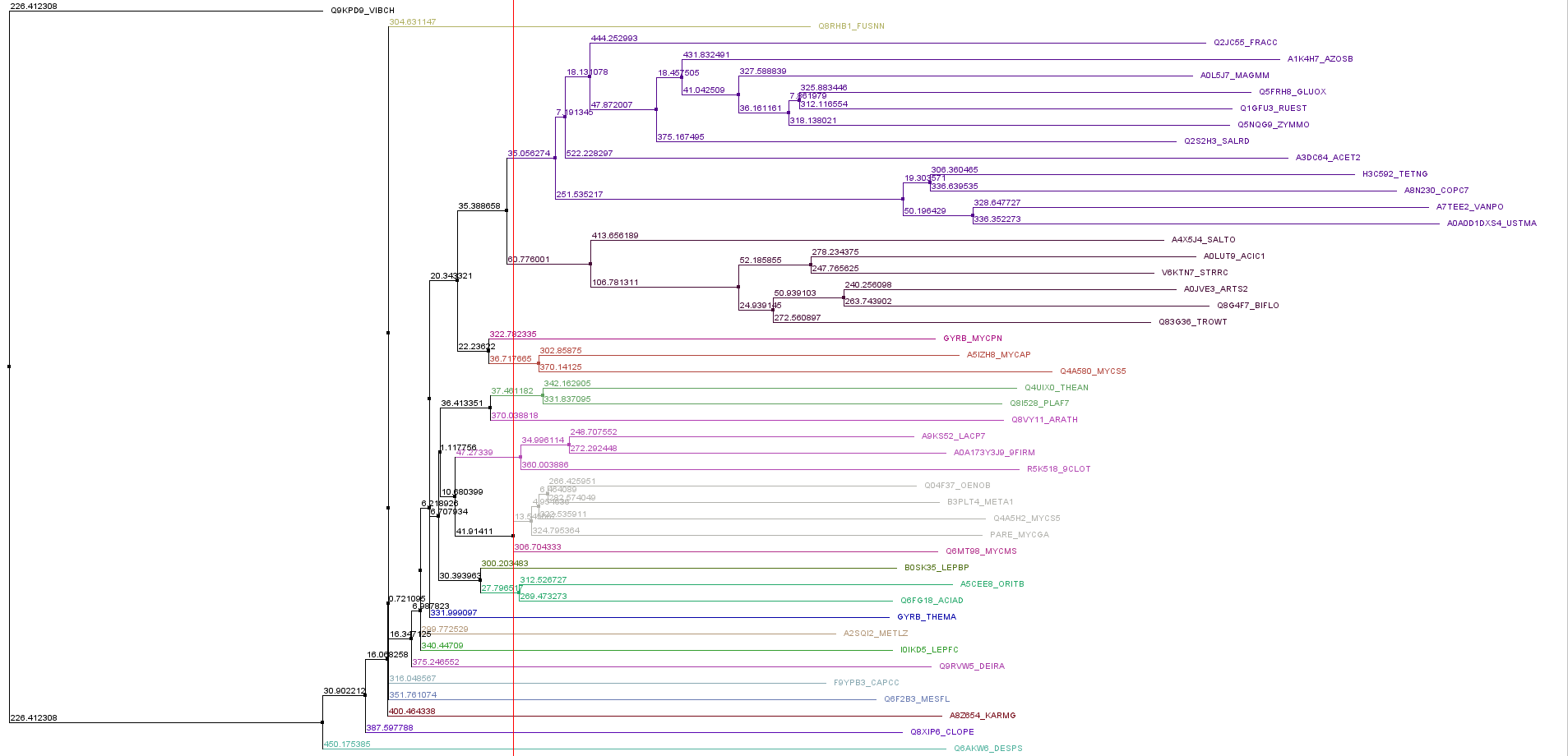
Далее я выбрала кладу (выделена фиолетовым) состоящую из 12 последовательностей и выделила в отдельное окно для поиска уникального консервативного мотива. Получился паттерн [TL]..GG.[HP] (168-173).
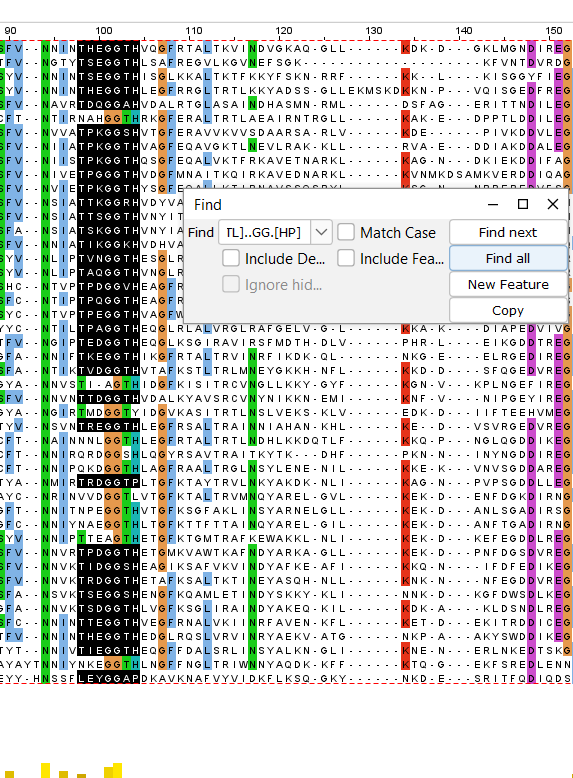
Паттерн нашелся в 35 последовательностях из 46, из чего можно сделать вывод, что он неспецифичен для данной клады.
3. PSI-BLAST.
Для этого задания я выбрала Q7VDL2. Это белок бактерии Prochlorococcus marinus, ингибитор деления клеток, блокирующий образование полярных перегородок Z-колец. Быстро колеблется между полюсами клетки, дестабилизируя филаменты FtsZ, которые сформировались до того, как созреют в полярные кольца Z. Предотвращает полимеризацию FtsZ.
PSI-BLAST анализ проводился по базе данных Swiss-Prot (порог E-value 0,005). Всего было 3 итерации, результаты представлены в таблице 1:
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 146 | Q9AG20.1 | 0,005 | A8GFG7.1 | 0,005 |
| 2 | 188 | B6JKX0.1 | 7,00E^-08 | - | - |
| 3 | 188 | Q9ZM51.1 | 2,00E^-12 | A7H8E6.1 | 0,014 |
Табл. 1. Таблица итерации для PSI-BLAST.
Из результатов можно сделать вывод о хорошем качестве собранного семейства гомологов тк PSI-BLAST сошёлся за три итерации, наблюдается чёткая "ступенька" E-value между худшей "правильной" находкой и "лучшей" неправильной.
4. MEME и FIMO.
Для моего домена были скачаны 166 последовательностей белков из SwissProt. Из этого числа я выбрала 120 последовательностей и запустила поиск мотивов с помощью программы MEME:
meme pr10_120.fasta -o results -mod anr -minw 4 -maxw 8 -nmotifs 4

Далее я запустила FIMO, чтобы провести поиск найденных мотивов в изначальном наборе из 166 последовательностей.
fimo results/meme.txt protein-matching-PF00204.fasta
Было найдено 422 вхождения (мотивов) в изначальные последовательности.
5. Оценка представленности сайта GATC в геноме бактерии.
В этом задании нужно было использовать геном "своей" бактерии, в моем случае это Erwinia amylovora CFBP1430. Для оценки представленности я исользовала программу cbcalc на kodomo. sites.txt - файл со всеми перестановками последовательности GATC(без повторений).
cbcalc -s sites.txt -M -o result.tsv sequence.fasta
На основе данных из выдачи с помощью python я построила гистограмма контрастов GATC
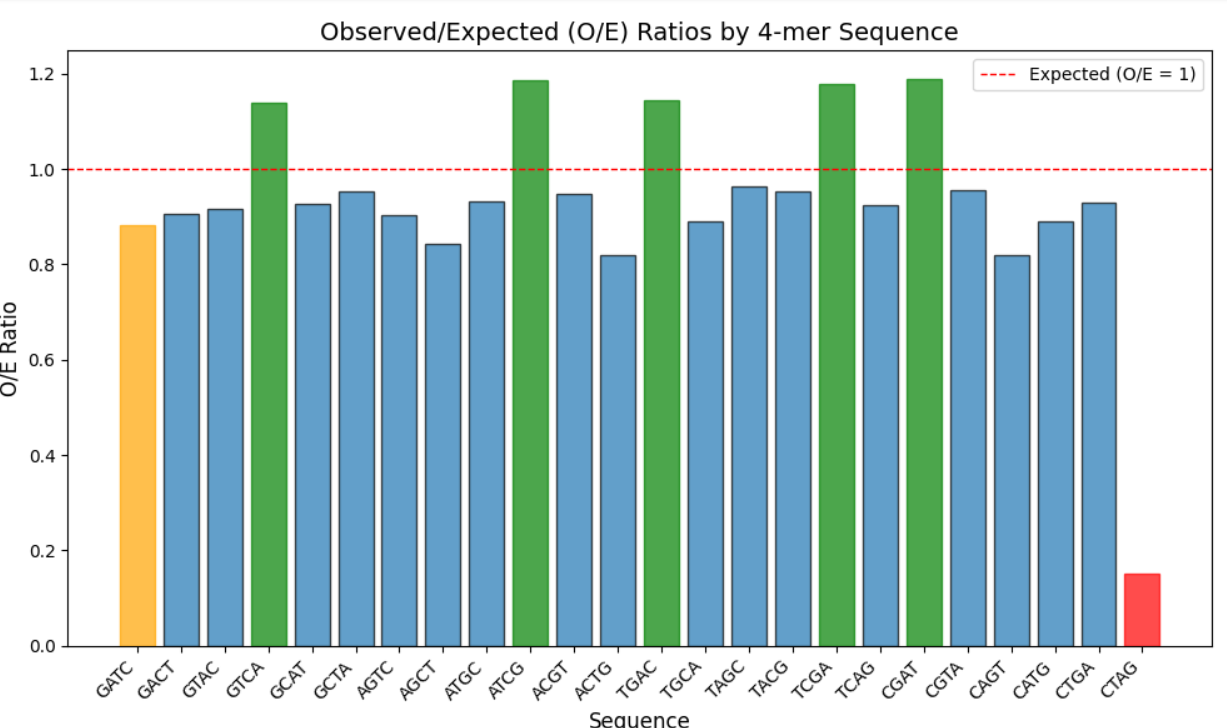
Как можно видеть на гистограмме представленность сайта метилирования GATC не очень высокая = 0.883. Возможно, сайтом метилирования является какой-то другой сайт.