Практикум 2.
1.Филогенетическая реконструкция и сравнение деревьев.
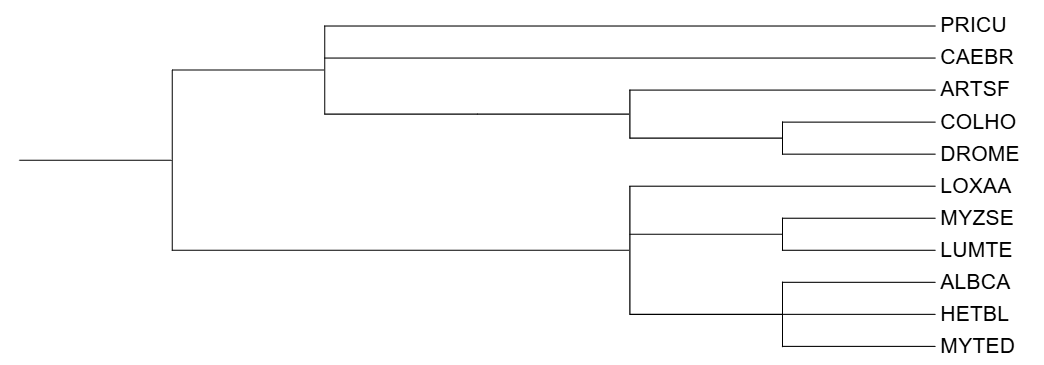



Давайте сравним полученное программами филогенетическое дерево с деревом, построенным в практикуме 1 с помощью NCBI Taxonomy. Отличий вышло довольно много.
Программа выдала, что Caenorhabditis briggsae(CAEBR) находится близко к Albinaria caerulea(ALBCA), однако судя по таксономии они относятся к разным кладам: Ecdysozoa(Линяющие) и Spiralia(Спиральные). Причем ALBCA относится к типу Mollusca, как MYTED и HETBL, также программа объединила вместе LOXAA и HETBL, хотя они относятся к разным типам. Скорее всего такие сильные различия связанны с самим алгоритмом программы, основанном на 'balanced minimum evolution'
Тут все примерно также, за исключением того, что программа объединила вместе Myzostoma seymourcollegiorum(MYZSE) и Heterololigo bleekeri(HETBL), судя по таксономии они относятся к одной кладе - Lophotrochozoa, однако к разным типам.