Практикум 3.
1. Реконструкция дерева по нуклеотидным последовательностям.
Для группы животных было построено дерево на основании последовательностей 12S рРНК. Не для всех видов нашлись их последовательности, поэтому необходимо было заменить их на другие виды, в качестве замен были выбраны максимально наиболее близкородственные организмы, единственной не совсем удачной заменой можно назвать Mytilus edulis -> Rhipicephalus sanguineus, т к они достаточно далеко друг от друга.
Выбраных животных и замену не подошедших можете посмотреть в файле.
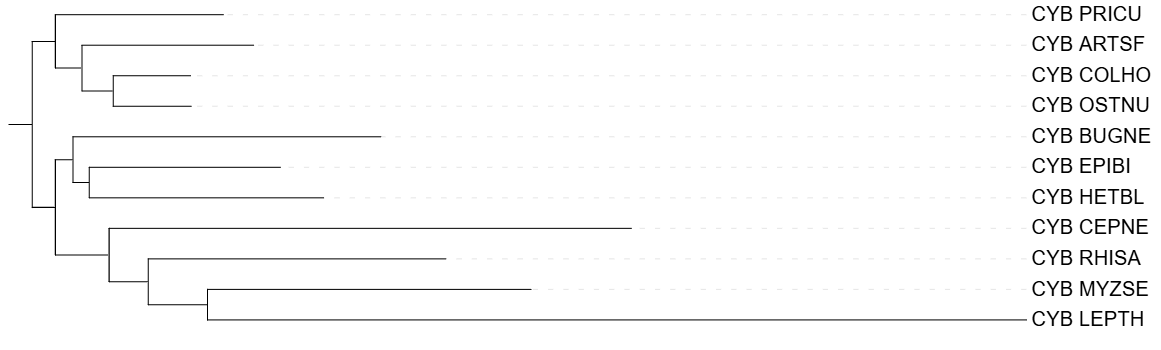
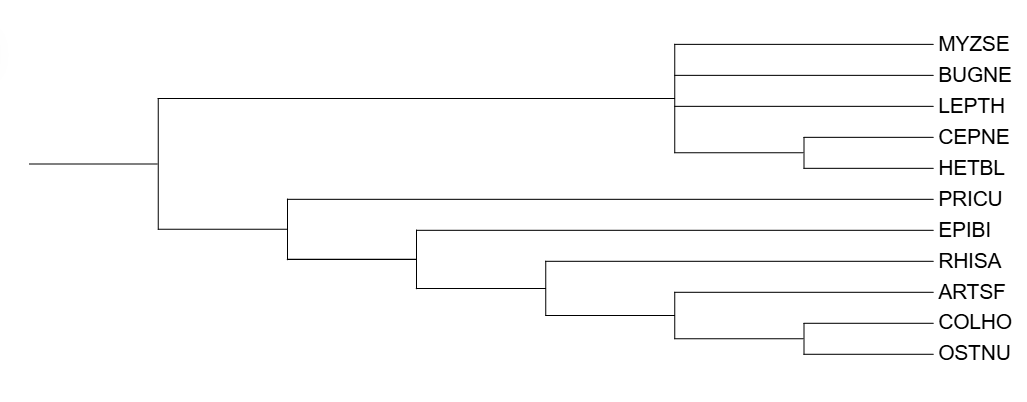
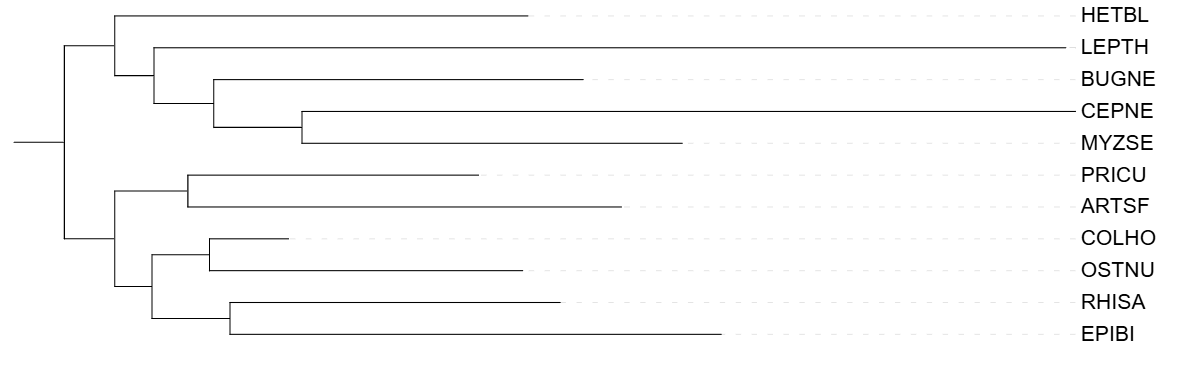
Сравнение деревьев NCBI Taxonomy и IQtree 12s.
Программа разделила Cepaea nemoralis(CEPNE) и Heterololigo bleekeri(HETBL), они относятся к одному типу Mollusca. Также она объединила вместе Rhipicephalus sanguineus(RHISA) и Epiperipatus biolleyi(EPIBI); Priapulus caudatus(PRICU) и Artemia franciscana(ARTSF).
Сравнение деревьев IQtree cyb b и IQtree 12s.
В отлтчии от IQtree 12s IQtree cyb b отделила EPIBI и RHISA от остальных животных, приндлежащих к Ecdysozoa, также она объединила MYZSE и LEPTH в одну группу, в целом дерево для 12s rRNA больше похоже на таксономическое.
2. Укоренение во внешнюю группу.
В качестве внешней группы я выбрала Felis catus(FELCA), относящуюся к Deuterostomia так как все выбранные мной организмы относятся к Protostomia.
Дерево не очень сильно изменилось и стало чуть больше похоже на NCBI Taxonomy, ARTSF и PRICU ушли в разные ветви, остались неверно сгруппированные клады:(CEPNE,MYZSE), (EPIBI,RHISA)
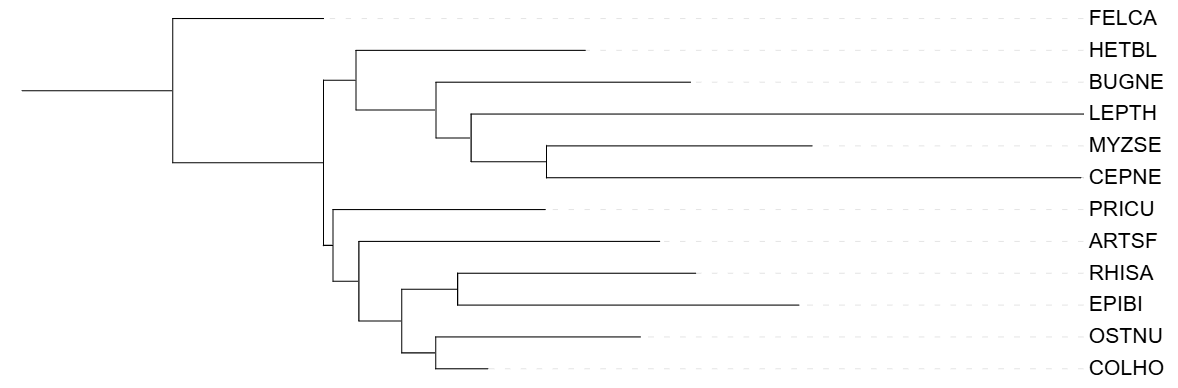
3. Бутстреп.
Для оценки достоверности построенных ветвей филогенетического дерева используется метод бутстрепа. В данном случае надо было использовать 100 реплик бутстрепа, что означает, что максимально возможная поддержка для ветви составляет 100. Чем ближе значение поддержки к 100, тем более надёжной считается соответствующая ветвь.
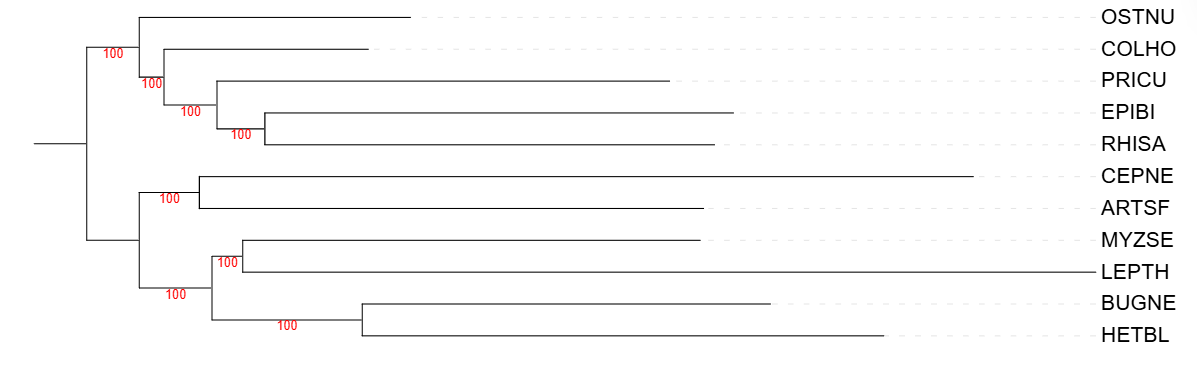
Дерево, созданное с помощью программы FastMe, значительно отличается от дерева, полученного в результате работы IQtree. На реконструированном дереве видно, что все ветви имеют высокие значения поддержки — 100, однако оно сильно отличается от эталонного. Для его построения была использована функция -r для удаления гэпов, так как без этого формируется неразрешенное дерево без клад. Однако поскольку 79% сайтов содержат гэпы, в итоге получились короткие последовательности. В результате оставшегося небольшого количества колонок метод Bootstrap сформировал деревья с максимальными уровнями поддержки.
