Практикум 4.
1. Составление списка гомологичных белков, включающих паралоги.
Для начала я выбрала протеомы бактерий из директории /P/y22/term4/Proteomes:
ACICJ, PROMH, ROSDO, RHIME, YERPE, THIDA, ECOLI.
Я скачала их в свою директорию с помощью команды:
cat ACICJ.fasta PROMH.fasta ROSDO.fasta RHIME.fasta YERPE.fasta THIDA.fasta ECOLI.fasta > ~/term4/pr4/id.fasta
Затем на основе выбранных протеомов я создала локальную базу данных для запуска blastp с помщью команды:
makeblastdb -in id.fasta -dbtype proteins
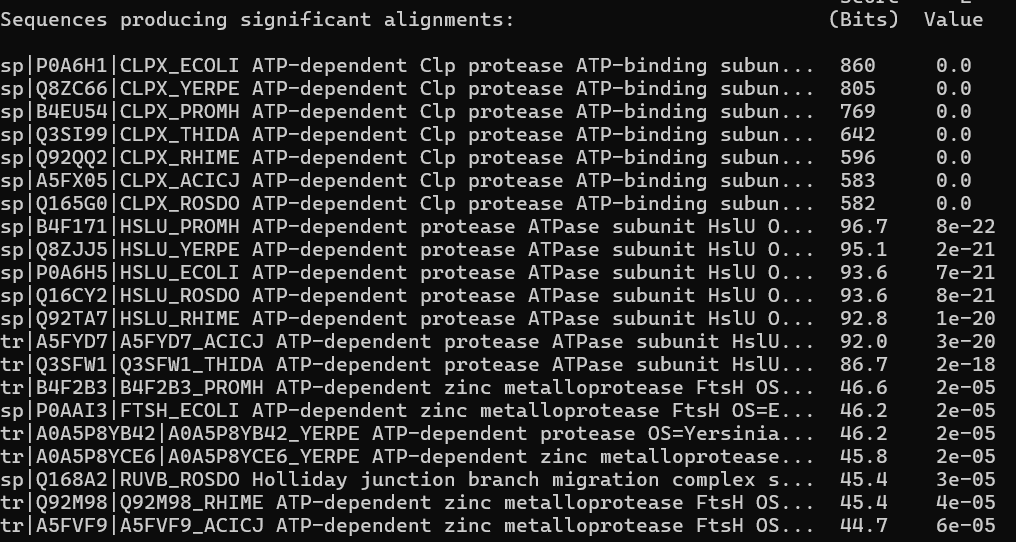
Затем запустила сам blastp, в качестве запроса была подана последовательность белка CLPX_ECOLI(мнемоника), порог E-value: 0.0001:
blastp -task blastp -query CLPX.fasta -db proteins -out blast.out -evalue 0.0001
Был получен следующий список находок.(см. рис. 1)
2.Реконструкция и визуализация.
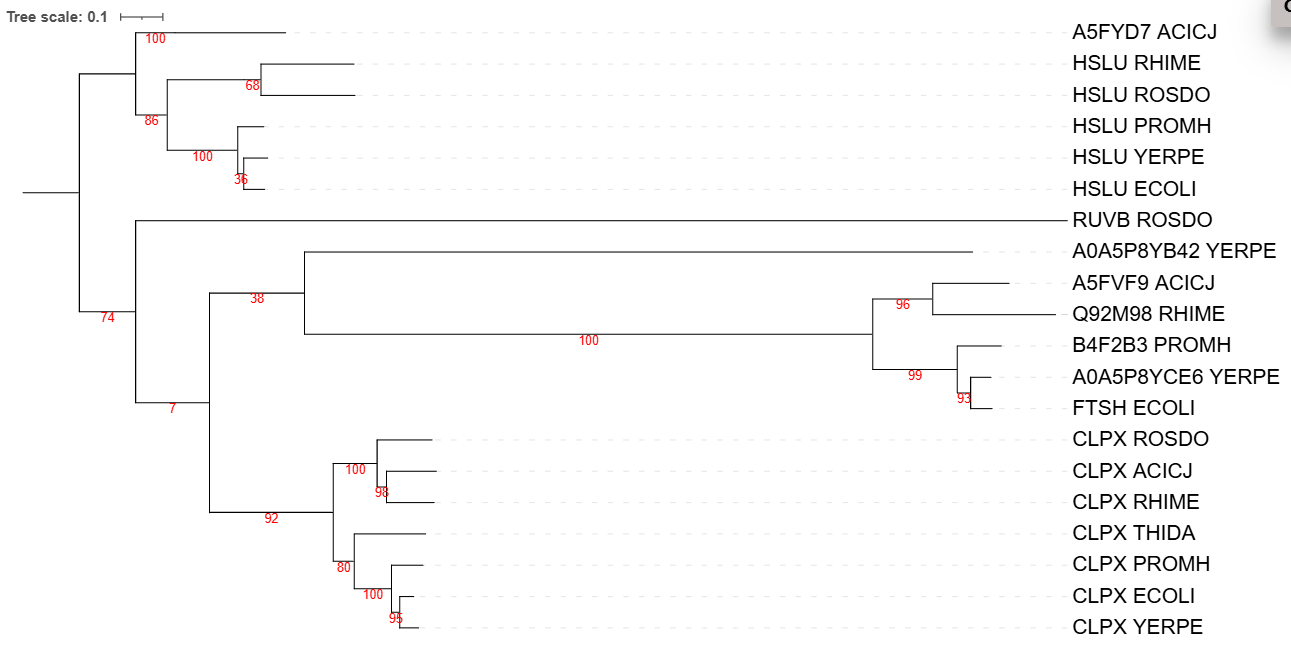
Далее я сделала выравнивание программой muscle, на основании которого после перевода в нужный формат .phy при помощи скрипта на питоне я построила дерево с помощью FastME, модель = MtREV, также была использована бутстреп-поддержка (100 реплик)
Формула дерева в формате Newick
Примеры ортологов: CLPX_ACICJ и CLPX_RHIME, HSLU_ROSDO и HSLU_RHIME, CLPX_ECOLI CLPX_YEPRE
Примеры паралогов: CLPX_RHIME и HSLU_RHIME, CLPX_ECOLI и HSLU_ECOLI, HSLU_ROSDO и CLPX_ROSDO
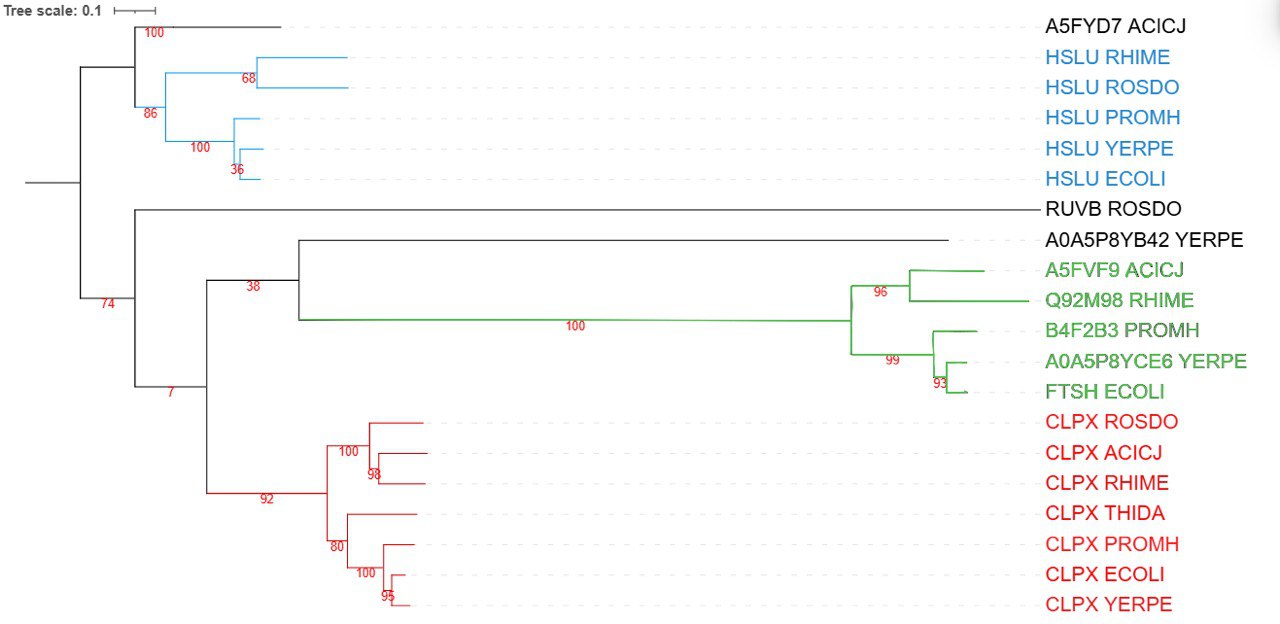

На рис.4 показаны схлопнутые ортологические группы, попробуем сравнить результат с эталонным деревом:
CLPX это компонент АТФ-зависимой специфичности протеазы Clp. АТФ-зависимые шапероны, которые также известны как разворачивающие ферментные системы, играют важную роль в процессе сворачивания и деградации белков. Они функционируют, связываясь с частично свернутыми или неправильно свернутыми белками и затем используя энергию АТФ для их разворачивания или деградации.
HSLU Субъединица АТФазы протеасомоподобного комплекса деградации; эта субъединица обладает шаперонной активностью. Связывание АТФ и его последующий гидролиз HslU необходимы для разворачивания белковых субстратов, впоследствии гидролизуемых HslV. HslU распознает N-концевую часть своих белковых субстратов и разворачивает их, прежде чем они направляются в HslV для гидролиза.
HSLU: ROSDO, RHIME, ECOLI, YERPE, PROMH. Cоответствует эталонному дереву.
Третья группа: RHIME, ECOLI, YERPE, PROMH, ACICJ. Cоответствует эталонному дереву.