Практикум 8.
1.Последовательность Shine-Dalgarno.
Для этого задания я выбрала Shine-Dalgarno sequence, Shine-Dalgarno box — сайт посадки рибосом на молекуле мРНК прокариот.
a. Название сигнала: Последовательность Shine-Dalgarno (SD)
b. Носитель сигнала: мРНК прокариот (бактерии, архей)
c. Кому адресован: Малая субъединица рибосомы. Комплементарной последовательности, называемой последовательностью анти-Шайна—Дальгарно, которая располагается на 3'-конце молекулы 16S рибосомной РНК.
d. Предназначение — как должен реагировать адресат:Комплементарное взаимодействие между последовательностями Шайна—Дальгарно и анти-Шайна—Дальгарно служит для помещения старт-кодона мРНК в P-сайт рибосомы для начала биосинтеза белка.
e. Сила сигнала: Сила сигнала может зависеть от:
Shine–Dalgarno — это последовательность, которая комплементарна к участку 3'-конца 16S рРНК рибосомы. Чем выше комплементарность, тем сильнее связывание и эффективнее инициация.
Оптимальное расстояние между SD-последовательностью и старт-кодоном составляет около 5–9 нуклеотидов. Если расстояние больше или меньше, эффективность связывания снижается.
Если SD-последовательность или старт-кодон «спрятаны» в шпильке или другой вторичной структуре РНК, это может затруднить доступ рибосомы, ослабляя сигнал.
f. Примеры сигнала:
g. Ссылки на литературу:
Hironao Wakabayashi, Chandani Warnasooriya, Dmitri N Ermolenko (2020). Extending the Spacing between the Shine-Dalgarno Sequence and P-Site Codon Reduces the Rate of mRNA Translocation. Journal of Molecular Biology, 2020 Jul 24;432(16):4612-4622. doi: 10.1016/j.jmb.2020.06.008. Epub 2020 Jun 13.
Bhattacharyya, S., Jacobs, W. M., Adkar, B. V., Yan, J., & Shakhnovich, E. I. (2018). Accessibility of the Shine-Dalgarno Sequence Dictates N-Terminal Codon Bias in E. coli, 70(5), Mol Cell. 2018 Jun 7;70(5):894-905.e5. doi: 10.1016/j.molcel.2018.05.008. Epub 2018 Jun 7.
Syue-Ting Kuo, Ruey-Lin Jahn, Yuan-Ju Cheng, Yi-Lan Chen, Yun-Ju Lee, Florian Hollfelder, Jin-Der Wen, and Hsin-Hung David Chou, (2020). Global fitness landscapes of the Shine-Dalgarno sequence. Genome Research, Published in Advance May 18, 2020, doi:10.1101/gr.260182.119
2.Сервис для поиска выбранного сигнала.
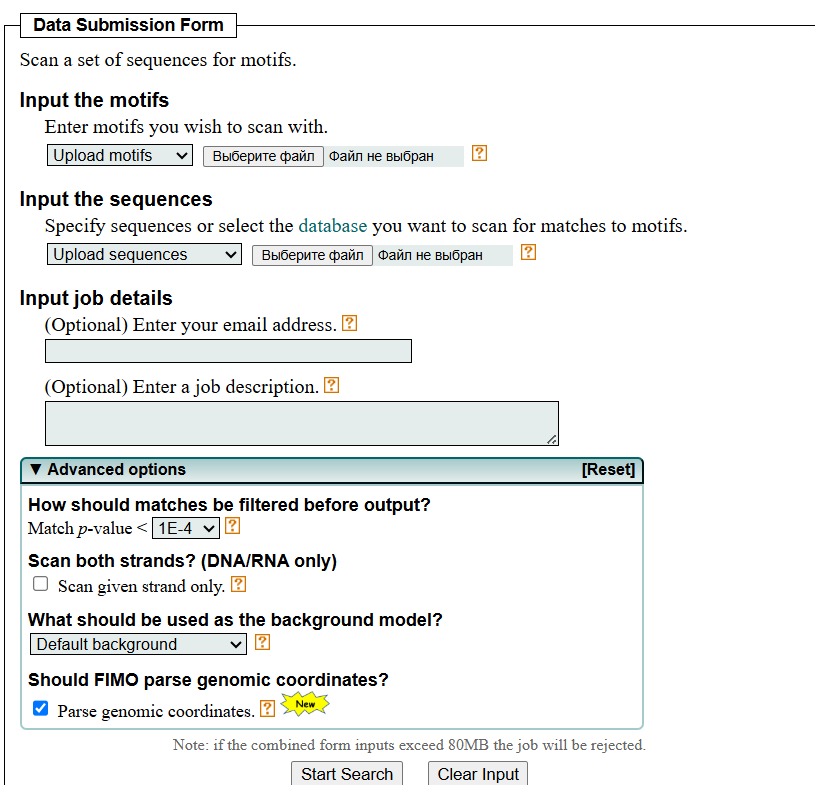
Бесплатных и доступных сервисов для поиска именно последовательности Шайна-Далгарно к сожалению я не нашла, но в данном случае можно воспользоваться сервисом FIMO
FIMO расшифровывается как «Find Individual Motif Occurrences» (найти отдельные вхождения мотива). Программа ищет в наборе последовательностей вхождения известных мотивов, обрабатывая каждый мотив независимо. Мотивы должны быть в формате MEME Motif Format. Веб-версия FIMO также позволяет вводить мотивы в дополнительных форматах.
Алгоритм FIMO
FIMO преобразует каждый введённый мотив в PSSM (позиционно-специфическую оценочную матрицу) с логарифмическими шансами и использует её для сканирования каждой входной последовательности. Он находит все позиции в последовательности, которые соответствуют мотиву и имеют статистически значимую оценку. Вы можете контролировать пороговое p-значение для значимости совпадений и выбрать, будет ли FIMO искать совпадения на обеих цепях, если последовательность комплементарна (например, для ДНК или РНК).