Занятие 7. A- и B-формы ДНК
- С помощью программы fiber пакета 3DNA построены A- и B-формы дуплекса ДНК, для чего
использовались следующие команды
fiber -a gatc-a.pdb и fiber -b gatc-b.pdb
Вид спиралей с торца.
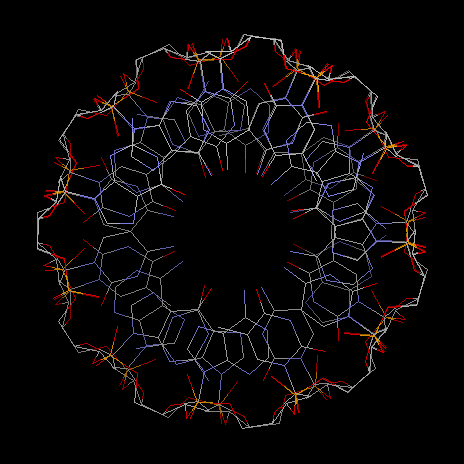


Спираль в A-форме (вид с торца) Спираль в B-форме (вид с торца) Спираль dna24 -
Таблица описания форм ДНК .
Данная на анализ ДНК файла dna24.pdb имеет больше нуклеотидов на один виток по сравнению с ДНК А- и В- формами, а соответственно она скручена сильнее. Также расстояния большой и малой бороздок данной ДНК сближены, т.е. теперь большая и малая бороздки менее отличимы друг от друга. По-видимому, из-за этого немного теряется специфичность к белкам транскрипции и трансляции, поэтому предполагается, что данная ДНК является неактивной(интронной) и экспрессируется очень редко.A-форма B-форма Файл dna24.pdb Тип спирали (правая или левая) правая правая правая Шаг спирали (Å) 28.03 33.75 в среднем 30.00 Число оснований на виток 10 11 12 Ширина большой бороздки 16.81(измерялась между атомами Т11А.Р-С24В.Р, Т11А.Р-С24А.Р и еще несколькими парами) 17.21(измерялась между атомами Т11А.Р-А26В.Р, Т27В.Р-А10А.Р и еще несколькими парами) 16.24(измерялась между атомами А10А.Р-А25В.Р, С13А.Р-G22B.Р и еще несколькими парами) Ширина малой бороздки 7.98(измерялась между атомами Т3А.Р-С24В.Р, Т23В.Р-С4А.Р и еще несколькими парами) 11.69(измерялась между атомами G25B.P-G5A.P, Т23В.Р-T7A.Р и еще несколькими парами) 13.38(измерялась между атомами G22B.Р-A5A.Р, U23В.Р-С4А.Р и еще несколькими парами)
-
Анализ структур ДНК, проведенный при помощи программ find_pair
и analyze.
Была введена команда find_pair -t xxxx.pdb stdout | analyze, где хххх.pdb - входной файл и получены следующие результаты:
Таблица выдачи программы find_pair и analyze.
Наибольший вклад в различия А- и В- конформаций ДНК вносят торсионные углы delta, zeta & chi. Такое различие обуславливает тот факт, что В-форма более растянута (имеет меньшую скрученность), чем А-форма.Замечено, что значения торсионных углов dna24 намного более близки к А-форме, чем к В-форме, поэтому dna24 более похожа на А-форму, что наглядно показано ниже:A-форма B-форма Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 174.8 41.7 79.0 -147.8 -75.1 -157.2 2 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 3 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 4 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 5 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 6 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2 7 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 8 C -51.7 174.8 41.7 79.0 -147.8 -75.0 -157.2 9 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 10 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 11 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 12 C -51.7 174.8 41.7 79.1 -147.7 -75.1 -157.2 13 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 14 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 15 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 16 C -51.7 174.8 41.7 79.1 --- --- -157.2 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -51.7 174.8 41.7 79.0 --- --- -157.2 2 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 3 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 4 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 5 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 6 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 7 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 8 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 9 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 10 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 11 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 12 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 13 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 14 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 15 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2 16 G --- 174.8 41.7 79.1 -147.7 -75.1 -157.2
Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 136.4 31.1 143.4 -140.8 -160.5 -98.0 2 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 3 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 4 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 5 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 6 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 7 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 8 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 9 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 10 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 11 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 12 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 13 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 14 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 15 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 16 C -29.9 136.4 31.1 143.4 --- --- -98.0 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -29.9 136.4 31.1 143.4 --- --- -98.0 2 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 3 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 4 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 5 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 6 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 7 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 8 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 9 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 10 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 11 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 12 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 13 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 14 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 15 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 16 G --- 136.4 31.1 143.4 -140.8 -160.5 -98.0
dna24 Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- --- 71.8 87.4 -131.8 -82.4 -175.5 2 C -61.0 159.6 54.1 80.2 -148.0 -74.3 -162.9 3 A -65.2 172.5 49.7 77.2 -152.8 -76.4 -163.6 4 G -56.5 175.6 50.9 80.6 -150.9 -74.8 -160.9 5 A -55.0 179.3 45.0 80.8 -160.4 -75.9 -154.5 6 G 143.0 -164.5 -173.0 81.0 -134.1 -70.4 0.9 7 U -64.1 175.8 50.8 81.8 -154.1 -71.3 -164.4 8 U -64.8 176.8 53.3 81.0 -153.2 -79.6 -158.6 9 A -57.3 170.2 46.7 80.4 -151.0 -80.5 -158.7 10 A -49.4 163.6 49.2 80.8 -148.3 -80.2 -160.1 11 A -57.7 164.4 54.1 81.8 -147.9 -79.2 -163.0 12 U -65.6 175.3 50.1 81.8 -143.2 -73.7 -154.4 13 C -66.0 171.4 53.3 81.2 -160.8 -62.7 -155.6 14 U -79.8 178.8 63.5 76.2 -167.6 -68.9 -169.5 15 G -76.4 -175.1 59.3 83.9 -148.5 -73.0 -163.5 16 C -56.1 170.3 53.8 79.6 --- --- -153.6 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -51.3 164.3 50.5 78.6 --- --- -154.0 2 G -68.7 179.5 54.9 83.0 -145.8 -76.3 -162.4 3 U 160.3 170.6 177.4 88.7 -143.4 -66.9 -172.5 4 C -50.1 167.0 43.8 78.8 -174.5 -62.3 -151.0 5 U -66.6 177.7 50.0 82.7 -141.9 -80.2 -157.3 6 A -62.6 161.6 59.5 82.5 -145.8 -82.2 -165.0 7 A -56.5 170.7 51.6 80.3 -146.5 -80.7 -160.8 8 A -58.8 171.3 46.3 80.3 -157.3 -72.5 -152.9 9 U -61.2 171.9 56.6 81.7 -152.3 -79.6 -162.2 10 U -68.0 175.2 50.6 81.4 -152.5 -74.7 -165.1 11 G 149.2 -172.4 -178.1 83.2 -138.0 -69.9 6.9 12 A -68.4 -170.7 50.3 84.7 -166.1 -69.5 -152.7 13 G -45.6 169.5 44.4 78.7 -159.1 -65.4 -161.5 14 A -62.6 177.3 45.1 78.9 -149.2 -80.4 -162.4 15 C -66.8 168.5 53.6 80.5 -151.5 -71.5 -159.6 16 G --- --- 67.2 85.2 -141.4 -74.2 -173.2
Общий вид спиралей.
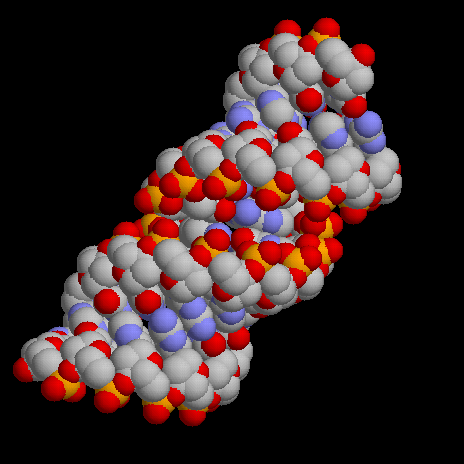

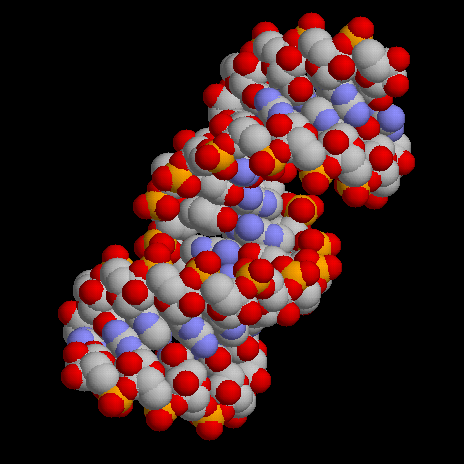
Спираль в A-форме Спираль в B-форме Спираль dna24 Результаты работы с программой pdb2img.
Вид структуры СВЕРХУ СБОКУ A-форма 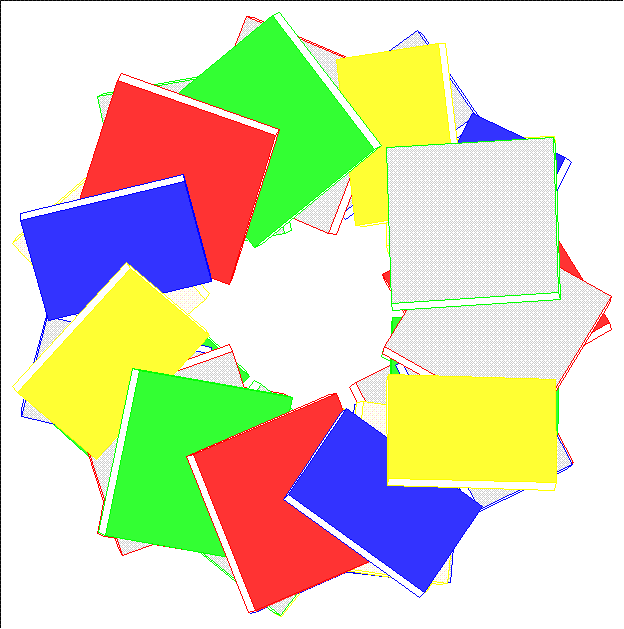

В-форма 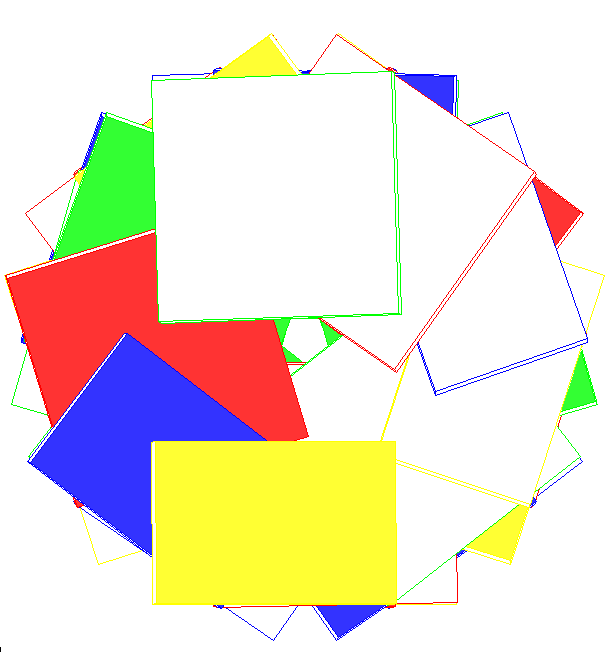
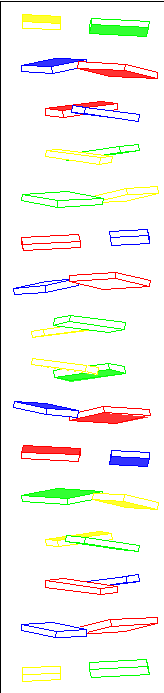
dna24