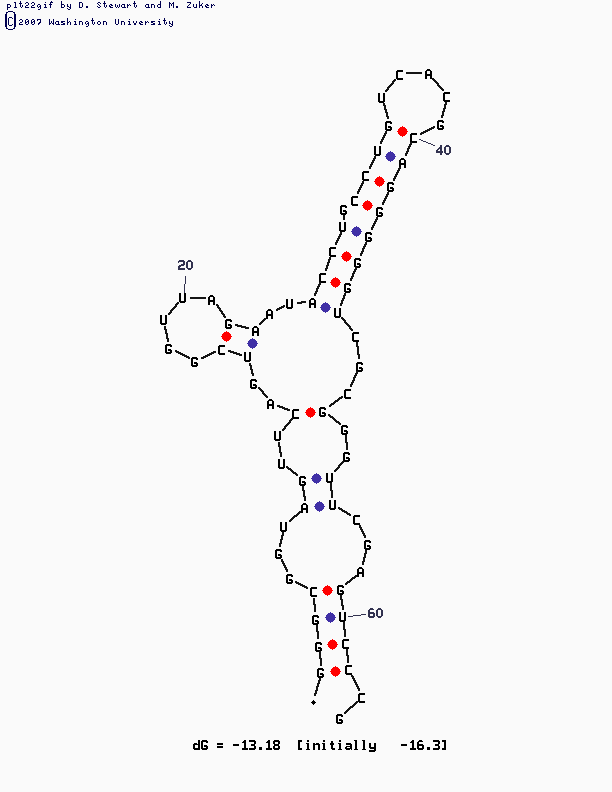
Длина - 14 нуклеотидов.
нуклеотиды. Хоть 607 и 649 входят в одну спираль, между ними нет ковалентной фосфодиэфирной связи, а они имеют лишь стекинг-взаимодествие с
нуклеотидами антипараллельной цепи, т.е. на
этом месте спираль прерывается. Еще 2 разрыва между 618, 658 и 661 нуклеотидами.
Спирали тРНК, её разрывы, а также 5'- и 3'- концевые атомы фосфора можно рассммотреть на рисунке ниже. Остовы нуклеотидов, входящих в
спирали, но не соединенных остовом со своей спиралью, представлены в проволочной модели толщиной 200.
рис 1. Вид спиралей в остовной модели
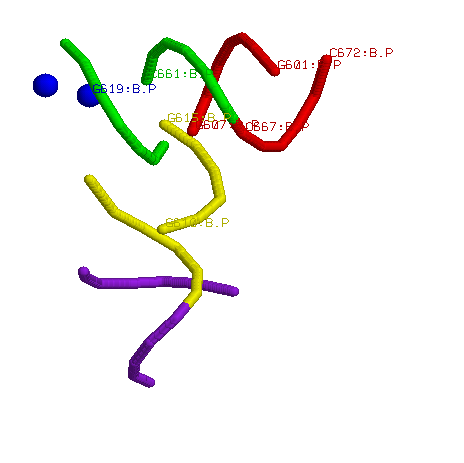
Для создания картинки была использована следующая последовательность команд в RasMol:
background white
select all
wireframe off
wireframe
select 649-655 or 661-672
backbone 200
color red
select 601-607 or 666-672
color red
backbone 200
select 658 and backbone
wireframe 200
color red
select 618 and backbone
wireframe 200
color red
select 638-644 or 622-632
backbone 200
color green
select 610-615
color green
backbone 200
select 608 and backbone
wireframe 200
color green
select 648 and backbone
wireframe 200
color green
select 656 and backbone
wireframe 200
color blue
select 619 and backbone
wireframe 200
color blue
select 655 and *.p
label
select 155 and *.p
label
select 610 and *.p
label
select 622 and *.p
label
select 644 and *.p
label
select 638 and *.p
label
select 632 and *.p
label
select 619 and *.p
label
select 656 and *.p
label
select 658 and *.p
label
select 618 and *.p
label
select 615 and *.p
label
select 648 and *.p
label
select 649 and *.p
label
select 601 and *.p
label
select 672 and *.p
label
select 607 and *.p
label
select 661 and *.p
label
restrict rna
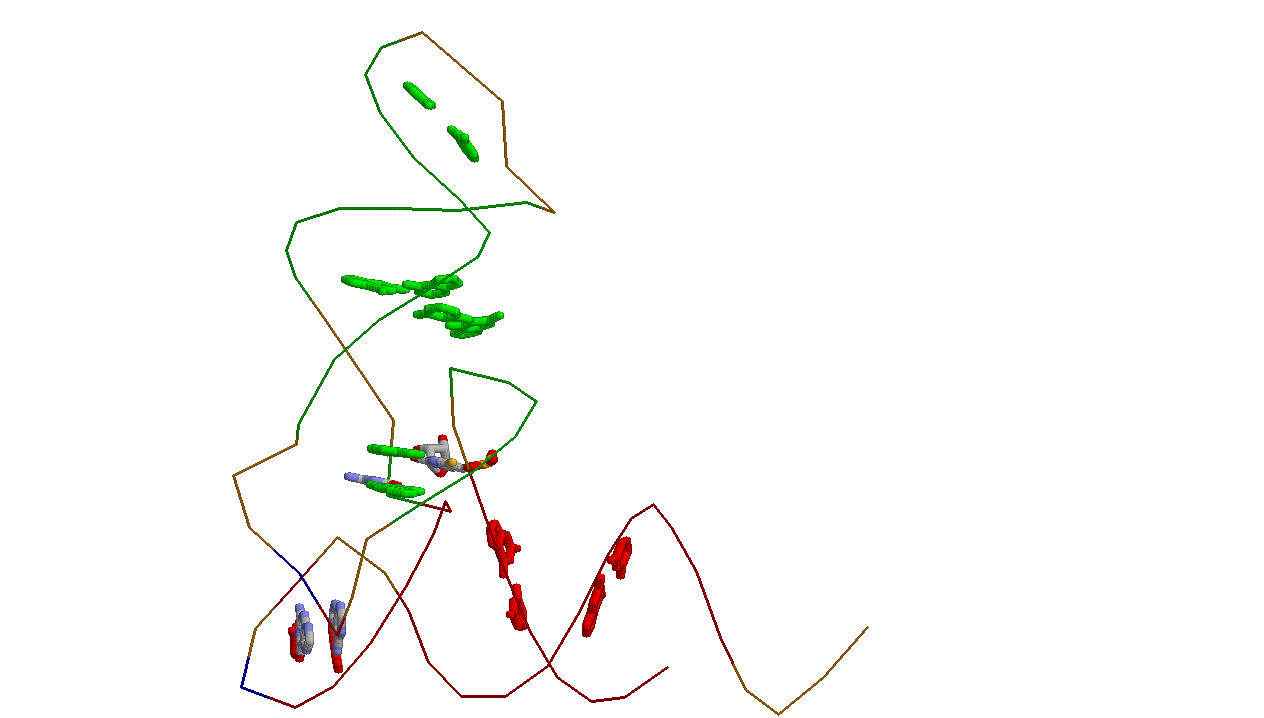
В данной структуре присутствуют внеспиральные стекинг-взаимодействия. Например, это взаимодействия между парами:
644-645-609
621-648
618-657-619
615-659-660
635-636
673-674-675
Внеспиральные азотистые основания, образующие стекинг-взаимодейиствия, представлены в проволочной модели толщины 40 и
окрашены в цвета химических элементов. 644 и 615 нуклеотиды принадлежат спиралям, но образуют стекинг-взаимодействия с внеспиральными
нуклеотидами, они на рисунке показаны в цвете своих спиралей
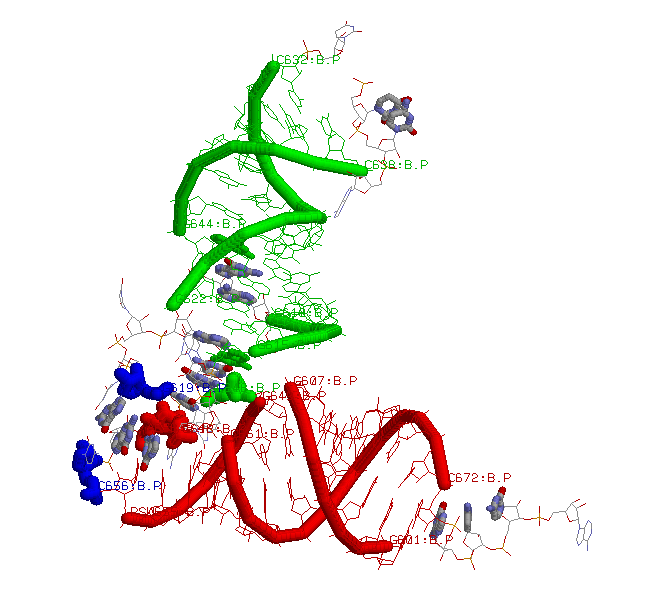
Чтобы лучше было видно взаимное расположение внеспиральных азотистых оснований, образующих стекинг-взаимодействия, приведем
следующую картинку:
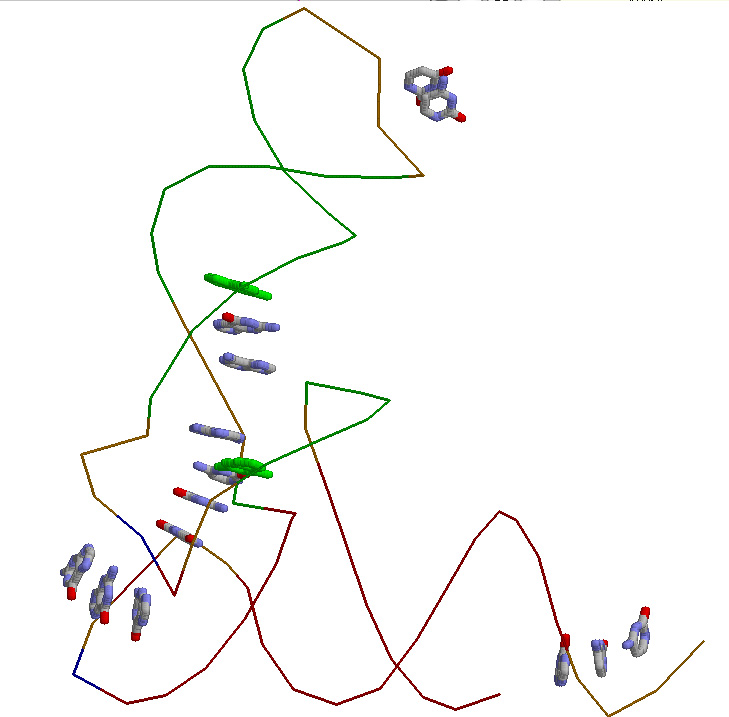
Также в структуре найдены водородные связи между азотистыми основаниями, не относящиеся к Уотсон-Криковским. Поиск проводился визуально
Найденные взаимодействия представлены в таблице:
| # | Азотистые основания |
| 1 | G 604-U 669 |
| 2 | G 649-PSU 665 |
| 3 | 5MU 654-A 658 |
| 4 | PSU 655-G 618 |
| 5 | C 638-C 632 |
| 6 | G 644-A 626 |
| 7 | G 610-U 625 |
| 8 | A 614-4SU 608 |
| 9 | G 615-C 648 |
и отражены на рисунке:
Расстояние между донорами и акцепторами водорода антипараллельных цепей в спиралях исследуемой РНК было в среднем 3 ангстрема.
Это расстояние было принято за длину водородной связи. Нестандартные Уотсон-Криковские взаимодействия были найдены только в двух парах
, которые на рисунке имеют желтый цвет (зачеркнутое основание не имеет водородных связей).
Cпирали РНК были исследованы визуально (напоминает А-форму ), а также измерили ширину большой и малой бороздки и шаг спирали.
Таблица результатов измерения .
| Тип спирали (правая или левая) |
правая |
| Шаг спирали (Å) |
28.87 |
| Ширина большой бороздки |
15.84(измерялась между атомами G602B.P-С673В.Р) |
| Ширина малой бороздки |
8.82(измерялась между атомами G602B.Р-PSU665B.Р) |
На основании этих наблюдений можно сделать вывод, что спираль РНК наиболее близка к А-форме ДНК.
Cначала программа einverted была запущена со значением 50 параметра Minimum score threshold. В результате программа выдала пустые файлы.
Затем параметр постепенно уменьшали, и на значении 15 программа выдала 1 спираль, на значении 5 и ниже - 2 спирали, которые были очень короткими
по сравнению с настоящими спиралями из файла PDB. Эти 2 спирали можно видеть на рисунке желтым цветом:
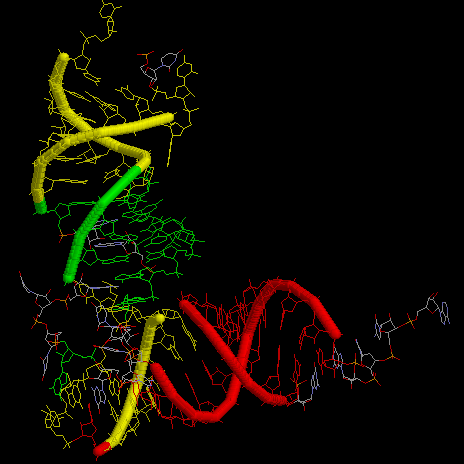
Выдача программы очень не похожа на действительные данные. Это может быть связано с её алгоритмом. По-видимому, он не учитывает неканонические
взаимодействия между нуклеотидами, а также модифицированные нуклеотиды, которые могли бы с высокой вероятностью оказаться в PDB-файле тРНК.
Программа запускалась следующей командой:
mfold SEQ=trna2.fasta P=...
параметр Р=... был в пределах от 10 до 70, но только во втором случае, при Р=15, программа выдала самый лучший, на мой взгляд, вариант
вторичной структуры. Остальные варианты были очень непохожи на "клеверный лист", либо их свободные концы были в очень неподходящих местах, там
где они в принципе не могут быть.