Создание репрезентативной выборки гомологов белка QUEC_BACSU
Эту задачу можно выполнить с помощью программы BLAST. Для этого были созданы запросы с параметрами, описанными в Таблице 1. Как видно из таблицы, поиск гомологов среди прокариот и среди эукариот производился отдельно.
| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Максимальное количество хитов |
| По прокариотам | protein blast(blastp) | RefSeq | Исключение Firmicutes и Eukaryota | 1E-15 | 2024 |
| По эукариотам | protein blast(blastp) | RefSeq | Только Eukaryota | 1 | 10 |
Таблица 1. Содержит описание параметров запроса и некоторые сведения о результатах.
Далее при помощи GenPept были составлены две выборки гомологов: среди прокариот и среди эукариот. Данные о составленных выборках представлены в Таблице 2. Очень любопытно, что 4 гомолога были найдены среди вирусов.| Домен | Филум/Царство | Название организма | Количество белков |
| Archaea | Euryarchaeota | Thermoplasmatales archaeon SCGC AB-540-F20 | 1 |
| Haloferax sulfurifontis | 1 | ||
| Thaumarchaeota | Candidatus Nitrosoarchaeum koreensis | 1 | |
| Candidatus Nitrososphaera gargensis Ga9.2 | 1 | ||
| Bacteria | Cyanobacteria | Cyanothece sp. CCY0110 | 1 |
| Xenococcus sp. PCC 7305 | 1 | ||
| Bacteroidetes | Bacteroides sp. 1_1_30 | 1 | |
| Bacteroides plebeius | 1 | ||
| Chlorobium | Chlorobium ferrooxidans | 1 | |
| Spirochaetes | Leptospira kirschneri | 1 | |
| Plactomycetes | Rhodopirellula sp. SWK7 | 1 | |
| Actinobacteria | Saccharothrix espanaensis DSM 44229 | 1 | |
| Aquificae | Hydrogenobaculum sp. HO | 1 | |
| Chlamydiae/Verrucomicrobia group | Verrucomicrobiae bacterium DG1235 | 1 | |
| Fibrobacteres/Acidobacteria group | Terriglobus roseus DSM 18391 | 1 | |
| Deinococcus-Thermus | Deinococcus deserti VCD115 | 1 | |
| g-proteobacteria | Wohlfahrtiimonas chitiniclastica | 1 | |
| Vibrio sinaloensis | 1 | ||
| Nitrospirae | Candidatus Nitrospira defluvii | 1 | |
| Viruses | - | Cellulophaga phage phiSM | 1 |
| Eukaryotes | Fungi | Cryptococcus gattii | 1 |
| Animalia | Amphimedon queenslandica | 2 | |
| Plantae | Ricinus communis | 1 | |
| Plantae | Ostreococcus tauri | 1 |
Таблица 2. Представленность различных филумов и царств в двух выборках.
Множественное выравнивание гомологов белка QUEC_BACSU
Из выбранных гомологов при помощи MUSCLE было составлено множественное выравнивание. Рисунок 1 иллюстрирует результат, представленный в JalView.
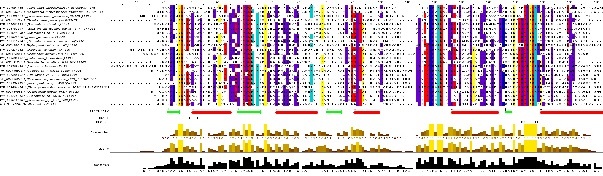
Результаты анализа множественного выравнивания гомологов белка QUEC_BACSU
В целом, консервативность выравнивания довольно низкая. Консервативность выравнивания должна быть выше в районе участков, участвующих в образовании вторичной структуры белка, т.к. замена аминокислоты в альфа-спирали или бетта-листе может привести к разрушению вторичной структуры. В данном выравнивании такую тенденцию можно выделить с трудом. Возможно, это связано с тем, что в структуре белка мало аморфных участков, а также с качеством выравнивания в целом. Большие проблемы с выравниванием есть в начале и в конце выравнивания. Вероятно, так происходит потому, что выравнены домены QueC, а переферические участки последовательностей, входящих в выравнивание, не гомологичны и были приобретены независимо.
На элемнеты вторичной структуры приходтся меньше колонок-гэпов, чем на аморфные участки, в то время как единственный выделенный мною блок представлен на аморфном участке. Это противоречие можно объяснить малым количеством блоков в принципе, то есть по нахождению одного блока нельза выделить какого-то закона их распределения. Местонахождение всего одного блока ничего нам не говорит. В качестве блоков я искал 6 и больше идущих подряд консервативных аминокислот.
Консервативность участка, связывающего лиганд (РО43-), слабая. Возможно, это связано с простотой лиганда в целом. Рис. 2.1, 2.2 и 2.3 изображают
трехмерную структуру участка, связывающего лиганд, с соотвтственно 70%, 50% и 25% порогом cut-off для цвета.
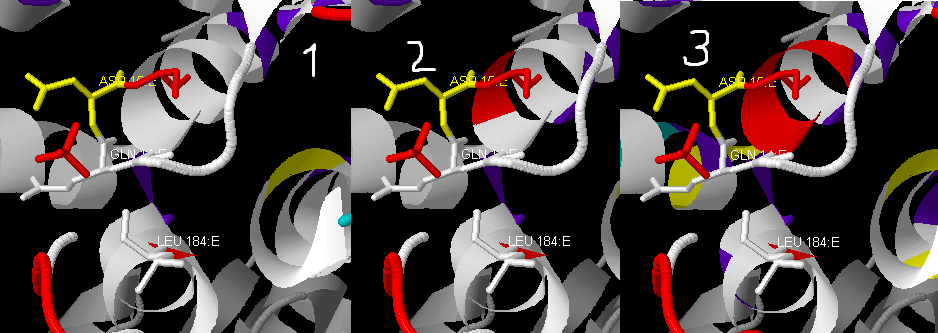
Аминокислотные замены в лиганд-связывающем участке не связаны сходством аминокислот по радикалу. Сложно сказать, есть ли среди гомологов неактивные в плане связывания лиганда по этому выравниванию. Отчасти потому, что лиганд имеет слишком простую структуру и может найтись много конфигураций, которые будут его связывать.