Программа getorf пакета EMBOSS
Программа getorf находит ORF в заданной последовательности. Командойgetorf -minsize 30 -table 0 -find 1 d89965.entretбыла запущена программа с параметрами, требуемыми чтобы найденные ORF обладали желаемыми свойствами (длина > 29 а.о. и начинались старт-кодоном, а заканчивались стоп-кодоном).
CDS заданной последовательности: 163-435. Из найденных рамок считывания такие координаты соответствуют пятой. Swissprot AC: P0A7B8. Swissprot относит этот белок к E. coli. Видимо, архивный nr просто содержит устаревшие сведения, а кусочек генома E. coli подвергся секвенированию вместе с геномом крысы по случайности (из кишечника той же крысы, возможно).
Файлы-списки
Для получения списка последовательностей одного заданного белка нескольких вполне конкретных организмов были использованы следующие команды:
seqret sw:adh*_* infoseq -only -usa adh.fasta | grep -f list > list2 seqret @list2 list3.fasta
EnsEMBL
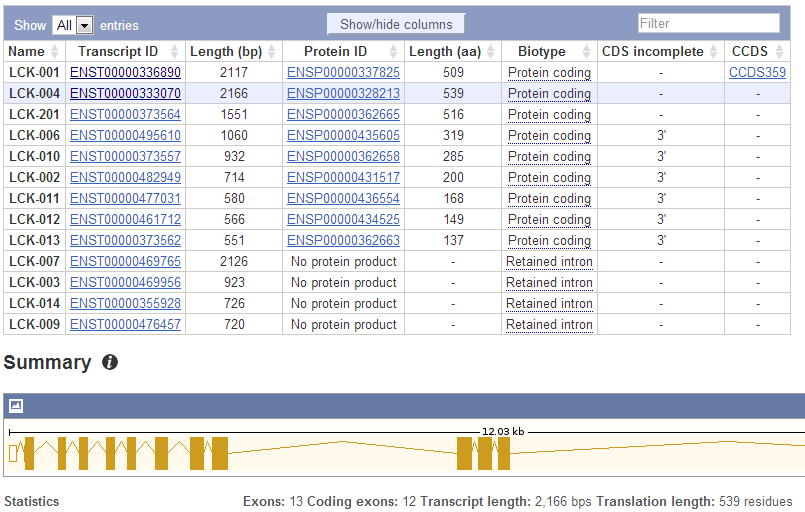
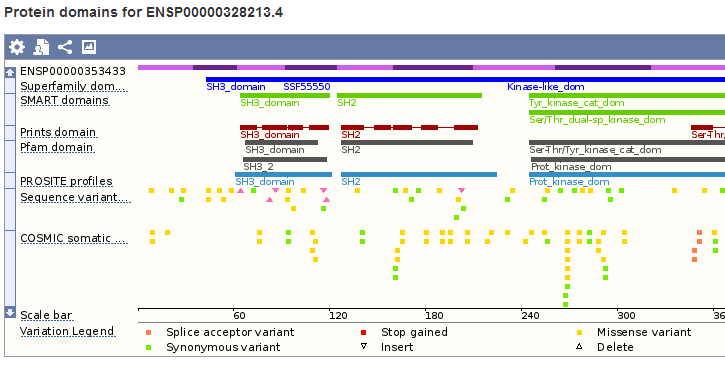
Для гена LCK: