A- и В- формы ДНК. Структура РНК
С помощью программы fiber пакета 3DNNA было построено три структуры: А- и В-формы ДНК gatc x 5 и gc x 10 Z-формы.
fiber -a gatc-a.pdb fiber -b gatc-b.pdb fiber -z gatc-z.pdb
В струтктуре цветом были выделены конкретные элементы спирали (см. рис. 1-4):
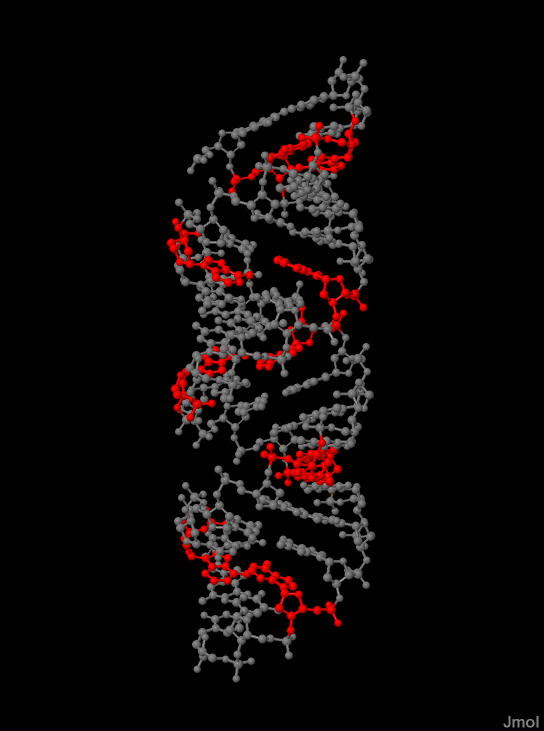
Далее с сайта PDB были получены две структуры: 1ksx (ДНК) и РНК (1qrt). Разрывов не было обнаружено ни в Днк, ни в РНК(рис. 5 и 6):
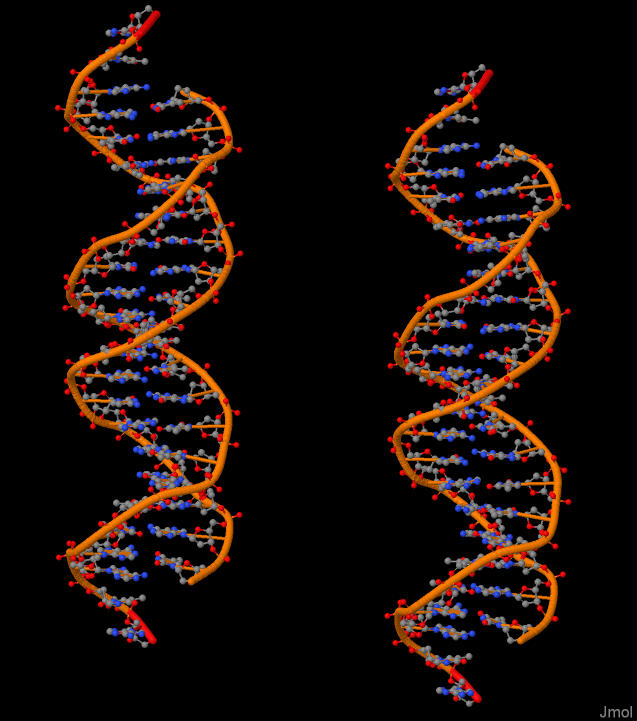
Большая и малая боороздки ДНК
Большая бороздка глубже, чем малая. При этом, как мы еще убедимся, это не значит, что большая борздка шире. На рис. 7 представлен пример бороздок на В-ДНК.
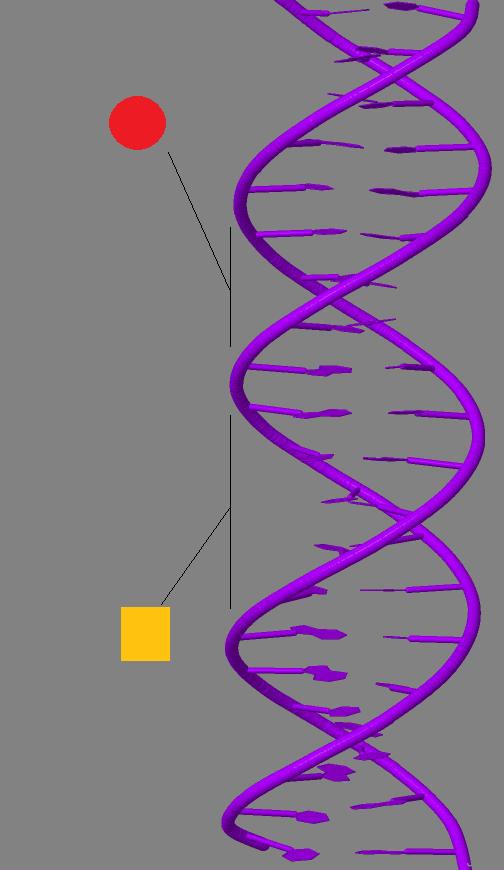

- В сторону большой бороздки обращены атомы с 16.n1 по n3.
- В сторону малой бороздки обращены атомы с 28.n3 по c5.
- Остальные основания: n1 и с6, вегда обращены в сторону сахаро-фосфатного остова.
Средствами JMol я также сравнил три фрмы ДНК, а резуьтаты сравнения представлены в таблице 1.
| A-форма | B-форма | *Z-форма | |
| Тип спирали (правая или левая) | Правая | Правая | Левая |
| Шаг спирали (A) | 28 | 33,75 | 43,5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 17 | 17 | 18 |
| Ширина малой бороздки | 8 | 12 | 10 |
Таблица 1. Результаты сравнения A, B и Z форм ДНК.
Торсионные углы
С помощью опции JMol measure были найдены торсионные углы А и В форм (таблица 2).
| альфа | бетта | гамма | дельта | эпсилон | зета | хи | |
| А-форма | 64 | 174 | 41 | 79 | -147 | -75 | -157 |
| B-форма | -29 | 136 | 31 | 143 | 105 | -160 | -98 |
Таблица 2. Вычисленные торсионные углы
find_pair, analyze
После превращения командой remediator файла с расширением .pdb в старую форму, была запущена команда
find_pair -t XXXX.pdb stdout | analyzeс помощью которой были определены торсионные углы A-, B-форм. Они приведены в табл. 3 - для сравнения с измеренными "вручную".
| альфа | бетта | гамма | дельта | эпсилон | зета | хи | |
| А-форма | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-форма | -29.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98.0 |
Табл. 3. Тормионные углы, измеренные программами пакета 3DNA.
Больше всего торсионные углы отличаются у Z-формы - она и чисто визуально самая неоднородная из всех.
Были определены средние значения торсионных углов в спирали ДНК 1ksx (табл. 4)
| alpha | beta | gamma | delta | epsilon | zeta | chi |
| -18,37 | 31,45 | 18,48 | 141,01 | -136,83 | -88,42 | -116,48 |
Вот эта же таблица для РНК
| alpha | beta | gamma | delta | epsilon | zeta | chi |
| -46,8 | 20,5 | 55,7 | 85,0 | -142,4 | -80,0 | -116,2 |
Таблица 4. Средние значения торсионных углов.
Наиболее отклоняющиеся от средних значения торсионных углов у 15 G (гуанин) и у 7 C (цитозин).
Определение структуры водородных связей.
Выводы делались на основании выдачи программы find-pair:
Strand I Strand II Helix
1 (0.007) B:...2_:[..G]G-----C[..C]:..71_:B (0.005) |акцепторный стебель
2 (0.006) B:...3_:[..G]G-----C[..C]:..70_:B (0.008) |акцепторный стебель
3 (0.007) B:...4_:[..G]G-----C[..C]:..69_:B (0.004) |акцепторный стебель
4 (0.010) B:...5_:[..G]G-----C[..C]:..68_:B (0.010) |акцепторный стебель
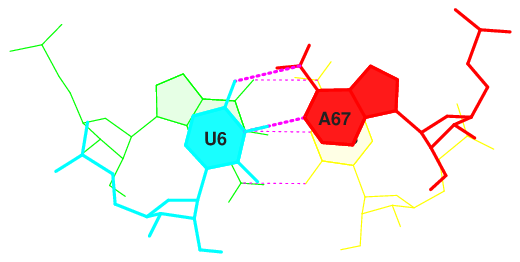
5 (0.008) B:...6_:[..U]U-----A[..A]:..67_:B (0.010) |акцепторный стебель
6 (0.013) B:...7_:[..A]Ax----U[..U]:..66_:B (0.005) |
7 (0.008) B:..49_:[..C]C-----G[..G]:..65_:B (0.007) |D-стебель
8 (0.009) B:..50_:[..G]G-----C[..C]:..64_:B (0.007) |D-стебель
9 (0.006) B:..51_:[..A]A-----U[..U]:..63_:B (0.009) |D-стебель
10 (0.004) B:..52_:[..G]G-----C[..C]:..62_:B (0.004) |D-стебель
11 (0.011) B:..53_:[..G]G----xC[..C]:..61_:B (0.003) |
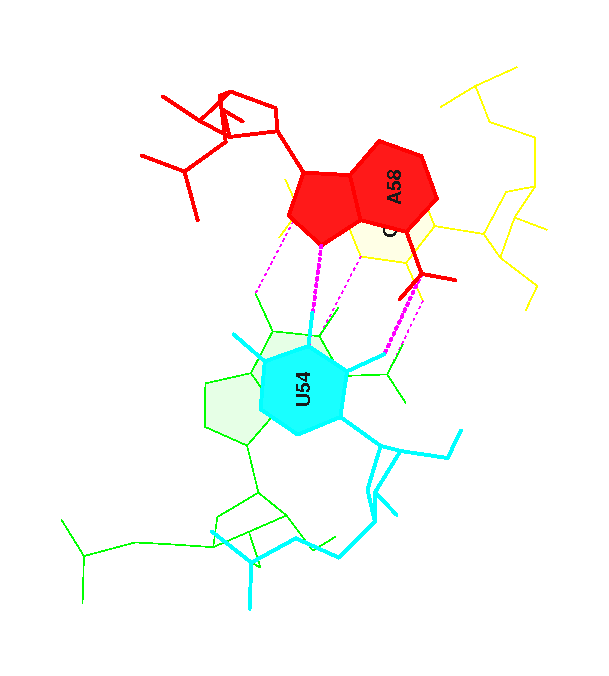
12 (0.007) B:..54_:[..U]U-*--xA[..A]:..58_:B (0.003) |
13 (0.010) B:..55_:[..U]Ux**+xG[..G]:..18_:B (0.010) x
14 (0.009) B:..37_:[..A]A-*---U[..U]:..33_:B (0.004) |
15 (0.007) B:..38_:[..U]U-*---U[..U]:..32_:B (0.005) |
16 (0.005) B:..39_:[..U]U-----A[..A]:..31_:B (0.003) |антикодоновый стебель
17 (0.004) B:..40_:[..C]C-----G[..G]:..30_:B (0.009) |антикодоновый стебель
18 (0.003) B:..41_:[..C]C-----G[..G]:..29_:B (0.007) |антикодоновый стебель
19 (0.005) B:..42_:[..G]G-----C[..C]:..28_:B (0.004) |антикодоновый стебель
20 (0.008) B:..43_:[..G]G-----C[..C]:..27_:B (0.007) |антикодоновый стебель
21 (0.005) B:..44_:[..C]Cx*---A[..A]:..26_:B (0.005) |
22 (0.010) B:..10_:[..G]G-----C[..C]:..25_:B (0.008) |Т-стебель
23 (0.005) B:..11_:[..C]C-----G[..G]:..24_:B (0.007) |Т-стебель
24 (0.009) B:..12_:[..C]C----xG[..G]:..23_:B (0.007) |Т-стебель
25 (0.004) B:..13_:[..A]A-**+xA[..A]:..45_:B (0.006) |
26 (0.004) B:..14_:[..A]A-*--xU[..U]:...8_:B (0.011) |
27 (0.006) B:..15_:[..G]Gx**+xC[..C]:..48_:B (0.011) x
28 (0.014) B:..19_:[..G]Gx---xC[..C]:..56_:B (0.005) +
29 (0.003) B:..46_:[..U]U-**+-U[..U]:..47_:B (0.007) +
Акцепторный стебель: 2-6=67-71;
D-стебель: 10-12=23-25;
T-стебель: 49-52=62-65;
Антикодоновый стебель: 39-43=27-31
Неканонические пары: 55-18, 44-26, 55-18, 38-32, 45-13.
Detailed H-bond information: atom-name pair and length [ON] 1 G-----C [3] O6 - N4 2.68 N1 - N3 2.87 N2 - O2 2.91 2 G-----C [3] O6 - N4 2.83 N1 - N3 2.74 N2 - O2 2.54 3 G-----C [3] O6 - N4 2.94 N1 - N3 2.88 N2 - O2 2.78 4 G-----C [3] O6 - N4 2.81 N1 - N3 2.86 N2 - O2 2.89 5 U-----A [2] N3 - N1 2.67 O4 - N6 2.95 6 A-----U [2] N6 - O4 2.60 N1 - N3 2.76 7 C-----G [3] O2 - N2 2.68 N3 - N1 2.68 N4 - O6 2.61 8 G-----C [3] O6 - N4 2.99 N1 - N3 2.79 N2 - O2 2.54 9 A-----U [2] N6 - O4 2.49 N1 - N3 2.55 10 G-----C [3] O6 - N4 3.08 N1 - N3 2.86 N2 - O2 2.60 11 G-----C [3] O6 - N4 2.82 N1 - N3 2.66 N2 - O2 2.41 12 U-*---A [2] O2 - N6 2.63 N3 - N7 3.01 -неканоническая пара 13 U-**+-G [2] O4'* O6 3.94 O2 - N2 2.65 -неканоническая пара 14 A-*---U [1] N6 - O2 3.54 -неканоническая пара 15 U-*---U [1] O4 - N3 3.14 -неканоническая пара 16 U-----A [2] N3 - N1 3.36 O4 - N6 3.54 17 C-----G [3] O2 - N2 2.43 N3 - N1 3.05 N4 - O6 3.57 18 C-----G [3] O2 - N2 2.50 N3 - N1 2.84 N4 - O6 3.13 19 G-----C [3] O6 - N4 2.98 N1 - N3 2.78 N2 - O2 2.48 20 G-----C [3] O6 - N4 2.87 N1 - N3 2.75 N2 - O2 2.59 21 C-*---A [2] O2 * N1 3.48 N3 - N6 3.29 -неканоническая пара 22 G-----C [3] O6 - N4 2.77 N1 - N3 2.71 N2 - O2 2.55 23 C-----G [3] O2 - N2 2.67 N3 - N1 2.81 N4 - O6 2.81 24 C-----G [3] O2 - N2 2.78 N3 - N1 2.75 N4 - O6 2.63 25 A-**+-A [2] N6 - N1 3.16 N1 - N6 3.01 -неканоническая пара 26 A-*---U [3] O2P* O4 3.70 N7 - N3 2.74 N6 - O2 2.46-неканоническая пара 27 G-**+-C [2] N1 - O2 2.80 N2 - N3 2.67 -неканоническая пара 28 G-----C [3] O6 - N4 2.61 N1 - N3 2.55 N2 - O2 2.46 29 U-**+-U [3] O3'* N1 3.46 O2'- N3 2.84 O2 * O4 3.40-неканоническая пара
D-стебель:
T-стебель: -2-6 - 71-67
Акцепторный стебель:
Антикодоновый стебель:
Были определены номера остатков, между которыми возможен стэкинг, на основании следющих данных из .out:
step i1-i2 i1-j2 j1-i2 j1-j2 sum 1 GG/CC 4.24( 2.95) 0.00( 0.00) 0.19( 0.00) 0.39( 0.00) 4.82( 2.95) 2 GG/CC 3.73( 2.42) 0.00( 0.00) 0.44( 0.00) 0.15( 0.00) 4.32( 2.42) 3 GG/CC 3.92( 2.54) 0.00( 0.00) 0.41( 0.00) 0.00( 0.00) 4.32( 2.54) 4 GU/AC 6.54( 3.63) 0.00( 0.00) 0.00( 0.00) 4.14( 2.56) 10.67( 6.19) - хорошее перекрывание 5 UA/UA 0.99( 0.09) 0.00( 0.00) 1.29( 1.04) 0.16( 0.00) 2.43( 1.13) 6 AC/GU 1.82( 0.78) 0.00( 0.00) 0.00( 0.00) 6.19( 3.15) 8.01( 3.93) 7 CG/CG 0.00( 0.00) 0.00( 0.00) 6.16( 3.35) 0.00( 0.00) 6.16( 3.35) 8 GA/UC 4.25( 2.45) 0.00( 0.00) 0.00( 0.00) 3.19( 0.93) 7.44( 3.38) 9 AG/CU 3.91( 3.53) 0.00( 0.00) 0.14( 0.00) 0.01( 0.00) 4.07( 3.53) 10 GG/CC 2.66( 1.16) 0.00( 0.00) 1.92( 0.11) 0.00( 0.00) 4.58( 1.27) 11 GU/AC 6.35( 3.70) 0.00( 0.00) 0.00( 0.00) 5.74( 2.68) 12.09( 6.38) - хорошее перекрывание 12 UU/GA 3.81( 1.83) 0.00( 0.00) 0.00( 0.00) 4.48( 2.48) 8.29( 4.31) 13 UA/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 14 AU/UU 4.76( 1.35) 0.00( 0.00) 0.00( 0.00) 5.85( 3.17) 10.62( 4.52) - хорошее перекрывание 15 UU/AU 2.07( 0.74) 0.00( 0.00) 0.00( 0.00) 5.12( 3.41) 7.19( 4.15) 16 UC/GA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 2.71( 1.19) 2.71( 1.19) 17 CC/GG 0.01( 0.00) 0.00( 0.00) 1.39( 0.00) 3.13( 1.62) 4.53( 1.62) 18 CG/CG 0.59( 0.00) 0.00( 0.00) 4.11( 1.37) 0.40( 0.00) 5.10( 1.37) 19 GG/CC 1.95( 0.57) 0.00( 0.00) 0.93( 0.00) 0.16( 0.00) 3.04( 0.57) 20 GC/AC 6.70( 3.73) 0.00( 0.00) 0.00( 0.00) 5.56( 3.63) 12.26( 7.36) - хорошее перекрывание 21 CG/CA 0.00( 0.00) 0.00( 0.00) 0.15( 0.00) 1.40( 0.80) 1.55( 0.80) 22 GC/GC 4.71( 1.81) 0.00( 0.00) 0.00( 0.00) 6.10( 3.04) 10.81( 4.85) - хорошее перекрывание 23 CC/GG 0.00( 0.00) 0.00( 0.00) 0.76( 0.00) 2.89( 1.35) 3.64( 1.35) 24 CA/AG 2.40( 0.85) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 2.40( 0.85) 25 AA/UA 0.00( 0.00) 1.71( 0.00) 0.00( 0.00) 0.00( 0.00) 1.71( 0.00) 26 AG/CU 4.24( 1.52) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 4.24( 1.52) 27 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 28 GU/UC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)Вот взаимоедействие 19-56 вне цепи:
Вот картинки (рис.8) с изображением некоторых из выбранных пар: