16S rRNA
Шесть последовательностей 16S рРНК (FINM2, LACAC, GEOKA, CLOTE, BACAN и STAES) удалось извлечь из полногеномных записей embl. Седьмая последовательность была найдена при помощи nblast в базе refseq_rna последовательности найденной ранее рРНК LACAC, как самой близкой филогенетически.
| Мнемоника вида | AC записи в EMBL | Начало последовательности | Конец последовательности | Цепь |
| LACAC | CP000033.3 | 59255 | 60826 | Прямая |
| CLOTE | AE015927 | 41801 | 43309 | Обратная |
| FINM2 | AP008971.1 | 611796 | 613319 | Прямая |
| STAES | AE015929 | 1722288 | 1723841 | Обратная |
| GEOKA | BA000043.1 | 10421 | 11973 | Прямая |
| BACAN | AE017225 | 9336 | 10845 | Обратная |
| STRPN | AE007317.1 | 15161 | 16674 | Обратная |
Таким образом, было получено выравнивание пяти последовательностей, по которому было построено дерево, представленное на картинке ниже.
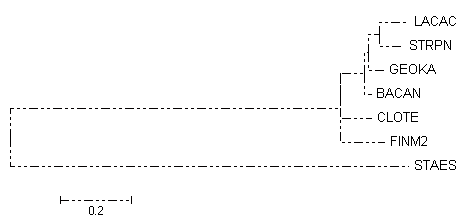
Дерево во многом не соответствует правильному:
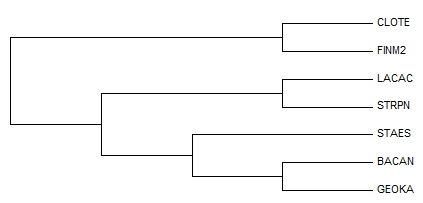
В правильном дереве нет ветвей {LACAC,STRPN,GEOKA,BACAN}против{STAES,CLOTE,FINM2} и {STAES,FINM2,BACAN,CLOTE}против{LACAC,STRPN,GEOKA}. В полученном нет {STAES,GEOKA,BACAN}против{LACAC,STRPN,CLOTE,FINM2}, {BACAN,GEOKA}против{LACAC,STRPN,CLOTE,FINM2,STAES}.
Построение и анализ дерева, содержащего паралоги
На основании proteo.fasta было построена БД для поиска посредством blastp гомологов белка CLPX_BACSU. Гомологи белка среди выбранных семи бактерий были найдены, по их выравниванию было построено дерево:

Красным здесь помечен случай эволюции гена в результате видообразования, синим - в результате дупликации гена.