Домен QueC
Для исследования был взят домен с ID QueC, AC PF06508 (pfam.xfam.org/family/PF06508. Домен катализирует первую стадию синтеза квеуозина, на которой синтезируется preQ0.
Все последовательности с доменом сохранены в .jvp и fasta.
Были выбраны две архитектуры с данным доменом:
| Архитектура | Число доменов | Кратко о втором домене | Число последовательностей |
| QueC, QueF | 2 | PF14489: участие в синтезе квеуозина | 166 |
| GATase_7, QueC | 2 | PF13537: глутамин амидотрансфераза | 22 |
Список всех архитектур, содержащих QueC, по ссылке: pfam.xfam.org/family/PF06508#tabview=tab1.
С помощью скрипта swisspfam-to-xls.py была получена сводная таблица с информацией о последовательностях, содержащих домен.
Затем с Uniprot были скачаны все последовательности по имеющимся AC и представлены в виде таблицы при помощи скрипта uniprot-to-taxonomy.py. Т.о., были получены данные о таксономической принадлежности последовательностей. Далее при помощи функции VLOOKUP эти данные были перенесены в основную таблицу, в которую также был добавлен столбец, в котором подсчитаны длины доменов в последовательностях.
По всей видимости, это любовь до гроба!
Запуск скрипта filter-alignment.py вкупе с ВПР дал понять, что одна из архитектур стречается только у одного организма, а вторая - у нескольких архей. Квеуозин - совсем не мейнстримное соединение.
Возьмем другое семейство доменов
Было взято сеймейство GspH (PF12019). Были выбраны две архитектуры:
| Архитектура | Число доменов | AC второго домена | Число последовательностей |
| N_methyl, GspH | 2 | PF07963 | 504 |
| N_methyl_2, GspH | 2 | PF13544 | 485 |
Далее были произведены все те же действия, что и с предыдущим семейством, в результате получился fasta project и таблица.
Для первой структуры был рассмотрен таксон Bacteria, для второй - Proteobacteria. Для первой архитектуры были рассмотрены подтаксоны Deinococcus и Proteobacteria, для второй - Betaproteobacteria и Gammaproteobacteria. Результат в табл. ниже:
| Архитектура | Число последовательностей | Betaproteobacteria | Gammaproteobacteria |
| N_methyl, GspH | 20 | 13 | 7 |
| N_methyl_2, GspH | 20 | 10 | 10 |
После этого при помощи filter_alignment.py были получены интересующие нас последовательности, обладающие искомой архитектурой. После некоторой обработки в jalview выравнивание стало выглядеть вот так.
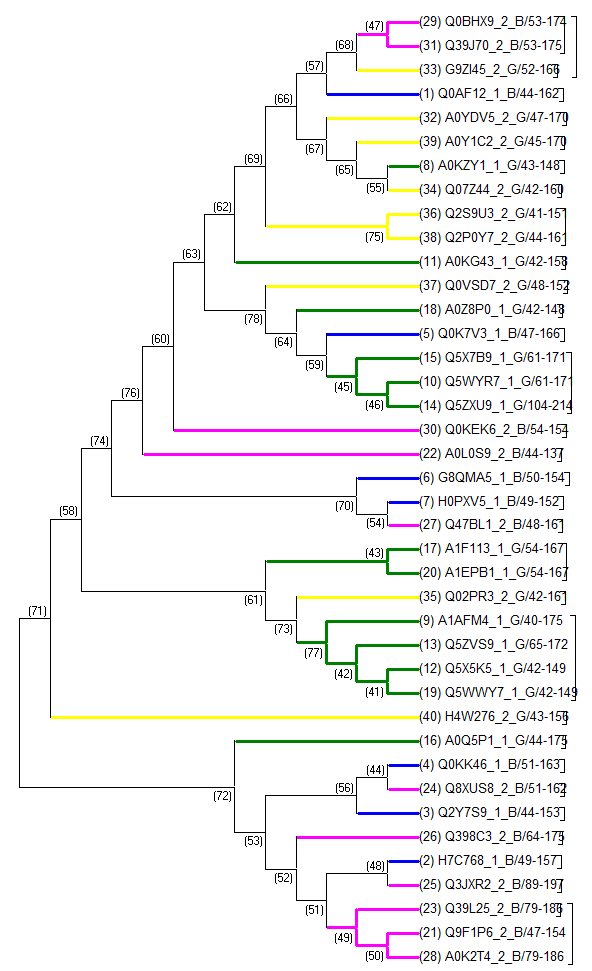
Составление дерева
Архитектура N_methyl, GspH была закодирована как А1, другая - как А2. B - Betaproteobacteria, G - Gammaproteobacteria.
C помощью МЕGA было построено дерево. Ветви раскрашены следующим образом:
- 1_B - синий
- 1_G - зелный
- 2_B - лиловый
- 2_G - желтый
Вывод
Общность основных кластеров в получившемся дереве основана в основном не на обладании общей архитектурой, а на общей таксономической принадлежности. Кластеры, содержащие последовательности одной архитектуры, принадлжащие к разным подтаксонам, имеют не более 3-5 листьев, гораздо меньше, чем таксономически схожие.
Можно сделать вывод об эволюционной нейстойчивости доменных архитектур GspH, но надо учитывать, что в силу необходимости был выбран таксон довольно узкий для наших задач (Proteobacteria). Надо полагать, общность последовательностей с одной архитектурой была бы ярче, если бы была возможность работать с таксоном более высокого уровня.