| Название | Мнемоника | Таксономия |
| Bacillus subtilis | BACSU | Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis group |
| Clostridium botulinum | CLOB1 | Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Clostridium tetani | CLOTE | Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Enterococcus faecalis | ENTFA | Bacteria; Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus |
| Finegoldia magna | FINM2 | Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiales incertae sedis; Clostridiales Family XI. Incertae Sedis; Finegoldia |
| Geobacillus kaustophilus | GEOKA | Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus |
| Lactobacillus acidophilus | LACAC | Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
| Lactobacillus delbrueckii | LACDA | Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
(((CLOTE,CLOB1),FINM2),(((LACDA,LACAC),ENTFA),(BACSU,GEOKA)));
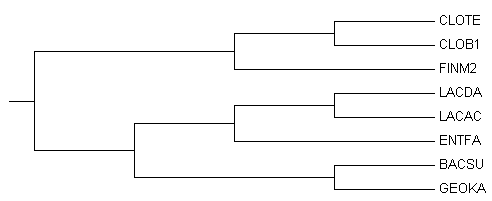
Дерево содержит пять нетривиальных ветвей:
1) {CLOTE,CLOB1,FINM2} против {LACDA,LACAC,ENTFA,BACSU,GEOKA}
2) {LACDA,LACAC,ENTFA} против {CLOTE,CLOB1,FINM2,BACSU,GEOKA}
3) {CLOTE,CLOB1,FINM2,LACDA,LACAC,ENTFA} против {BACSU,GEOKA}
4) {CLOTE,CLOB1} против {FINM2,LACDA,LACAC,ENTFA,BACSU,GEOKA}
5) {LACDA,LACAC} против {CLOTE,CLOB1,FINM2,ENTFA,BACSU,GEOKA}
{CLOTE,CLOB1,FINM2} - Clostridia,
{CLOTE,CLOB1} - Clostridiaceae,
{LACDA,LACAC,ENTFA,BACSU,GEOKA} - Bacilli,
{LACDA,LACAC,ENTFA} - Lactobacillales,
{LACDA,LACAC} - Lactobacillaceae,
{BACSU,GEOKA} - Bacillales.
Фактор элонгации трансляции Ts (мнемоника - EFTS).
Из Swiss-Prot были получены последовательности белков с данной функцией из отобранных бактерий.
С помощью программы muscle были получены выравнивания данных последовательностей. Дополнительное задание можно посмотреть здесь.
fprotpars -sequence EFTS_aligned.fasta -outfile efts.fprotpars
Скобочная формула (efts_aligned.treefile):
((((((CLOTE,CLOB1),FINM2),(GEOKA,BACSU)),ENTFA),LACDA),LACAC);
Это единственное (неукорененное) дерево, выданное программой. Топология этого дерева совпалает с правильной.

fprotdist -sequence EFTS_aligned.fasta -outfile efts.fprotdist

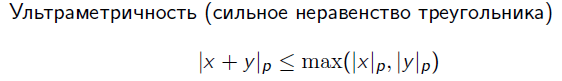
то есть все теугольники равнобедренны (a=b всегда, a>=c, b>=c).
(LACAC,BACSU)=0.830089
(BACSU,CLOB1)=0.826901
(LACAC,CLOB1)=1.002405
0.830089 и 0.826901 меньше третьего, ультраметричность не выполняется.
Аддитивность: из трёх сумм расстояний между четырьмя точками A,B,C,D две равны между собой и больше третьей (AB+CD=AC+BD>AD+BC).
(LACAC,LACDA)+(ENTFA,GEOKA)=0.386283+0.511004=0.897287
(LACAC,ENTFA)+(LACDA,GEOKA)=0.714161+0.738932=1.453093
(LACDA,ENTFA)+(LACAC,GEOKA)=0.711666+0.801072=1.513386
Аддитивность не выполняется, если не округлять значения.
1.Алгоритм Neighbor-Joining (по умолчанию, выдаёт неукоренённое дерево с длинами ветвей; не предполагает молекулярных часов)
fneighbor -datafile efts.fprotdist -outfile efts.neighbor-joining -outtreefile eftsNJ.tree

Скобочная формула:
(LACDA:0.18941,(ENTFA:0.25376,((BACSU:0.18515,GEOKA:0.15788):0.08865, (FINM2:0.20853,(CLOB1:0.20884,CLOTE:0.16024):0.21712):0.14315):0.03959):0.26602,LACAC:0.19687);
2.Алгоритм UPGMA (выдаёт укоренённое дерево с длинами ветвей. Его можно применять, если справедлива гипотеза молекулярных часов, т.е. матрица расстояний не слишком далека от ультраметрической)
fneighbor -datafile efts.fprotdist -outfile efts_upgma.fneighbor -outtreefile efts_upgma.tree -treetype u
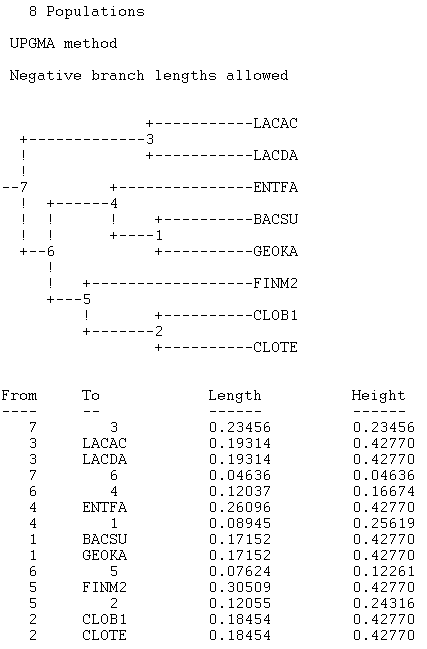
Скобочная формула:
((LACAC:0.19314,LACDA:0.19314):0.23456,((ENTFA:0.26096,(BACSU:0.17152, GEOKA:0.17152):0.08945):0.12037,(FINM2:0.30509,(CLOB1:0.18454, CLOTE:0.18454):0.12055):0.07624):0.04636);
Результаты можно посмотреть здесь.