Укоренение в среднюю точку
Дерево, построенное методом neighbor-joining,(LACDA:0.18941,(ENTFA:0.25376,((BACSU:0.18515,GEOKA:0.15788):0.08865, (FINM2:0.20853,(CLOB1:0.20884,CLOTE:0.16024):0.21712):0.14315):0.03959):0.26602,LACAC:0.19687);
было скопировано в файл intree (без расширения). Далее была запущена программа retree.exe. После соглашения со всеми настройками (по умолчанию), программа построила дерево:
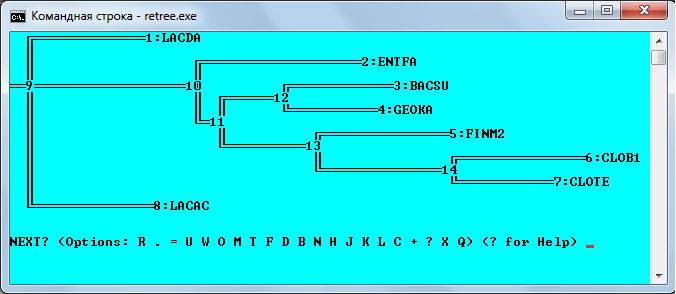
Т.к. необходимое дерево должно быть укорененно в среднюю точку ("Midpoint root the tree") был выбран параметр M. Вот, что получилось:
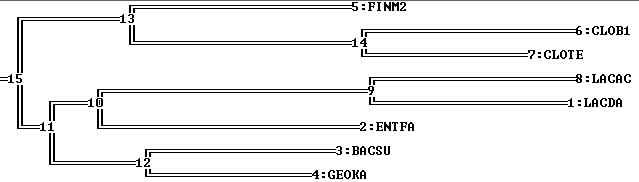
Укоренение произошло в следующую ветвь:
(FINM2,(CLOB1,CLOTE)) vs (((LACAC,LACDA),ENTFA),(BACSU,GEOKA))
Данное дерево соответствует правильному. С деревом, построенным методом максимальной экономии, сделать аналогичную опреацию не получится, т.к. метод fprotpars не основывается на молекулярных часах и не выдает длины ветвей. Также не имеет смысла применять данную программу к дереву, построенному методом UPGMA, т.к. он выдает уже укоренённое дерево, кстати, в моём случае укоренённые деревья получились разными.
Использование внешней группы
-к файлу с невыровненными последовательностями белков сначала была добавлена последовательность белка того же семейства из кишечной палочки (в качестве внешней группы):seqret sw:efts_ecoli stdout >> EFTS_2.fasta
-затем было построено выравнивание всех последовательностей и результат дан на вход программе fprotpars:
muscle -in EFTS_2.fasta -out EFTS_2_aligned.fasta
fprotpars -sequence EFTS_2_aligned.fasta -outfile efts_2.fprotpars
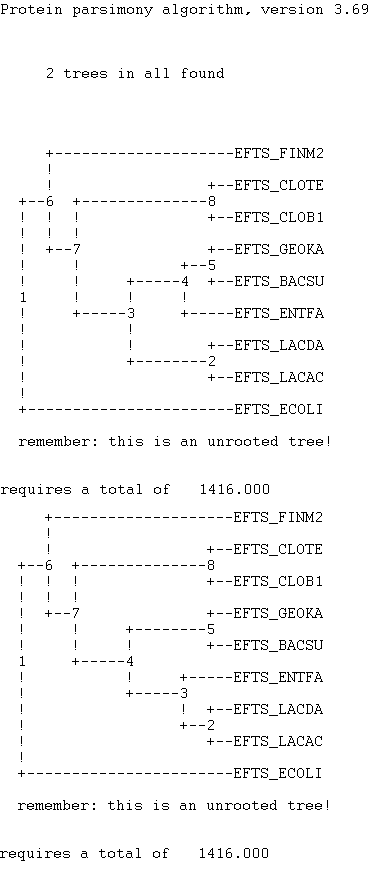
Получилось 2 неукоренённых дерева:
После обработки программой retree, указав в качестве действия "select an Outgroup", а в качестве номера - тот, что программа retree присвоила листу ECOLI (9), получилось следующее:
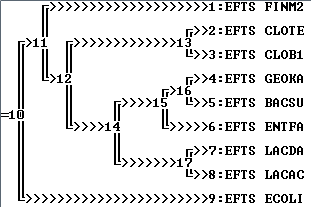
Если убрать аутгруппу, то полученное укорененное дерево не соответствует укоренению правильного дерева, положение листа EFTS ENTFA другой (в предыдущих деревьях он находится в ветви ((LACAC,LACDA),ENTFA), а не в ((GEOKA,BACSU),ENTFA).
Бутстрэп
-Создано 100 бутстрэп-реплик выравнивания белков протеобактерий программой fseqboot: результат.-По полученным репликам созданы деревья программой fprotpars:
fprotpars -sequence efts_aligned.fseqboot -outfile efts_2_fsq.fprotpars
Cкобочные формулы и изображения полученных деревьев.
-Из полученных деревьев создано единое дерево по принципу "расширенного большинства" (extended majority rule tree):
fconsense -in efts_aligned.treefile -outfile efts_aligned.fconsense
Результат
Ветви, не получившие большинства:
(GEOKA,BACSU,ENTFA) vs (CLOTE,CLOB1,FINM2,LACDA,LACAC) получила поддержку 32.50
Такая же ветвь получилась в №2 (использование аутгруппы).
(GEOKA,ENTFA,LACDA,LACAC) vs (CLOTE,CLOB1,FINM2,BACSU) получила поддержку 1.50
(BACSU,ENTFA) vs (CLOTE,CLOB1,FINM2,GEOKA,LACDA,LACAC) получила поддержку 0.50
Здесь все положение "портит" BACSU.
Дерево, построенное бустрэп-анализом соответствует дереву, построенному с использованием аутгруппы. Также, оно идентично дереву, посроенным программой fprotpars без бутсрэп-анализа.