2onk, 1zcd, 1l0l, 3k9v, 1afo;
2k4t, 1tly, 1ek9, 1a0s, 3fid.
Представленные α-спиральные трансмембранные белки связываются с мембраной с помощью алифатических α-спиралей, полностью гидрофобных α-спиралей, гиброфобных петель.
Трансмембранные β-баррели закреплены засчет полностью гидрофобных или алифатических β-тяжей, алифатических петель. Общая структура у этого типа белков более однотипная, чем у предыдущего.
б)Сравнение топологии трансмембранных частей двух белков, с α-спиральными трансмембранными участками и с трансмембранными β-баррелями:
| PDB код | Число цепей | Тип (спираль, баррель) |
Число трансмембранных участков в цепи | Число остатков в одном трансмембранном
участке (типичное, минимальное, максимальное) |
(*) Толщина мембраны в ангстремах (расстояние между атомами на границах трансмембранных участков |
| 2onk | 10 | спираль | 12 TM вторичных структур, 2 TM субъединицы (С, D) | 21, 14, 31 | 32.0 ± 1.7 Å |
| 1ek9 | 3 | баррель | 12 TM вторичных структур, 3 ТМ субъединицы | 11, 9, 13 | 24.6 ± 1.4 Å |
Из записи в Uniprot следует, что данный белок принадлежит к суперсемейству АВС, у него есть 2 АВС ТМ домена типа 1. Каждей ТМ домен состоит из 6 α-спиралей из 21 а.о.
С помощью программы предсказания трансмембранных спиралей по последовательности (сервис TMHMM) получены следующие результаты: data
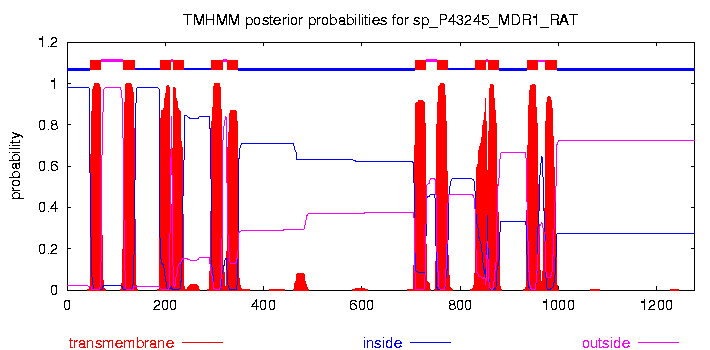
В TMHMM предсказано то же количество спиралей (12), что и в аннотации. Но, есть расхождения по координатам:
| Uniprot, координаты | Uniprot, длина | ТМНММ, координаты | ТМНММ, длина |
| 50-70 | 21 | 47-69 | 23 |
| 119-139 | 21 | 115-137 | 23 |
| 189-209 | 21 | 189-211 | 23 |
| 216-236 | 21 | 216-238 | 23 |
| 296-316 | 21 | 294-316 | 23 |
| 327-347 | 21 | 326-348 | 23 |
| 710-730 | 21 | 708-730 | 23 |
| 755-775 | 21 | 753-775 | 23 |
| 832-852 | 21 | 831-853 | 23 |
| 854-874 | 21 | 857-879 | 23 |
| 937-957 | 21 | 937-959 | 23 |
| 968-988 | 21 | 974-996 | 23 |
Полученные данные в формате msf. В файле разными цветами размечены трансмембранные участки (светло-зеленый MDR1_RAT, темно-зеленый MDR3_MOUSE), внеклеточные (голубой MDR1_RAT, синий MDR3_MOUSE) и внутриклеточные фрагменты (красный MDR3_MOUSE). Разметка согласно БД Uniprot.
| Число а.к. остатков | |
| Всего а.к. остатков в последовательности | 1277 |
| Остатки, предсказанные TMHMM как локализованные в мембране (всего) | 276 |
| Правильно предсказали - совпадают с 3D предсказанием (true positives, TP) | 235 |
| Предсказали не то, что нужно (а.о. предсказаны как мембранные, а по данным 3D таковыми не являются, false positives, FP) | 23 (+2 а.о. напротив гэпа [не учитывались]) |
| Правильно не предсказали ( не предсказаны, и по данным 3D не находятся в мембране, true negatives, TN) | 974 |
| Не предсказали то, что нужно (остатки по данным 3D находятся в мембране, false negatives, FN) | 27 |
| Чувствительность (sensivity) = TP / (TP+FN) | 0.8969 |
| Специфичность (specificity) = TN / (TN+FP) | 0.9763 |
| Точность(precision) = TP /(TP+FP) | 0.9106 |
| Сверхпредсказание = FP/ (FP+TP) | 0.0892 |
| Недопредсказание = FN / (TN+FN) | 0.0269 |
© Anastasia Maslova, 2011