Подготовка файлов координат и топологии
Начнем с того, что подготовим файл координат и файл топологии. В прошлом занятии нам был предоставлен gro файл с 38 молекулами этана. Создадим индекс файл котором будет группа из одной молекулы этана.make_ndx -f box_38.gro -o 1.ndx
(После запуска команды появляется приглашение к вводу. Для ознакомления с помощью надо нажать кнопку "h" + enter. Далее был выбран остаток номер 1
ri 1 -> enter -> появилась новая группа (3 r_1 : 8 atoms) -> q
Полученный файл: 1.ndx
Теперь создадим gro файл с одной молекулой и зададим ячейку. editconf -f box_38.gro -o et1.gro -n 1.ndx #зададим ячейку и расположим молекулу по центру ячейку
При запуске ediconf надо выбрать номер соответствующей группе из одной молекулы:
Select a group for output: Group 0 ( System) has 304 elements Group 1 ( Other) has 304 elements Group 2 ( ETH) has 304 elements Group 3 ( r_1) has 8 elements Select a group: 3 Selected 3: 'r_1'editconf -f et1.gro -o et.gro -d 2 -c
Файлы: et.gro, et1.gro
Исправим файл топологии et.top (->et1.top) из прошлого задания. В разделе [ molecules ] изменено количество молекул этана.
Разные параметры контроля температуры.
Дано 5 файлов с разными параметрами контроля температуры:be.mdp - метод Берендсена для контроля температуры.
vr.mdp - метод "Velocity rescale" для контроля температуры.
nh.mdp - метод Нуза-Хувера для контроля температуры.
an.mdp - метод Андерсена для контроля температуры.
sd.mdp - метод стохастической молекулярной динамики.
Скрипт по работе с 5-ю системами одновременно. Для посчета энергии на запрос программы надо выбрать 10 (потенциальная энергия)->enter->11 (кинетическая энергия)->enter->0 (закончить выбор параметров). Полученные файлы:
et_be.pdb, et_vr.pdb, et_nh.pdb, et_an.pdb, et_sd.pdb.
Данные о потециальной и кинетической энергии: et_be_en.xvg, et_vr_en.xvg, et_nh_en.xvg, et_an_en.xvg, et_sd_en.xvg.
Наблюдения, сделанные на основе визуального анализа:
В методе Берендсена в начале видны небольшие колебания по длине связи и вращение молекулы, но затем скорость вращения начинает достаточно быстро увеличиваться, амплитуда колебаний затихать. Поскольку термостат должен обеспечить постоянную энергию, то такая система, на мой взгляд, недостоверна.
В методе Андерсена видны небольшие колебания по длинам связей и валентным углам, однако молекула при этом не вращается. Вполне возможно, такое состояние характерно для достаточно низких температур при образовании молекулой кристалла.
В методе Нуза-Хувера наблюдаются небольшие колебания и вращение по связи С-С. Молекула находится в заторможенной конформации, но также видна и заслонённая.
В методе стохастической молекулярной динамики молекула движется очень быстро.
Метод "Velocity rescale" и метод Нуза-Хувера очень похожи друг на друга, но разница в том, что в Velocity rescale вращение по связи меньше, а амплитуда колебаний больше.
Графики изменения энергий
С помощью Gnuplot были построены графики изменения энергий (рекомендуемый вид - dot-plot).set datafile commentschars "#@&"
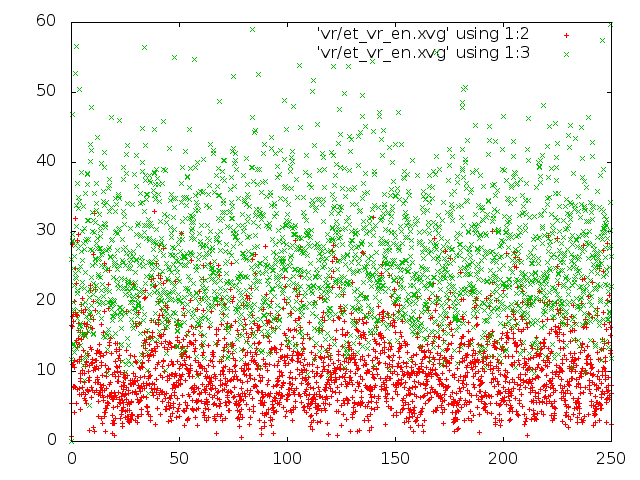
plot "./et_be_en.xvg" using 1:2, "./et_be_en.xvg" using 1:3
....
plot "./et_sd_en.xvg" using 1:2, "./et_sd_en.xvg" using 1:3
Зеленым цветом выделена кинетическая энергия, и зеленым потенциальная.
1. В методе Берендсена потенциальная энергия быстро уменьшается, кинетическая выходит на плато.
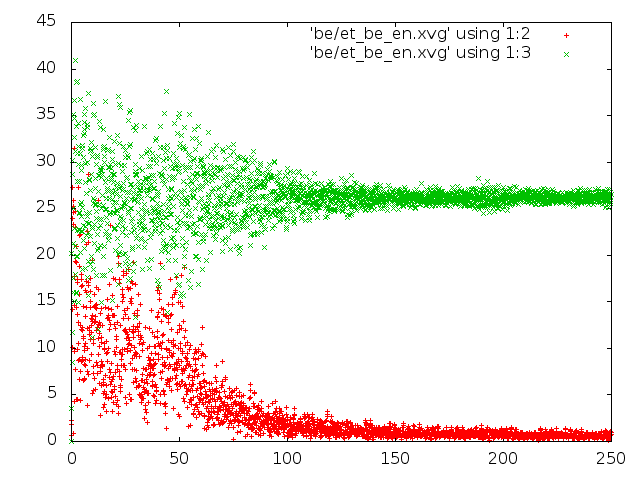
2. В методе Андерсена значения потенциальной и кинетической энергий примерно одинаковые и достаточно маленькие, поэтому данный термостат негодится для передачи молекуле необходимой энергии для того, чтобы она попала в глобальный минимум.
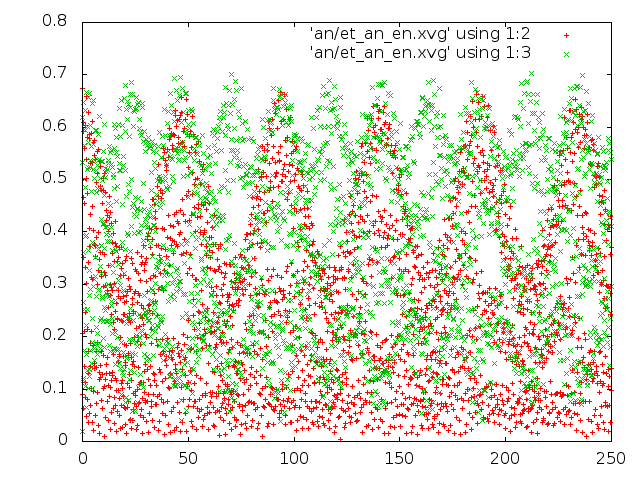
3. В методе Нуза-Хувера наблюдаются единичные достаточно высокие значения кинетической энергии.
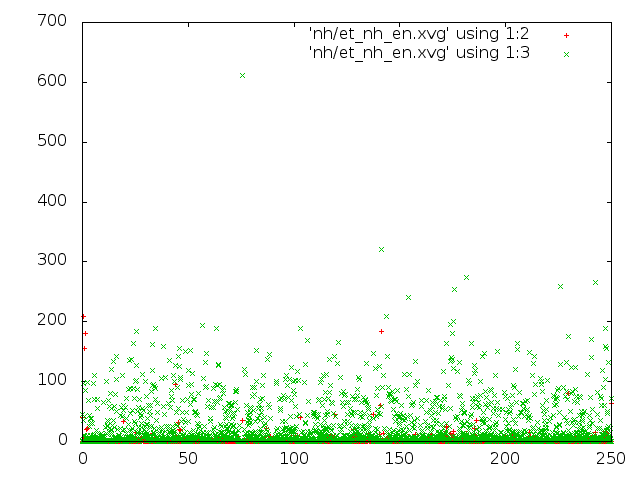
3. В методе стохастической молекулярной динамики значения кинетической энергии примерно постоянны.
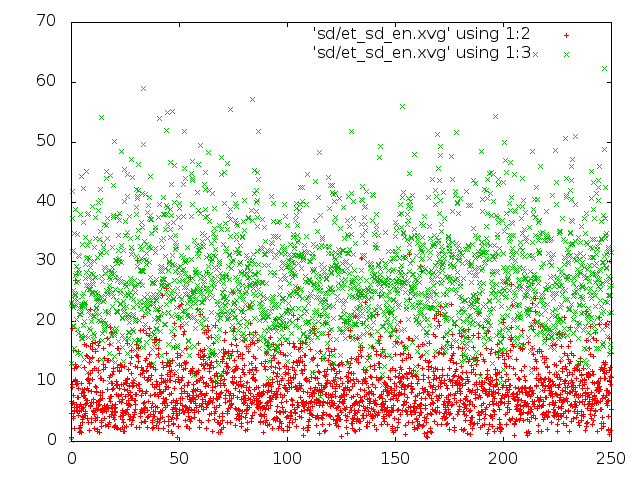
4. Метод "Velocity rescale" спохож на метод стохастической молекулярной динамики.
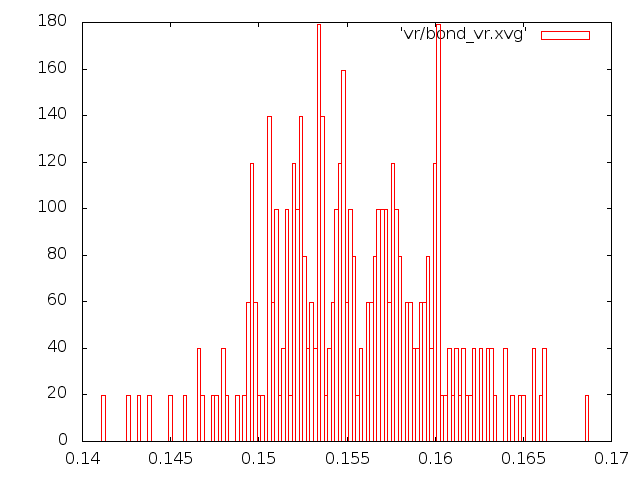
Графики распределения длин связей
Рассмотрим распределение длинны связи С-С за время моделирования. Сначала создадим индекс файл с одной связью. В текстовом редакторе создадим файл b.ndx со следующим содержимым:[ b ]
1 2
И запустим утилиту по анализу связей g_bond (закомментируем предыдущие команды в скрипте):
g_bond -f et_${i}.trr -s et_${i}.tpr -o bond_${i}.xvg -n b.ndx
Графики распределения длин связей были также построены в Gnuplot.
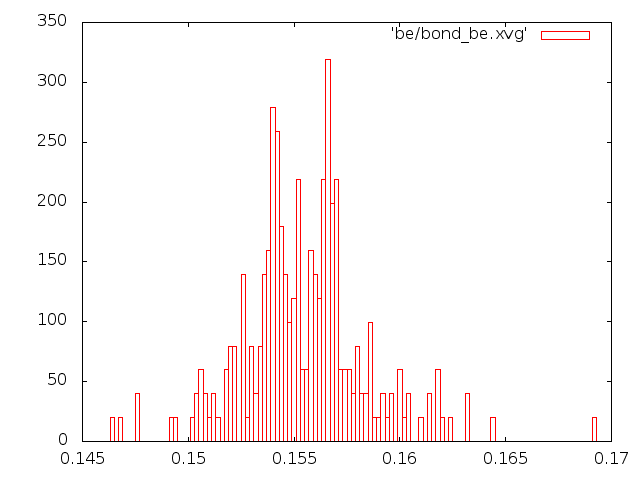
метод Берендсена
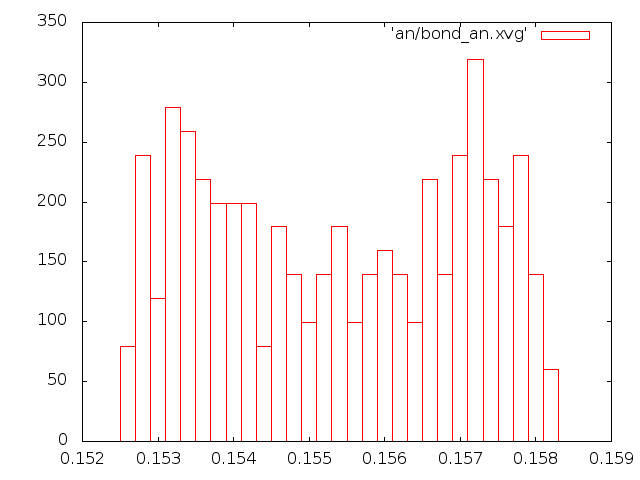
метод Андерсенa
метод Нуза-Хувера
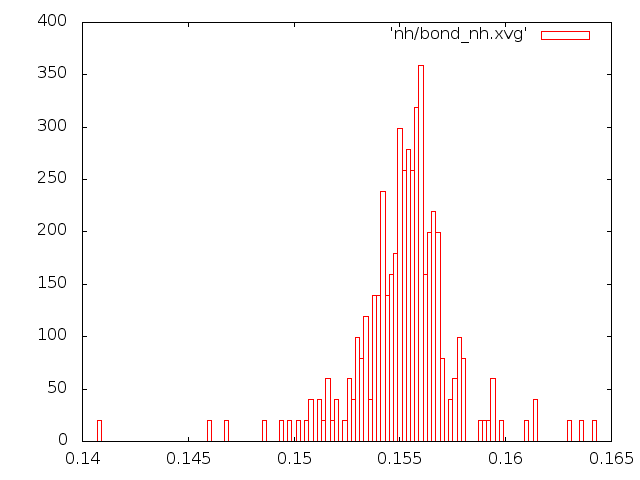
метод стохастической молекулярной динамики
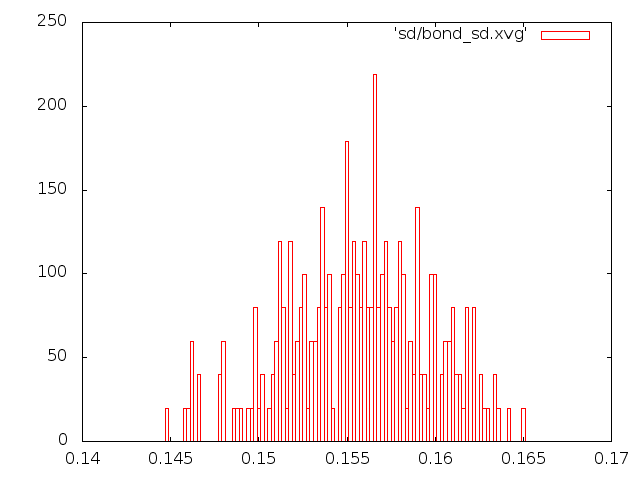
метод "Velocity rescale"