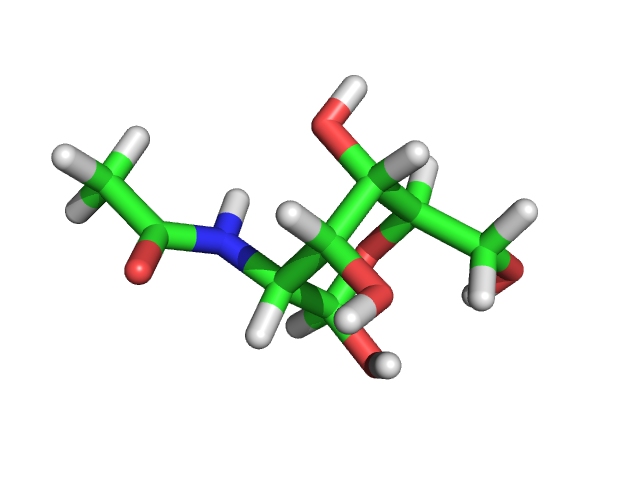
obgen nag.smi > nag.mol
babel -imol nag.mol -opdb nag.pdb
export PATH=${PATH}:/home/preps/golovin/progs/bin
vina --config vina.cfg --receptor seq.B99990005.pdbqt --ligand nag.pdbqt --out nag_prot.pdbqt --log nag_prot.log
Просмотрим файл nag_prot.log и запишем энергии 3 лучших расположений и геометрическую разницу между ними:
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.0 0.000 0.000
2 -5.0 1.733 4.956
3 -4.9 10.760 12.186
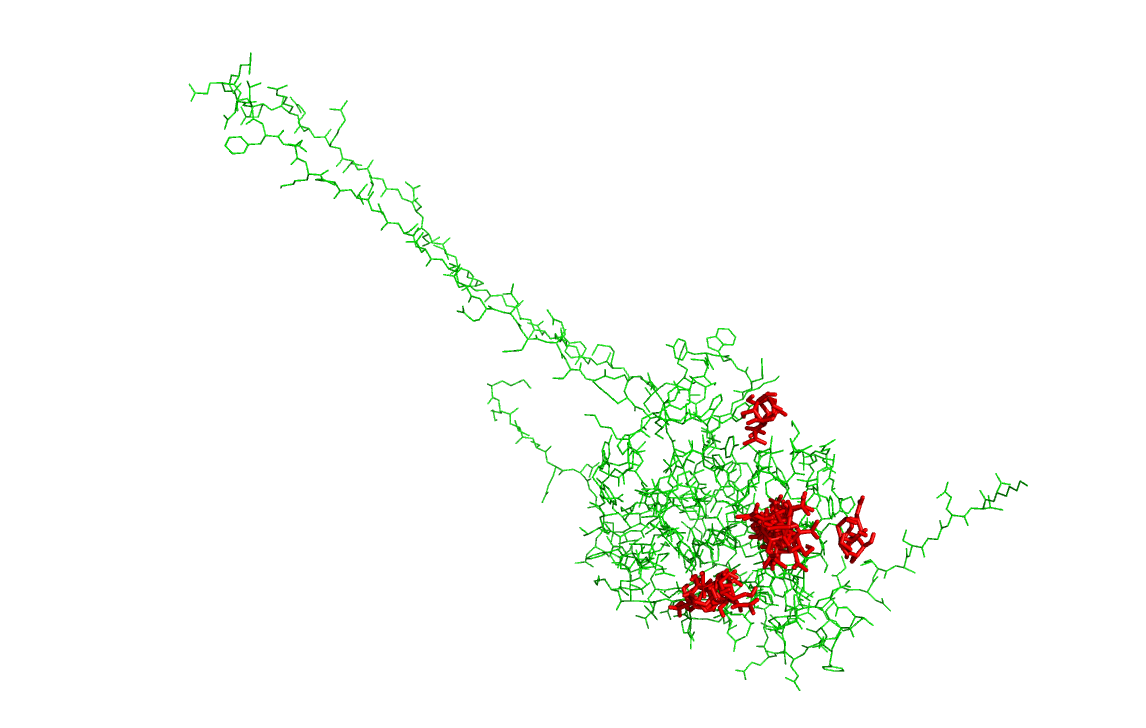
Файлы nag_prot.pdbqt и seq.B99990005.pdbqt были загружены в PyMOL. Все состояния на одной картинке изображены ниже:
python /usr/share/pyshared/AutoDockTools/Utilities24/prepare_flexreceptor4.py -r seq.B99990005.pdbqt -s ASN190_LYS194_ALA196_SER85
и проведём докинг:
vina --config vina.cfg --receptor prot_rigid.pdbqt --flex prot_flex.pdbqt --ligandM
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.0 0.000 0.000
2 -5.0 1.711 4.079
3 -4.8 2.573 4.561
Докинг с подвижными радикалами считаются немного дольше, чем докинг без подвижных радикалов. Файлы nag_prot_flex.pdbqt (отмечено лиловым) и seq.B99990005_rigid.pdbqt (отмечено красным) были загружены в PyMOL (зеленым покрашено seq.B99990005.pdbqt, синим - nag_prot.pdbqt):
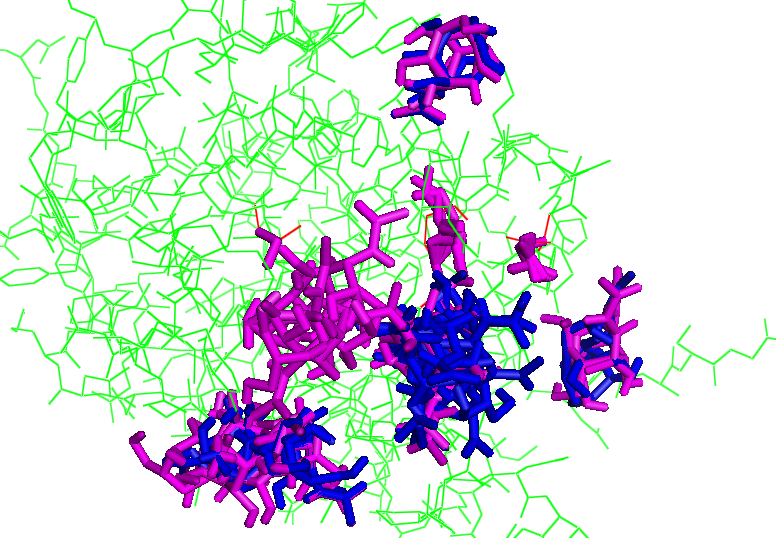
В данном случае свое положение меняет не только лиганд, но и три аминокислоты белка (Asn190, Lys194, Ser85). С биологической точки зрения лучше делать подвижный докинг, поскольку белок не является неподвижной жесткой структурой.
OH (nag_oh.smi)
NH2 (nag_nh2.smi)
H (nag_h.smi)
Ph (nag_Ph.smi)
Для каждого из этих лигандов проведен обыкновенный докинг:
OH (nag_prot_oh.log, nag_prot_oh.pdbqt)
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.0 0.000 0.000
2 -4.9 2.557 3.676
3 -4.7 10.416 11.883
NH2 (nag_prot_nh2.log, nag_prot_nh2.pdbqt)
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.3 0.000 0.000
2 -5.2 2.439 4.918
3 -4.9 2.639 4.788
H (nag_prot_h.log, nag_prot_h.pdbqt)
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.3 0.000 0.000
2 -5.2 2.439 4.918
3 -4.9 2.639 4.788
Ph (nag_prot_ph.log, nag_prot_ph.pdbqt)
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -6.0 0.000 0.000
2 -6.0 21.997 24.407
3 -5.9 3.225 5.290
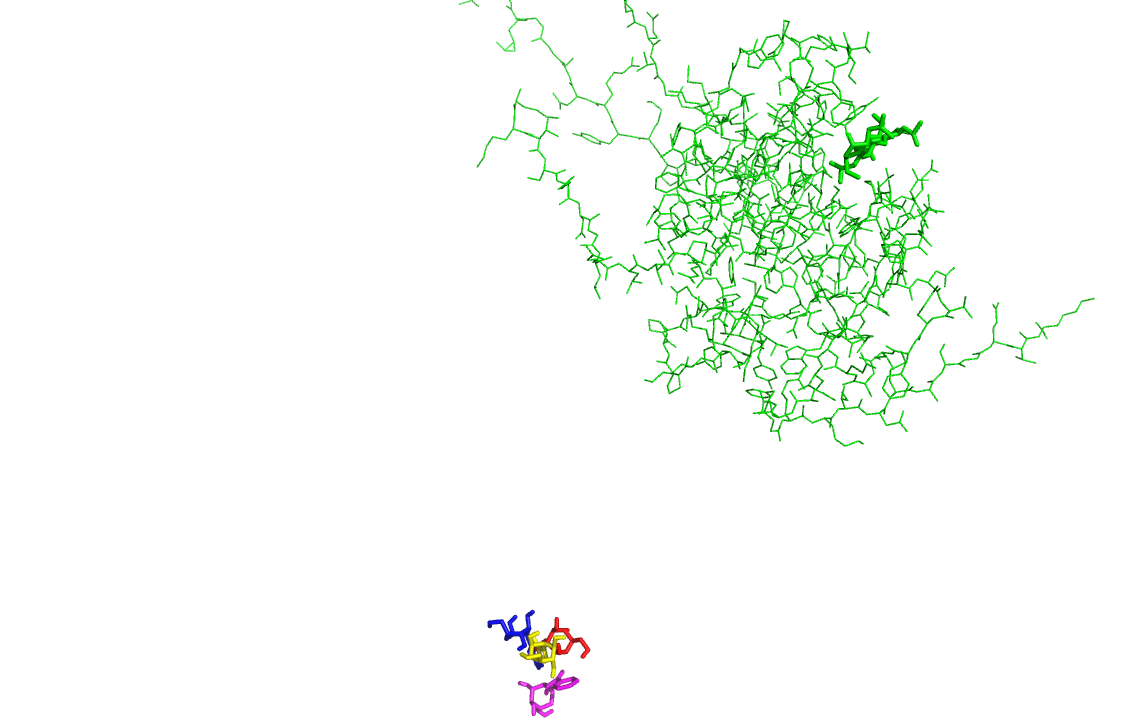
Красным отмечен лиганд с Н, синим - с NH2, желтым - с OH, лиловым - с Ph, зеленым - результат моделирования. На картинке видно, что докинг прошел неудачно.
OH (nag_oh_prot_flex.log, nag_oh_prot_flex.pdbqt)
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.1 0.000 0.000
2 -5.0 1.396 3.962
3 -4.9 1.855 3.859
NH2 (nag_nh2_prot_flex.log, nag_nh2_prot_flex.pdbqt)
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.2 0.000 0.000
2 -5.2 1.960 3.965
3 -4.7 8.287 10.354
Ph (nag_ph_prot_flex.log, nag_ph_prot_flex.pdbqt)
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -6.0 0.000 0.000
2 -5.9 12.918 20.835
3 -5.8 12.923 20.115
OH
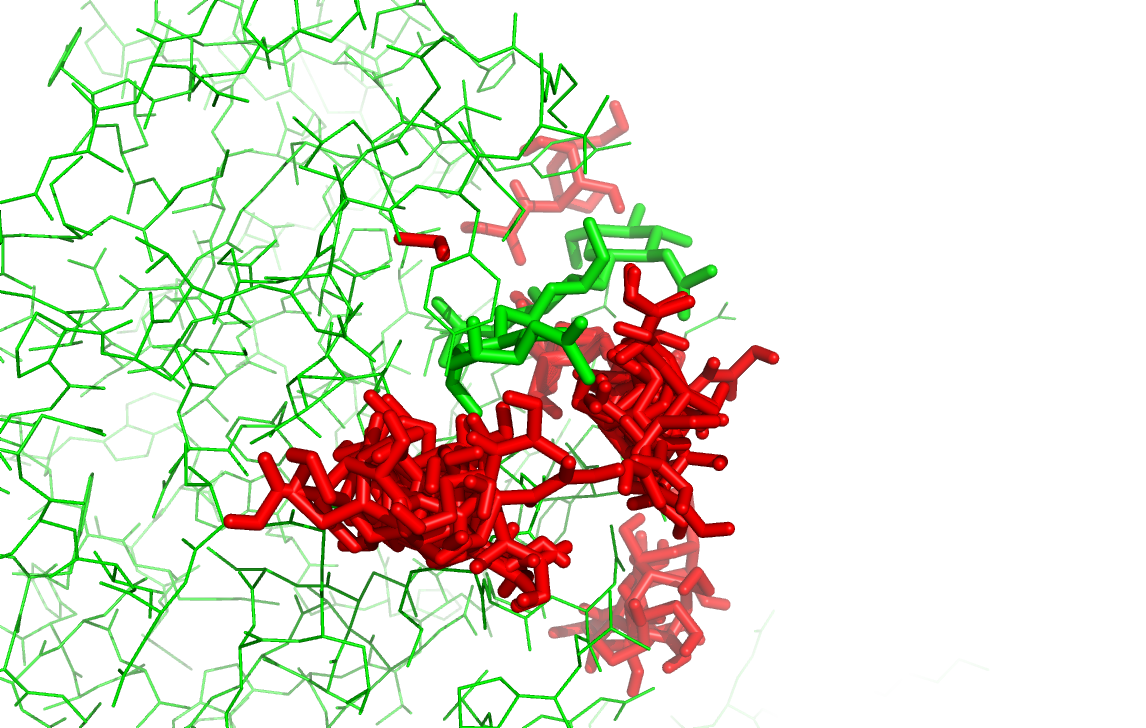 NH2
NH2 Ph
Ph 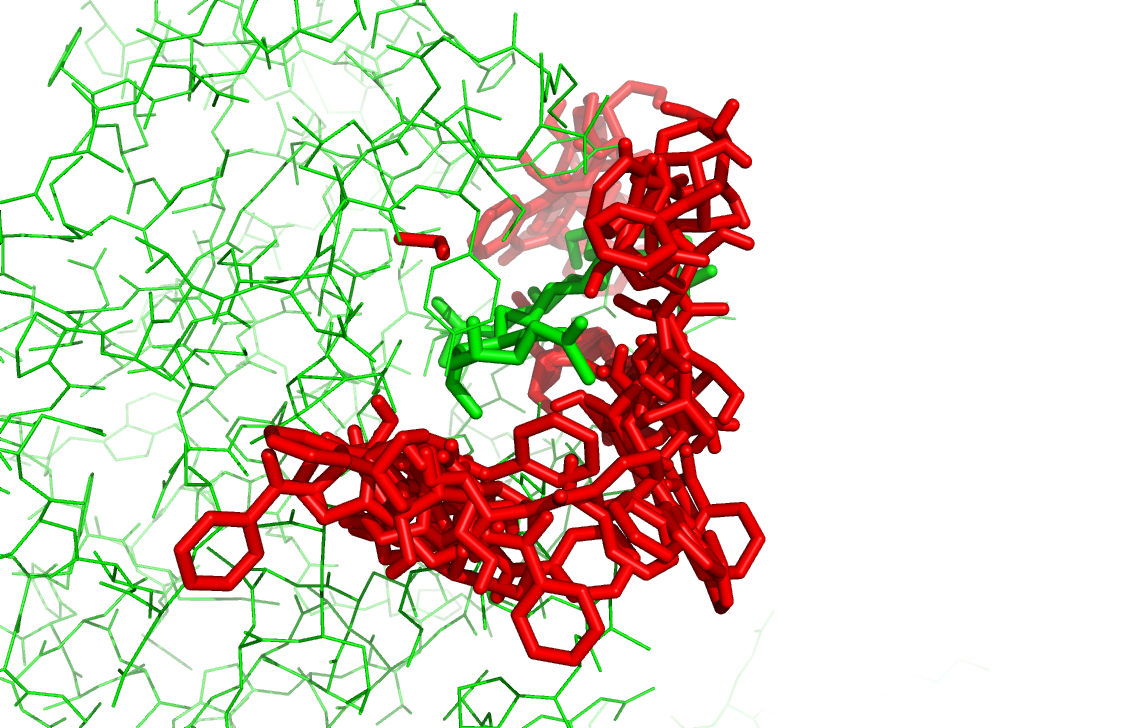
Видно, что одни лиганды помещаются в белке более глубоко, а другие менее. Наиболее компактно располагается лиганд с ОН, хотя афинность у лиганда с Ph наилучшая.
Последние два пункта подтверждают предположение о том, что докуинг целесообразнее проводить для подвижного белка.