Cравнительный анализ канонической ДНК и стеблей тРНК
Задание 1
Контиг: CP002687.1. Длина: 18585056 п.н. Выдача состоялда лишь из 5 вариантов, из которых это самый короткий. Ниже показана схема расположения генов на данном контиге. Взят участок 9311М - 9313М. Длина участка: 2000 п.н.
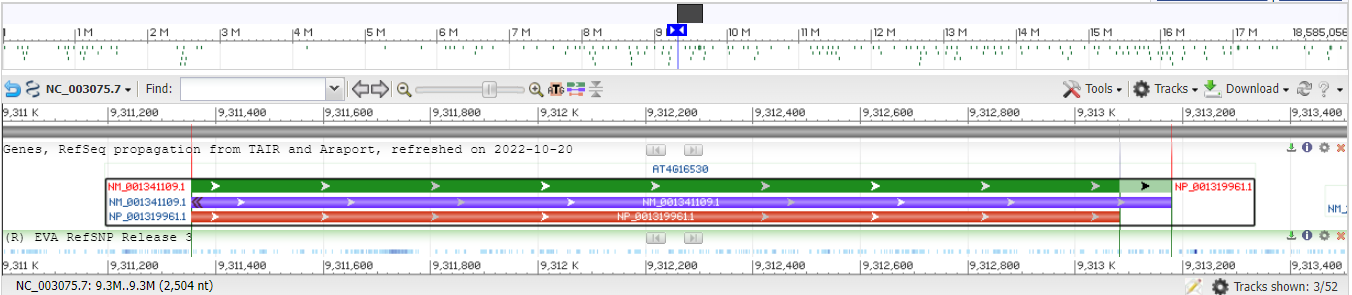
Зеленым цветом обозначен геном, светло-зеленым показана нетранслируемая область. Фиолетовым - мРНК; Красным - CDS.
Fasta файл с участком последовательности контига.
Далее был осуществлен поиск BLAST по эукариотическим последовательностям для контига. Исключил таксон Arabidopsis, типы используемых BLAST:
1. blastn - используется, если нужно получить нуклеотидные последовательности, гомологичные данной. При этом запрос не транслируется и поиск идет по нуклеотидной базе данных. Например, нужно найти гомологи гена тРНК.
Параметры и результаты: якорь - 11, максимальное количество находок - 1000. Находки - 418. Был найден данный участок в хромосомах других организмов. Найдено предсказанных фрагментов - 124.
2. megablast - используется, когда известно, какому организму принадлежит секвенированная последовательность, т.к. алгоритм быстро ищет высоко сходные последовательности.
Параметры и результаты: длина слова 28, максимальное количество находок - 100. Находки - 44. Предсказанных последовательностей большинство.
3. blastx - получает нуклеотидную последовательность и возвращает гомологичные белки, транслируя запрос. Есть ген, нужно найти среди эукариот гомологи белка, который ген кодирует.
Параметры и результаты: длина слова 6, максимальное количество находок - 100. После результатов изменил макс кол-во находок на 5000. Находки - 190. Много неохарактеризованных белков, присутствуют предсказанные последовательности.
4. tblastx - получает нуклеотидную последовательность и находит гомологичные варианты, транслируя запрос. Есть нуклеотидная последовательность, сравниваем с нуклеотидной, ищем гомологичные гены.
Параметры и результаты: перебрал разные варианты параметров (кол-во находок, длина якоря и.т.д), но поиск результатов не дал
Задание 2
В установленом на свой ПК BLAST+ проиндексировал последовательность генома Arabidopsis thaliana с помощью команды:l
>makeblastdb -in genomic.fna -dbtype nucl
После чего был использвоан blastn (параметры по умолчанию) с последовательностями 16S и 23S рРНК Escherichia coli. Выбрал blastn, потому что Arabidopsis thaliana и Escherichia coli двольно далеки друг от друга эволюционно, следовательно имеют сильные различия в пос-тях рРНК. Для выравнивания использовались команды:
>blastn -task blastn -query 16S_rRNA_ecoli.fna -db genomic.fna -out 16S_rRNA_ecoli.out
>blastn -task blastn -query 23S_rRNA_ecoli.fna -db genomic.fna -out 23S_rRNA_ecoli.out
Для 16S нашлось около 20 хитов. Часть из них стоит исключить, опираясь на e-value, не будем брать во внимание находки с e-value больше 0.05. Итого вышло 11 гомологов. Лучшая локализована в хлоропласте. Что ожидаемо, т.к. 16S у эукариот встречается в составе малых субъединиц рибосом в органеллах (в частности в плазмидах).
Для 23S рРНК насчиталось около 40 находок. 12 из них не подходят из-за высокого значения e-value. Лучшая также располагается в хлоропласте, что тоже ожидаемо.