База данных OPM
Для выполнения взял белок внешней мембраны грам-отрицательных бактерий (Uniprot OMPA, PDB - 1bxw)
| Толщина гидрофобной части | 25,2Å |
| Координаты трасмембранных участков | 1(7- 15), 2(35- 44), 3(51- 58), 4(76- 85), 5(93- 101), 6(120- 130), 7(137- 145), 8(160- 169) |
| средняя длина трасмембранного региона | 8 |
| Положение | Внешняя мембрана Грам-отрицательной бактерии |
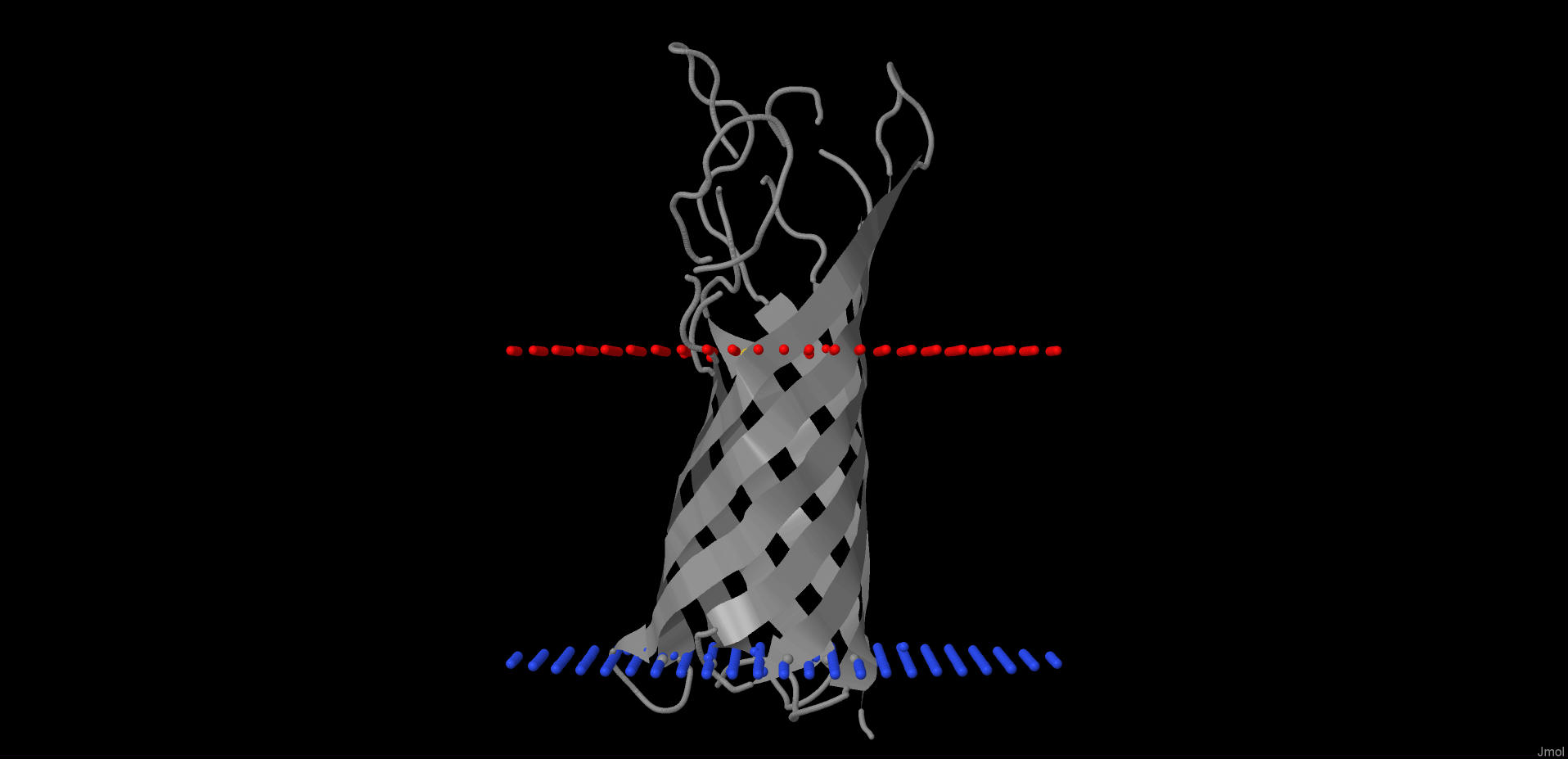
Красным цветом часть со стороны межмебранного пространства, синимс торона цитоплазмы
DeepTMHMM
Сервис DeepTMHMM позволяет предсказывать трансмембранные участки по первичной структуре белка. Сначала я загрузил последовательность белка TSGBD_HALVD. Результаат ниже:
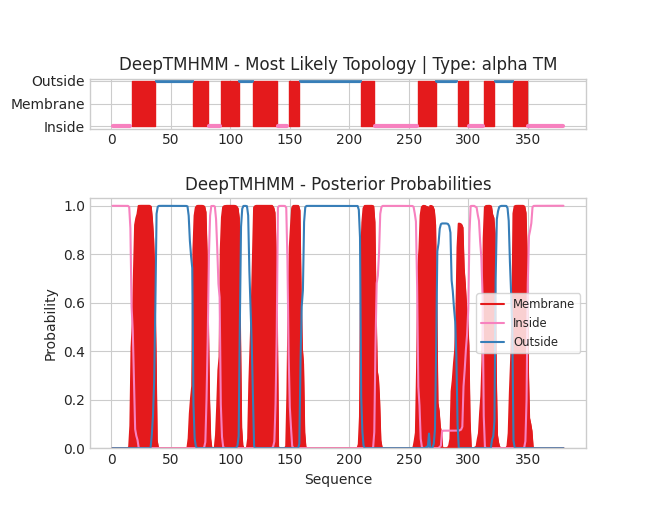
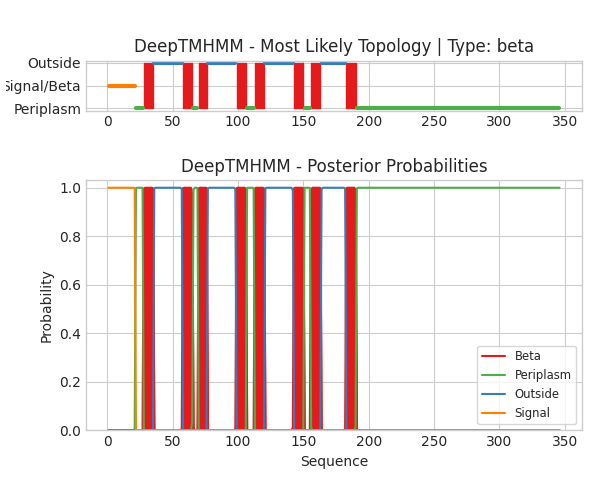
Характеристика графической выдачи: на оси ординат предсказания положения участков относительно мембраны (верхний маленький график). На нижнем на вертикали вероятность какого-либо положения остатка относительно мембраны. На оси абсцис координаты остатков.
Для альфа белка сервис предсказал 10 трансмембранных участков, однако один из участков в отличии от других имеет вероятность немного ниже, в целом качество предсказания это сильно не испортило. Для бета-листового белка программа предсказала 8 трансмембранных структур, что совпадает с информацией в БД OPM.
PPM
Вспомним что мне был выдан TSGBD_HALVD, являющийся транспортным белком глюкозы ABC у Haloferax volcanii (археи). На этот раз его последовательность я- Number of Membranes: 1
- Type of membrane: Archaebacteria cell membrane
- Allow curvature: no
- Topology (N-ter): in (по DeepTMHMM)
- Include heteroatoms, excluding water and detergents, for positioning in membrane: no (по UniProt)
| Толщина гидрофобной части | 29.4 ± 0.8 ÅÅ |
| Координаты трасмембранных участков | 1(15- 36), 2(62- 85), 3(90- 103), 4(114- 142), 5(147- 162), 6(210- 227), 7(253- 274), 8(290- 305), 9(306- 322),10(336- 355) |
| средняя длина трасмембранного региона | 18.3 |
| Положение | Внешняя мембрана архей |
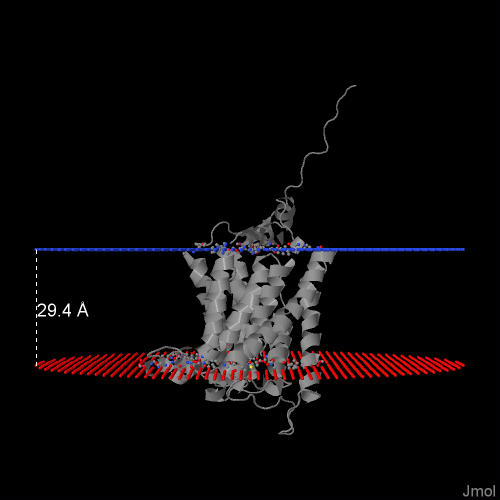
Сравнение результатов
Сначала рассмотрим выдачи DeepTMHMM и PPM для TSGBD_HALVD. Колличество трансмембранных участков вышло одинаковым, в обоих случаях по 10. Предсказание структуры из Uniprot имеет выскую достоверность т.к. почти все участки имеют больше 90% достоверности. Концевые участки с сильно низким показателем достоверности также оказались схожими с предсказанием.
Далее выдачи DeepTMHMM (координаты: 1(28-35) 2(58-65) 3(70-76) 4(99-106) 5(113-120) 6(143-150) 7(156-163) 8183-190) и БД OPM для OMPA. Количество участков хоть и совпадает, но имеют сильно отличающиеся координаты, это вероятно связано, что для сервиса DeepTMHMM последовательность была взята из БД Uniprot, т.к. она содержит в себе сигнальный пептид (оранжевый график на выдаче). На длину этого пептида скорее всего и сдвинуты координаты.