Практикум 4
Примечание: я использовал общий список бактерий при составлении базы данных, поэтому я по неосторожности, кроме всех своих, добавил лишнюю бактерию (мнемоника: STRAW), и в дальнейшем работал с ней.
Для удобства продублирую часть первого практикума с выбраными актинобактерияии и деревомНазвание |
Мнемоника |
Acidothermus cellulolyticus |
ACIC1 |
Arthrobacter sp. |
ARTS2 |
Bifidobacterium longum |
BIFLO |
Clavibacter michiganensis |
CLAMS |
Corynebacterium diphtheriae |
CORDI |
Corynebacterium efficiens |
COREF |
Leifsonia xyli |
LEIXX |
Поиск гомологов
Поиск гомологов CLPX_ECOLI осуществлялся командами:
cat ACIC1.fasta ARTS2.fasta BIFLO.fasta CLAMS.fasta CORDI.fasta COREF.fasta LEIXX.fasta STRAW.fasta > teo.fasta
makeblastdb -dbtype prot -in teo.fasta -out ind_db
blastp -query P0A6H1.fasta -num_threads 4 -db ind_db -evalue 0.001 -out blastp.txt
По результатам выводим таблицу
| Blast res | Score(Bits) | e-value |
|---|---|---|
| sp|Q820F8|CLPX_STRAW ATP-dependent Clp protease ATP-binding subun... | 535 | 0.0 |
| sp|Q8FN57|CLPX_COREF ATP-dependent Clp protease ATP-binding subun... | 518 | 0.0 |
| sp|Q6NFU7|CLPX_CORDI ATP-dependent Clp protease ATP-binding subun... | 518 | 0.0 |
| sp|A0LSV2|CLPX_ACIC1 ATP-dependent Clp protease ATP-binding subun... | 517 | 0.0 |
| sp|A0JXL2|CLPX_ARTS2 ATP-dependent Clp protease ATP-binding subun... | 516 | 0.0 |
| sp|Q6AFZ6|CLPX_LEIXX ATP-dependent Clp protease ATP-binding subun... | 509 | 0 |
| sp|B0RAS4|CLPX_CLAMS ATP-dependent Clp protease ATP-binding subun... | 491 | 1e-173 |
| sp|Q8G5R1|CLPX_BIFLO ATP-dependent Clp protease ATP-binding subun... | 432 | 9e-150 |
| tr|A0K1M3|A0K1M3_ARTS2 ATPase AAA-2 domain protein OS=Arthrobacte... | 54.3 | 5e-08 |
| tr|Q8G871|Q8G871_BIFLO Protease OS=Bifidobacterium longum (strain... | 51.2 | 7e-07 |
| tr|Q8FMH5|Q8FMH5_COREF Putative endopeptidase Clp ATP-binding cha... | 47.0 | 1e-05 |
| tr|Q6NFB1|Q6NFB1_CORDI ATP-dependent Clp protease ATP-binding sub... | 45.8 | 3e-05 |
| tr|A0LW31|A0LW31_ACIC1 AAA ATPase, central domain protein OS=Acid... | 45.4 | 4e-05 |
| tr|A0LRB8|A0LRB8_ACIC1 ATP-dependent zinc metalloprotease FtsH OS... | 43.5 | 2e-04 |
| sp|Q8G6B7|RUVB_BIFLO Holliday junction ATP-dependent DNA helicase... | 42.7 | 2e-04 |
| sp|A0LR74|FTSH_ACIC1 ATP-dependent zinc metalloprotease FtsH OS=A... | 43.1 | 2e-04 |
| tr|Q6ACQ0|Q6ACQ0_LEIXX ATP-dependent zinc metalloprotease FtsH OS... | 43.1 | 2e-04 |
| tr|Q8G3S2|Q8G3S2_BIFLO ATP-dependent zinc metalloprotease FtsH OS... | 43.1 | 2e-04 |
| sp|A0JXB1|RUVB_ARTS2 Holliday junction ATP-dependent DNA helicase... | 42.4 | 3e-04 |
| tr|Q82QV8|Q82QV8_STRAW Putative AAA family ATPase OS=Streptomyces... | 41.6 | 3e-04 |
| tr|A0JR82|A0JR82_ARTS2 ATP-dependent zinc metalloprotease FtsH OS... | 41.6 | 5e-04 |
| tr|A0K236|A0K236_ARTS2 AAA ATPase, central domain protein OS=Arth... | 41.6 | 5e-04 |
| tr|Q82EE9|Q82EE9_STRAW ATP-dependent zinc metalloprotease FtsH OS... | 41.6 | 5e-04 |
| tr|Q82EB8|Q82EB8_STRAW Putative ATP-dependent Clp protease OS=Str... | 41.6 | 7e-04 |
| tr|B0RHW4|B0RHW4_CLAMS ATP-dependent zinc metalloprotease FtsH OS... | 41.2 | 8e-04 |
| tr|Q6NF92|Q6NF92_CORDI ATP-dependent zinc metalloprotease FtsH OS... | 40.8 | 0,001 |
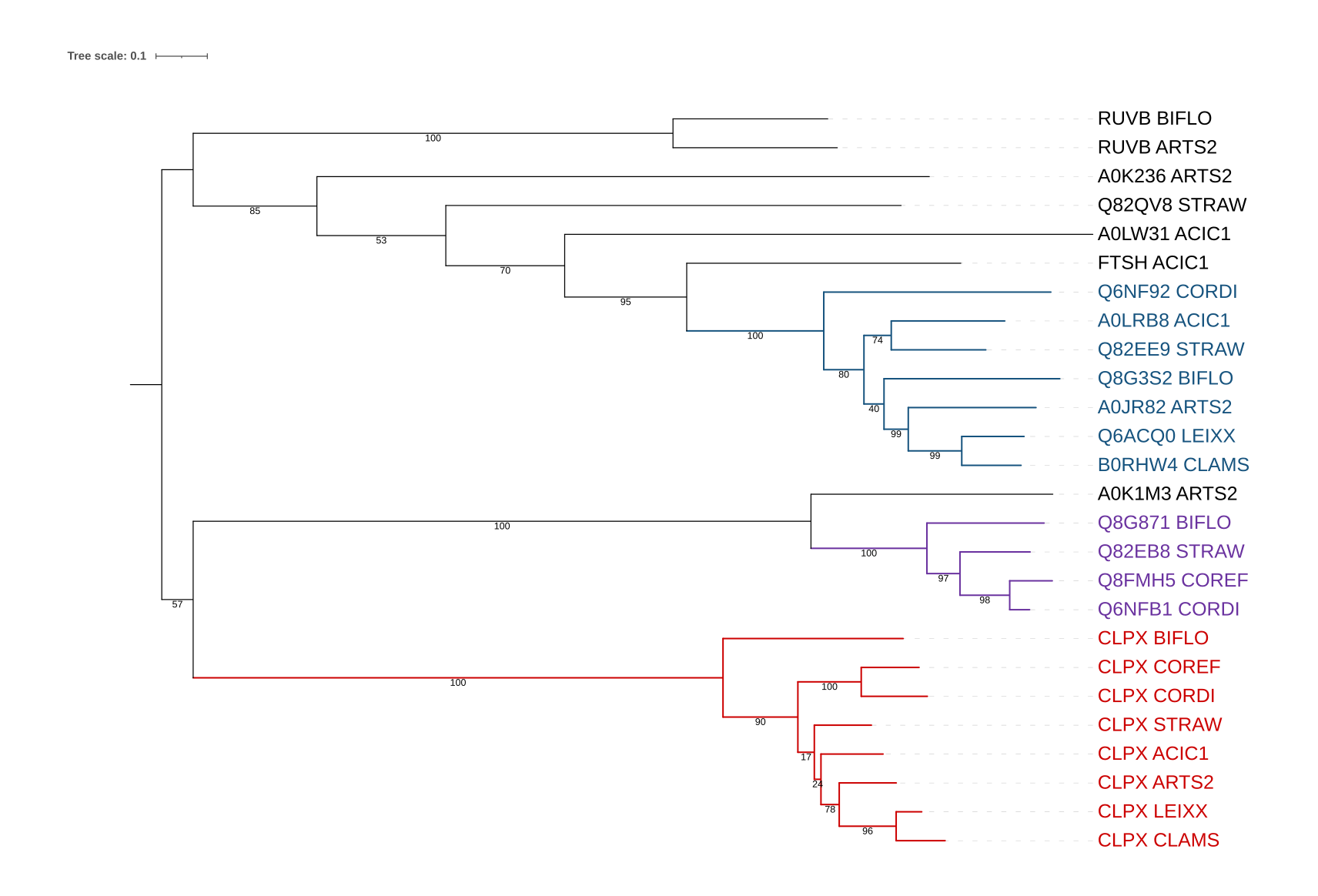
Реконструкция филогении
Реконструктция проводилась программой FastMe на сайте NGPhylogeny.fr с рекомендоваными параметрами. Результат представлен ниже:
Скобочная формула
Примеры парологов: CLPX_BIFLO и RUVB_BIFLO; CLPX_ACIC1 и FTSH_ACIC1; Q82EE9_STRAW и CLPX_STRAW. Примеры ортологов: CLPX_STRAW и CLPX_ACIC1; Q8G3S2_BIFLO и A0JR82_ARTS2; Q82EB8_STRAW и Q8FMH5_COREF
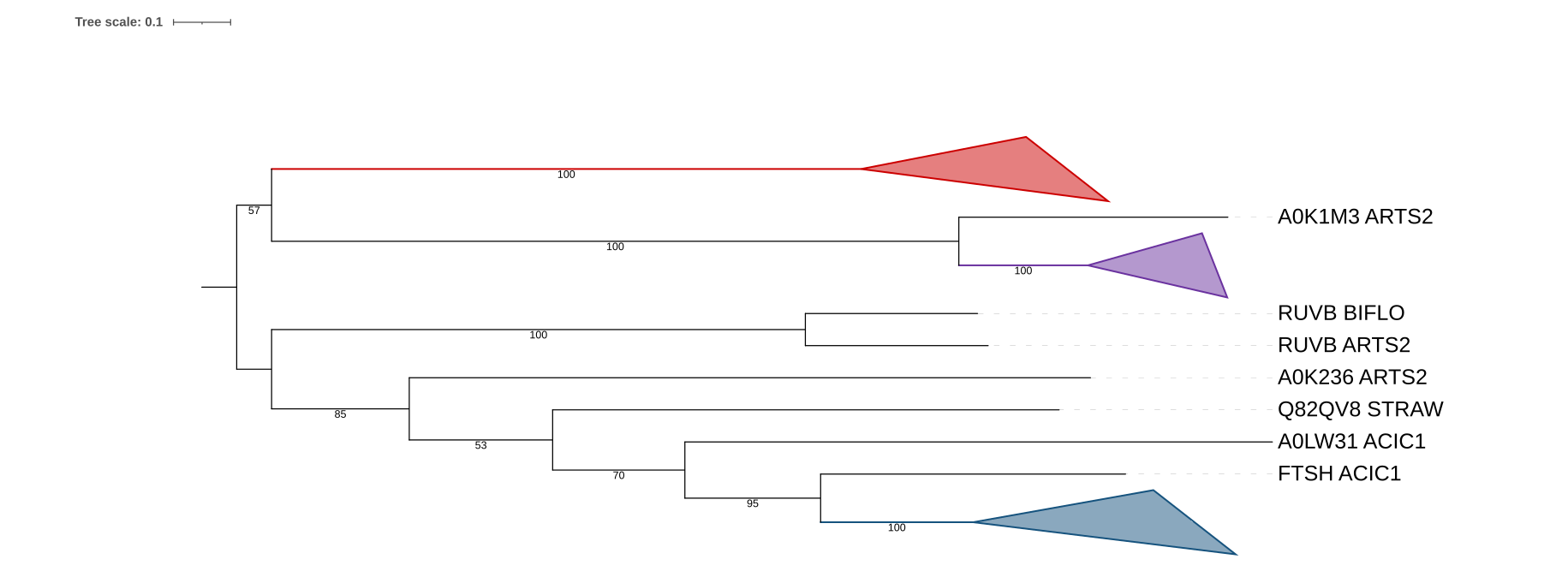
Видим три крупные группы:
- Красный цвет: группа ClpX АТФ-связывающих субъединиц протеаз Clp. Ветвь включает в себя все исходные белки бактерии (лишней в том числе). Ветви с исходным деревом схожие, к примеру ветви объеденяющие белки LEIXX и CLAMS/CORDI и COREF.
- Фиолетовый цвет: группа АТФ-зависимых цинковых металлопротеаз. Сходства в филогении есть, но белки вошли не все.
- Синий цвет : белки связанные с Clp-протеазами. В группу вошла большая часть белков, в филогении бактерий есть отличия.