Одним из организмов для выравнивания я, недолго думая, решил выбрать Staphylococcus aureus, как один из самых популярных видов бактерий. Затем я решил строить выравнивания с помощью blastn и megablast, двигаясь к уменьшению близости между видами по филогенетическому дереву рода Staphylococcus. Для этого были использованы данные статьи Lamers RP et al. "Phylogenetic relationships among Staphylococcus species and refinement of cluster groups based on multilocus data" (doi: 10.1186/1471-2148-12-171).
Выравнивание я делал по единственной хромосоме соответствующих организмов, AC которых искал с помощью новой версии сайта NCBI, выбирая уровень сборки не ниже хромосомного.
Однако в процессе этих выравниваний внутри рода Staphylococcus, я обнаружил, что даже самые далекие в нем виды от Staphylococcus aureus с помощью megablast выравниваются довольно хорошо, и значительных отличий от blastn нет.
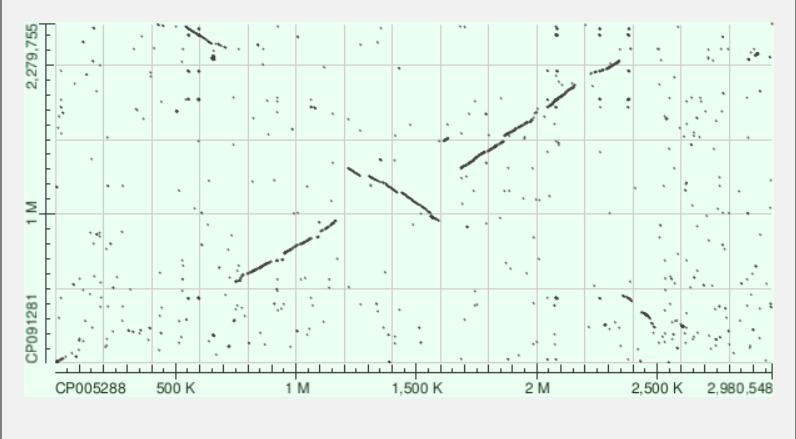
Тогда я решил посмотреть выравнивание Staphylococcus aureus с видом близкого к Staphylococcus рода Macrococcus (а именно Macrococcus canis). Результаты мне показались подходящими, их я и привожу ниже:

Из преведенных выше данных видно, что хорошо выравниваются последовательности на участке от 500К до 2500К п.н. хромосомы Staphylococcus aureus. Этот участок выравнивается на большую часть хромосомы Macrococcus canis. Предполагаю, что внутри этого большого участка произошло целых 3 инверсии: сначала большая (весь сходный участок), потом поменьше внутри первой (750K-2450K п.н. хромосомы Staphylococcus aureus), а затем "маленькая" внутри первых двух (1200К-1600К п.н. хромосомы Staphylococcus aureus). Также в хромосомах обоих видов можно заметить вставки (например, на участке сходства самая крупная - 1600К-1690К п.н. хромосомы Staphylococcus aureus
Также можно отметить, что точка начала хромосомы (то есть где "мысленно" разрезать кольцевую бактериальную хромосому), а также цепь у обоих видов были выбраны одинаково. Это можно заключить из сходства участка около 0 п.н. обеих хромосом и из общей выровненности вдоль главной диагонали (за исключением, конечно, участков негомологий и инверсий). Именно из этого я сделал вывод, что произошло именно 3 инверсии, а не 2 при разных выбранных цепях (как можно было бы подумать не будь сходного участка в районе 0 п.н.)