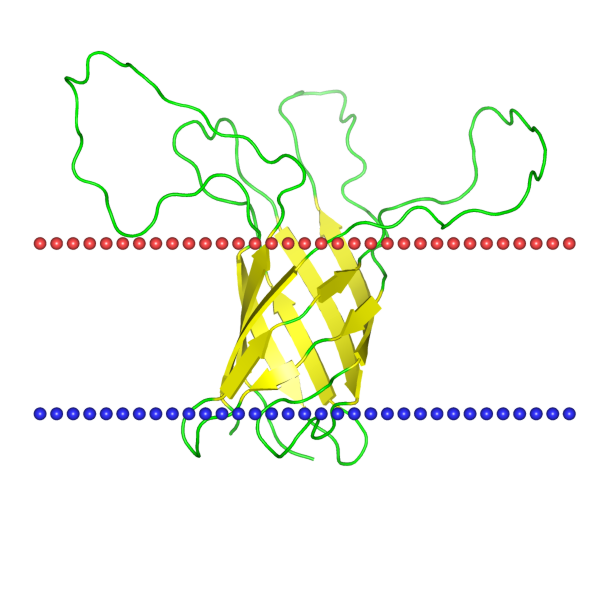
С помощью поиска по уровням классификации в базе данных OPM я выбрал из белков, содержащих бета-бочки белок Outer membrane protein H (OprH) - белок внешней мембраны H. Он интересен тем, что отвечает за лекарственную устойчивость у Pseudomonas aeruginosa. Его идентификаторы PDB и Uniprot: 2LHF и Q51486_PSEAI соответственно. Полученные из OPM параметры и изображение белка приведены в Таблице 1 и на Рисунке 1:
Таблица 1. Некоторые параметры белка OprH
| Толщина гидрофобной части белка в мембране | 20.6 Å |
| Координаты трансмембранных участков | 5-12, 41-48, 55-60, 74-83, 91-101, 119-130, 137-144, 170-177 |
| Среднее количество остатков в одном β-тяже белка | 8.9 |
| В какой мембране находится белок | внешняя мембрана Грам- бактерии |

Для выданного мне α-спирального белка и для выбранного в задании №1 β-листового белка был запущен сервис DeepTMHMM. Результаты текстовой выдачи программы доступны по ссылкам ниже. Графические результаты приведены на Рисунках 2-3.
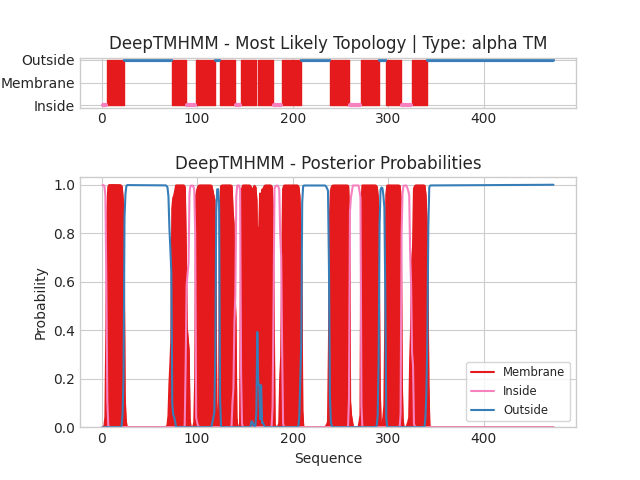
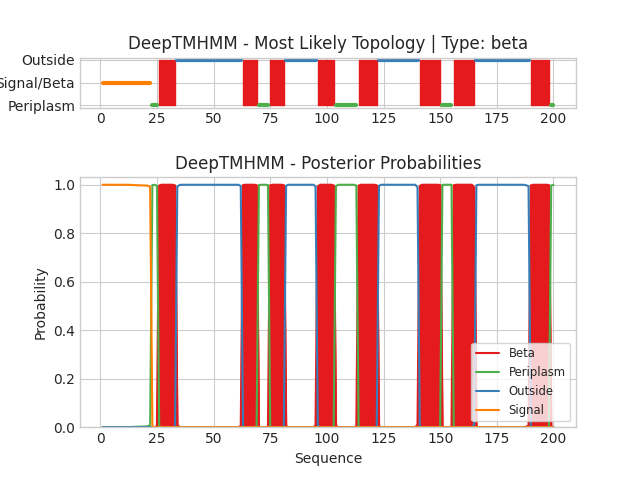
Мне достался белок с идентификатором RGTD_RHIL3. Он представляет собой Lipid A galacturonosyltransferase RgtD - Липид А галактуронозилтрансфераза. При запустке алгоритма были использованы следующие параметры:
Результаты работы программы приведены в Таблице 2 и на Рисунке 4.
Таблица 2. Некоторые параметры белка RgtD
| Толщина гидрофобной части белка в мембране | 30.5 ± 1.0 Å |
| Координаты трансмембранных участков | 5-23, 70-91, 100-122, 123-138, 146-164, 165-179, 187-206, 232-259, 271-290, 295-310, 319-341 |
| Среднее количество остатков в одном β-тяже белка | 20.1 |
| В какой мембране находится белок | внутренняя мембрана Грам- бактерии |
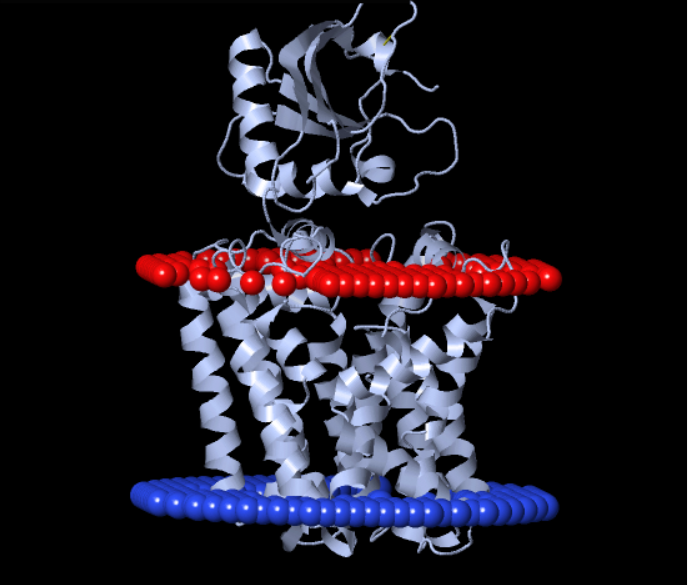
В целом результаты предсказания трансмембранных спиралей выданного мне белка совпадают у алгоритмов DeepTMHMM и PPM. Всего найдено 11 спиралей. Точные координаты спиралей, однако, могут варьировать на несколько аминокислотных остатков. В базе данных OPM выданный мне белок RgtD я не нашел. Модель белка в Uniprot достоверно предсказывает трансмембранные спирали, есть предсказания с низкой достоверностью в периплазматических участках белка. Я думаю, что достоверность модели оказывает влияние на результат работы PPM, т.к. я использовал именно эту модель в качестве входных данных для PMM.