Протеомы выбранных бактерий были объединены в один файл и этот файл был проиндексирован программой makeblastdb. Далее был проведен поиск программой blastp гомологов белка CLPX_ECOLI с порогом на E-value 0,001. Список находок приведен ниже (в скобках приведено значение E-value):
Q1AVT0|Q1AVT0_RUBXD (0.0)
P9WPB9|CLPX_MYCTU (0.0)
Q6NFU7|CLPX_CORDI (0.0)
A0LSV2|CLPX_ACIC1 (0.0)
Q47MU4|Q47MU4_THEFY (0.0)
Q6AFZ6|CLPX_LEIXX (0.0)
Q8G5R1|CLPX_BIFLO (5.91e-150)
Q1AU05|Q1AU05_RUBXD (2.03e-07)
Q8G871|Q8G871_BIFLO (4.53e-07)
Q6NFB1|Q6NFB1_CORDI (2.24e-05)
A0LW31|A0LW31_ACIC1 (2.44e-05)
Q47MZ2|Q47MZ2_THEFY (7.50e-05)
A0LRB8|A0LRB8_ACIC1 (1.06e-04)
Q1AY82|Q1AY82_RUBXD (1.07e-04)
Q8G6B7|RUVB_BIFLO (1.14e-04)
A0LR74|FTSH_ACIC1 (1.29e-04)
Q6ACQ0|Q6ACQ0_LEIXX (1.29e-04)
Q8G3S2|Q8G3S2_BIFLO (1.29e-04)
Q47KU4|Q47KU4_THEFY (3.25e-04)
P9WQN3|FTSH_MYCTU (4.72e-04)
Q1AV13|FTSH_RUBXD (6.58e-04)
Q6NF92|Q6NF92_CORDI (6.90e-04)
Q6NGK1|Q6NGK1_CORDI (8.35e-04)
Q1ATZ9|Q1ATZ9_RUBXD (8.67e-04)
FASTA файл был получен с помощью JalView по выдаче blastp. Формула (Newick) дерева полученного с помощью FastME доступна по ссылке ниже:
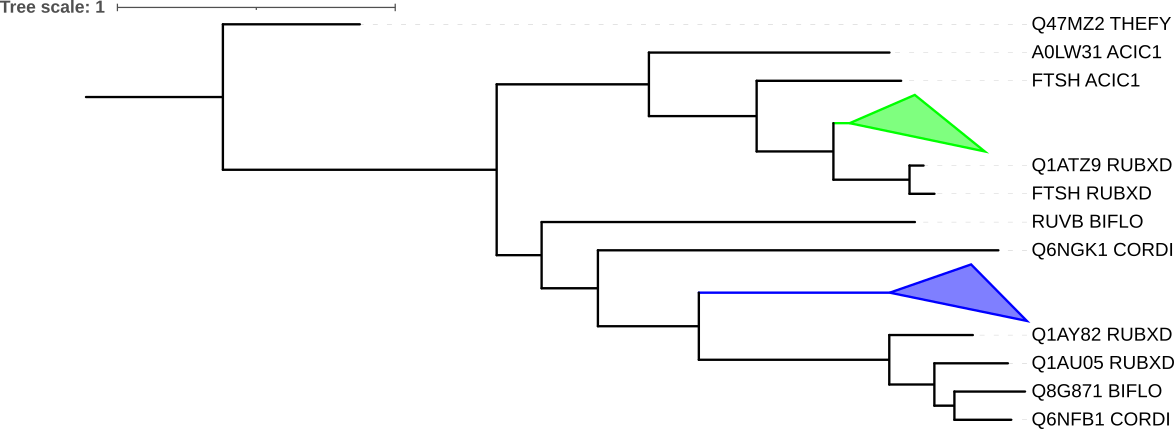
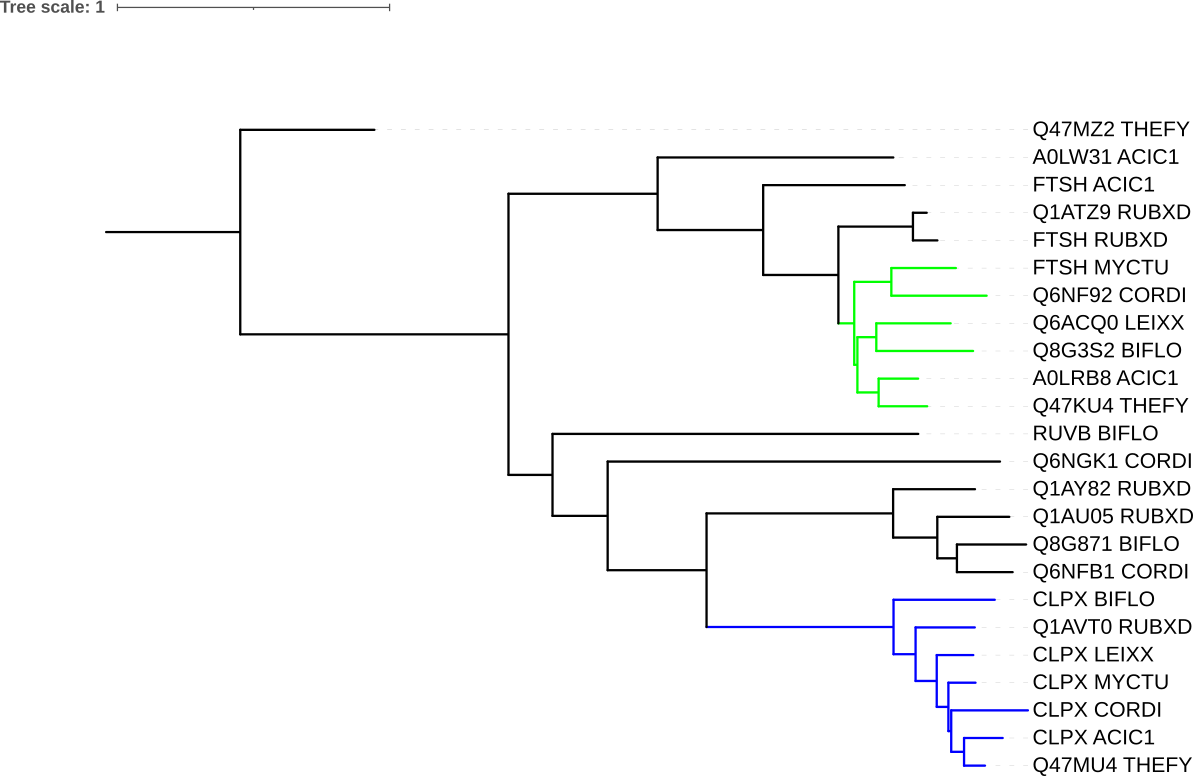
Паралоги: Q1AU05_RUBXD и Q1AY82_RUBXD, Q6NFB1_CORDI и CLPX_CORDI, Q8G871_BIFLO и CLPX_BIFLO
Ортологи: Q47MU4_THEFY и CLPX_ACIC1, Q8G871_BIFLO и Q6NFB1_CORDI, FTSH_MYCTU и Q6NF92_CORDI
Ниже представлено 2 изображения полученного дерева: с окраской ортологических групп (более 3 видов) цветом и со сворачиванием этих ортологических групп.