В качестве сигнала в геноме для описания я выбрал теломерные повторы. Это короткие повтоярющиеся последовательности (у млекопитающих, например, последовательность TTAGGG [1]), которые необходимы для маркировки концов хромосом. Этот сигнал адресован теломер-связывающим белкам, которые свяжутся с этими последовательностями и, с помощью других привлеченных белков, сформируют структуру, называемую Т петля, которая защищает концы хромосом. Стоит отметить, что теломерные повторы это сильные сигналы, т.к. каждый теломерный регион в наших хромосомах защищен.
Сслыка на источники информации:
1 - Červenák F, Juríková K, Sepšiová R, Neboháčová M, Nosek J, Tomáška L. Double-stranded telomeric DNA binding proteins: Diversity matters. Cell Cycle. 2017;16(17):1568-1577. doi: 10.1080/15384101.2017.1356511. Epub 2017 Jul 27. PMID: 28749196; PMCID: PMC5587031.
В качестве сигнала я выбрал последовательность Шайна-Дальгарно бактерии Bacillus Subtilis штамма SRCM103923. Были скачаны последовательность генома и хромосомная таблица и по ним, с помощью скрипта, любезно предоставленного Андреем Малышевым, были получены последовательности длиной 20 нуклеотидов перед генами. Последовательность анти-Шайна_Дальгарно у Bacillus Subtilis - ССТССТ, по принципу комплементарности были отобраны те последовательности из регионов перед белок-кодирующими генами, которые имеют не больше 1 некомплементарной пары. Это было сделано с помощью моего скрипта, доступного по ссылке:
Скрипт для отбора сигналовЭти данные представляют собой обучающую выборку и на их основе была построена матрица PWM с помощью моего скрипта по ссылке ниже (в качестве значения псевдокаунтов было выбрано число 0.1 для каждого нуклеотида):
Скрипт для построения PWMТаблица 1. Матрица PWM для последовательности Шайна-Дальгарно бактерии Bacillus Subtilis
| Баз. частоты | 1 | 2 | 3 | 4 | 5 | 6 | |
| A | 0.281 | 1.06 | -1.50 | -1.70 | 1.16 | -1.78 | -1.06 |
| G | 0.219 | -1.10 | 1.41 | 1.44 | -1.11 | 1.44 | 1.29 |
| T | 0.281 | -1.29 | -2.28 | -3.29 | -2.75 | -2.49 | -1.38 |
| C | 0.219 | -1.85 | -2.97 | -2.67 | -3.04 | -3.63 | -1.74 |
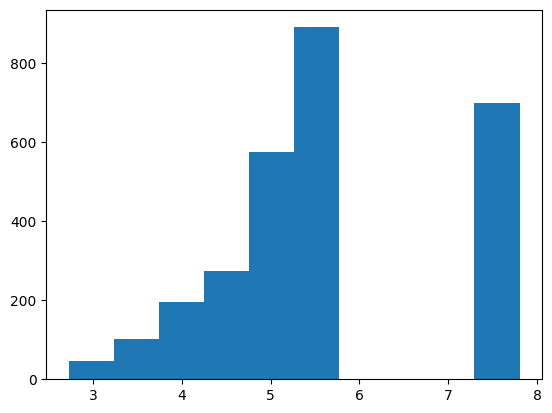
Далее были отобраны выборки, для которых предполагается наличие сигнала (участки перед генами) и отрицательный контроль - участки того же гена, но сдвинутые на 30 п.н. Были посчитаны веса для каждой последовательности из выборок по нашей PWM матрице, а также веса для обучающей выборки. Результат можно увидеть на Гистограммах 1-3 и Таблице 2:

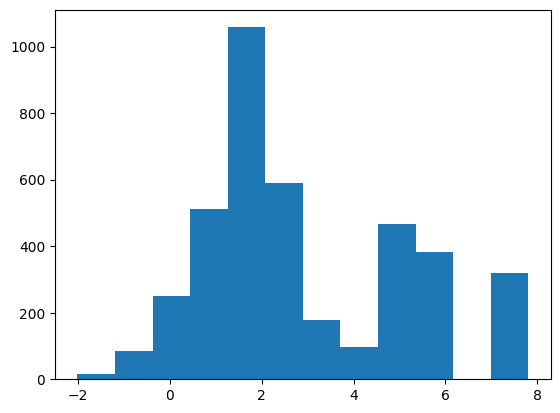
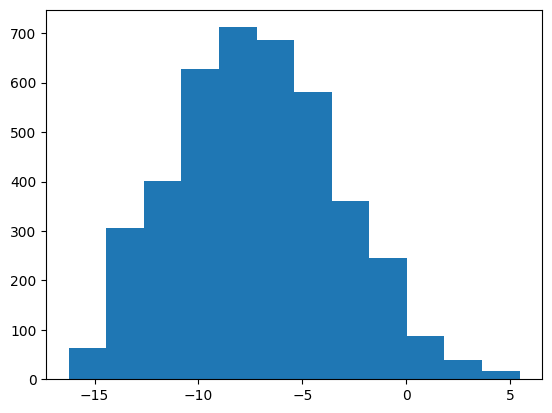
На основе приведенных гистограмм был выбран порог веса 0.5 и для него посчитана следующая таблица с результатами поиска сигналов в последовательностях из разных выборок:
Таблица 2. Результаты поиска в разных выборках последовательности Шайна-Дальгарно бактерии Bacillus Subtilis
| Обучающая выборка | Последовательности перед генами Bacillus Subtilis | Негативный контроль | |
| Сигнал (+) | 2781 | 3581 | 111 |
| Сигнал (-) | 0 | 375 | 4017 |
Таким образом, отработанный подход позволяет довольно эффективно отличать выборки с сигналами от выборок без сигнала. Вероятно он будет работать не только для последовательностей Шайна-Дальгарно разных бактерий, но и для других консервативных сигналов.
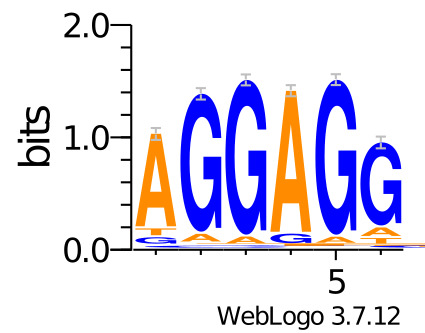
По обучающей выборке была построена матрица информационного содержания с помощью моего скрипта по ссылке ниже (сама матрица приведена в Таблице 3):
Скрипт для построения матрицы информационного содержанияТаблица 3. Матрица информационного содержания для последовательности Шайна-Дальгарно бактерии Bacillus Subtilis
| Баз. частоты | 1 | 2 | 3 | 4 | 5 | 6 | |
| A | 0.281 | 1.25 | -0.14 | -0.13 | 1.51 | -0.12 | -0.15 |
| G | 0.219 | -0.12 | 1.83 | 1.92 | -0.12 | 1.92 | 1.47 |
| T | 0.281 | -0.14 | -0.09 | -0.05 | -0.07 | -0.08 | -0.14 |
| C | 0.219 | -0.09 | -0.05 | -0.06 | -0.05 | -0.03 | -0.10 |
А затем LOGO был построен с помощью сервиса WebLOGO 3:
Число сайтов GAATTC было вычислено в полном геноме E.coli O157:H7 Sakai с помощью моего скрипта, доступного по ссылке ниже:
Скрипт для подсчета сайтовЧисло сайтов оказалось равным 801, что явно ниже ожидаемого по случайным причинам числа сайтов 1245. Если считать число сайтов в геноме распределенным по Пуассону, то вероятность получить такое число сайтов или меньше по случайным причинам составляет 1,55*10^-41 (посчитано в WolframAlpha). Наблюдаемое явления можно объяснить тем, что данный сайт является сайтом рестрикции EcoRI - рестриктазы, которая экспрессируется в E.coli для защиты от бактрериофагов. По видимому, несмотря на защиту этих сайтов в геноме бактерии метилированием (рестриктаза не режет метилированную ДНК), данные сайты все равно находятся под давлением отбора.