-
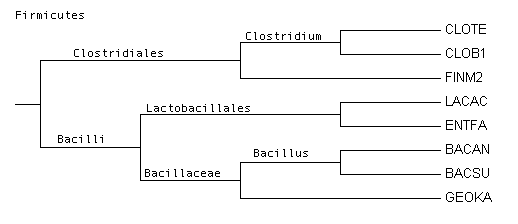
1. Таксоны выбранных бактерий.
| Таксоны | Мнемоника |
| Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group; Bacillus anthracis | BACAN |
| Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis group; Bacillus subtilis | BACSU |
| Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium; Clostridium botulinum | CLOB1 |
| Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium; Clostridium tetani | CLOTE |
| Bacteria; Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus; Enterococcus faecalis | ENTFA |
| Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiales incertae sedis; Clostridiales Family XI. Incertae Sedis; Finegoldia; Finegoldia magna | FINM2 |
| Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus; Geobacillus kaustophilus | GEOKA |
| Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus; Lactobacillus acidophilus | LACAC |
-
2. Функция: рибосомный белок S2.
Пример команды при получении последовательностей из банка Swiss-Prot: seqret sw:RS2_GEOKA -outseq RS2_GEOKA.fasta
-
5. Реконструкция дерева программой fprotpars.
(((CLOTE/1-23,CLOB1/1-23),(GEOKA/1-23,(((LACAC/1-25,FINM2/1-26),ENTFA/1-26),BACSU/1-24))),BACAN/1-23)
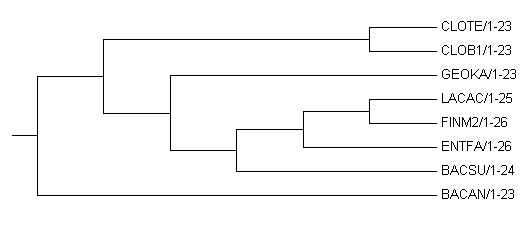
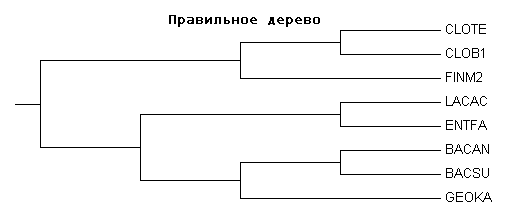
Есть большие отличия от правильного дерева.Например, BACAN и BACSU неправдаподобно далеко находятся друг от друга, FINM2 не в своей кладе.
Нет ветви {CLOTE,CLOB1,FINM2}vs{LACAC,ENTFA,BACAN,BACSU,GEOKA}. Вместо нее {CLOTE,CLOB1,BACAN}vs{LACAC,ENTFA,BACSU,GEOKA,FINM2}.
Ветвь {LACAC,ENTFA,FINM2}vs{CLOTE,CLOB1,BACAN,BACSU,GEOKA} вместо {LACAC,ENTFA}vs{CLOTE,CLOB1,FINM2,BACAN,BACSU,GEOKA}
-
6. Эволюционные расстояния между последовательностями программой fprotdist.
BACAN BACSU CLOB1 CLOTE ENTFA FINM2 GEOKA LACAC
BACAN/1-23 0.000000 0.129099 0.368206 0.352468 0.295251 0.397747 0.169656 0.438014
BACSU/1-24 0.129099 0.000000 0.371016 0.384315 0.346445 0.491174 0.122655 0.463836
CLOB1/1-23 0.368206 0.371016 0.000000 0.134211 0.397702 0.382930 0.374811 0.466398
CLOTE/1-23 0.352468 0.384315 0.134211 0.000000 0.422162 0.387458 0.340334 0.478190
ENTFA/1-26 0.295251 0.346445 0.397702 0.422162 0.000000 0.551788 0.346016 0.446305
FINM2/1-26 0.397747 0.491174 0.382930 0.387458 0.551788 0.000000 0.401789 0.532973
GEOKA/1-23 0.169656 0.122655 0.374811 0.340334 0.346016 0.401789 0.000000 0.470001
LACAC/1-25 0.438014 0.463836 0.466398 0.478190 0.446305 0.532973 0.470001 0.000000
Аксиома ультраметричности (d (A,B) ≤ max(d (A,C), d (B,C))) во взятом случайно примере выполняется:
d(LACAC,CLOTE) ≤ max( d(LACAC,CLOB1), d(CLOTE,CLOB1) ) <=> 0.478190 ≤ max(0.466398, 0.134211)(с погрешностью 2,5 процента, т.е. левая часть практически рана максмуму из правой)
Аддитивность (если есть четыре последовательности A,B,C,D, то из трёх сумм 1) d(A,B) + d(C,D) 2) d(A,C) + d(B,D) 3) d(A,D) + d(B,C) две равны между собой и больше третьей):
1) d(CLOTE, CLOB1) + d(LACAC, ENTFA) = 0,134211 + 0,446305 = 0,580516
2) d(CLOTE, LACAC) + d(CLOB1, ENTFA) = 0,478190 + 0,397702 = 0,875892
3) d(CLOTE, ENTFA) + d(LACAC, CLOB1) = 0,422162 + 0,466398 = 0,888560
Аддитивность для данного примера с учетом погрешности(1,5%) выполняется.
-
7. Две реконструкции дерева программой fneighbor, используя два алгоритма: UPGMA и Neighbor-Joining.
Neighbor-Joining:
+---BACSU/1-24 +-3 ! +--GEOKA/1-23 ! ! +----------ENTFA/1-26 ! ! ! ! +---CLOB1/1-23 4---5 +----1 ! ! +--2 +---CLOTE/1-23 ! ! ! ! ! +-6 +-------------FINM2/1-26 ! ! ! +--------------LACAC/1-25 ! +--BACAN/1-23
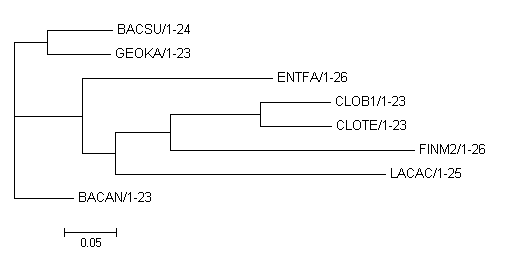

присутствует ветвь {CLOTE,CLOB1,FINM2,LACAC}vs{BACAN,BACSU,GEOKA,ENTFA} вместо {LACAC,ENTFA}vs{CLOTE,CLOB1,FINM2,BACAN,BACSU,GEOKA}
{BACSU,GEOKA}vs{BACAN,CLOTE,CLOB1,FINM2,LACAC,ENTFA} вместо {BACAN,BACSU}vs{GEOKA,CLOTE,LACAC,ENTFA,CLOB1,FINM2}
UPGMA:
+---BACAN/1-23 +-----3 ! ! +---BACSU/1-24 +-4 +-1 ! ! +---GEOKA/1-23 ! ! +-5 +---------ENTFA/1-26 ! ! ! ! +---CLOB1/1-23 +-6 +------2 ! ! +---CLOTE/1-23 --7 ! ! +------------FINM2/1-26 ! +-------------LACAC/1-25
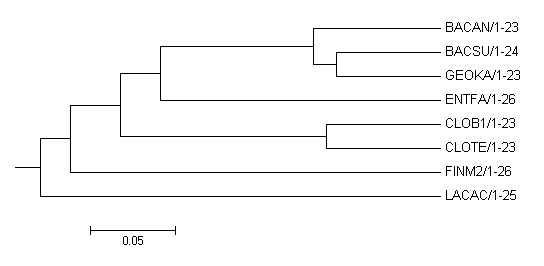

{BACAN,BACSU,GEOKA,ENTFA}vs{CLOTE,CLOB1,FINM2,LACAC} вместо {LACAC,ENTFA,BACAN,BACSU,GEOKA}vs{CLOTE,CLOB1,FINM2}(LACAC не в своей ветви)
{BACSU,GEOKA}vs{BACAN,CLOTE,CLOB1,FINM2,LACAC,ENTFA} вместо {BACAN,BACSU}vs{GEOKA,CLOTE,LACAC,ENTFA,CLOB1,FINM2}
Оба дерева построенные с помощью fneighbor отличаются от правильного, но построенное по алгоритму UPGMA ближе к правильному(отличия приведены выше).