-
1. Укоренение в среднюю точку.
Укоренение, я считаю, произошло правильно, т.к. делит дерево на правильные ветви ({CLOTE,CLOB1,FINM}vs{BACAN,BACSU,ENTFA,LACAC,GEOKA}).
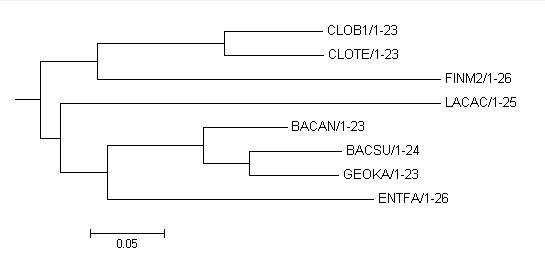
Можно сравнить с правильным:
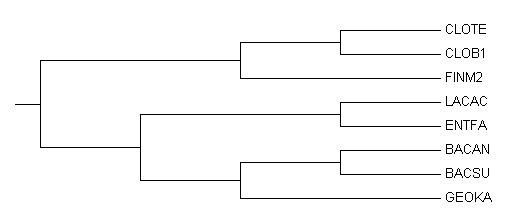
-
2. Использование внешней группы.
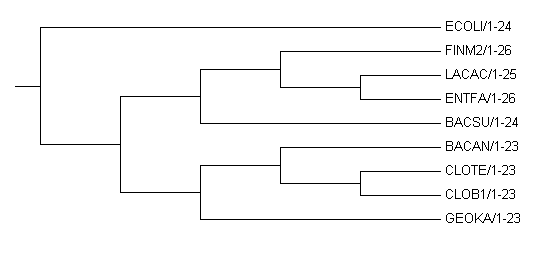
Дерево получилось не похожим на правильное, но здесь проблема не в укоренении, а в том, что построенное программой fprotpars дерево уже сильно отличалось от правильного(например, ветвь {BACAN,CLOB1,CLOTE,GEOKA}vs{FINM,ENTFA,LACAC,BACSU}).
-
3. Бутстрэп.
+---------------BACSU +--72.5-| | | +-------ENTFA +--44.0-| +--77.5-| | | +-------FINM2 +--81.0-| | | | +-----------------------GEOKA | | +-------| +-------------------------------BACAN | | | | +-------CLOB1 | +-------------------------100.0-| | +-------CLOTE | +-----------------------------------------------LACAC Species in order: 1. LACAC 2. CLOTE 3. CLOB1 4. GEOKA 5. FINM2 6. ENTFA 7. BACSU 8. BACAN Правильные ветви, которые не были включены: ...*..** 8.00 ...*.*** 5.00 Некоторых правильных ветвей совсем нет среди вариантов.
Не могу сказать, что улучшилась реконструкция филогении по сравнению с результатом fprotpars на исходном выравнивании, но изменилась, хотя все равно далека от правильной(например, присутствует ветвь {CLOB1,CLOTE,LACAC}vs{FINM2,BACAN,BACSU,ENTFA,GEOKA}(FINM2 не в своей ветви)). Возможно, выбранный белок слишком консервативен, чтобы строить филогенетические деревья для близких таксонов.