Работа с UniProt
1. Введение
Для анализа мне был предложен белок DNA integrity scanning protein DisA, выделенный из организма Thermotoga maritima. Данный белок находит поврежденные участки ДНК (создает глобулярный фокус, который быстро сканирует хромосому, останавливаясь на поврежденных участках ДНК).DisA откладывает начало споруляции до тех пор, пока не произойдет репарация (N.B гомологи DisA также встречаются у неспорулирующих бактерий).1
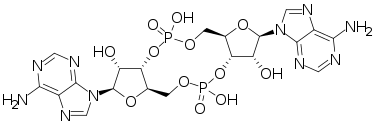
В структуре белка присутствует циклический диаденозинмонофосфат. Белок синтезирует циклический диаденозинмонофосфат (cyclic-di-AMP или с-di-AMP), который является вторичным посредником у бактерий и архей.2 Было показано, что DisA катализирует синтез с-di-AMP из 2 молекул ATP.3,4
Информация об организме
Thermotoga maritima(Organism ID: 243274)-это анаэробная,гипертермофильная, грамотрицательная,неспорулирующая эубактерия палочковидной формы. Была изолирована из геотермально нагретого морского дна. Обитает в температурных диапозонах от 55 до 90°С, оптимальное значение составляет около 80°С.5
Данный апплет показывает структуру белка Disa в комплексе с dATP и Mn2+,
а также шаростержневую модель с-di-AMP
2. Таблица с информацией о белке
Для того, чтобы найти этот белок в базе UniProt, была использована форма Retrieve/ID mapping(from EMBL/GenBank/DDBJ CDS to UniProtKB). Основная информация о белке, полученная из базы данных UniProt, предcтавлена в таблице 1.
| Таблица 1. Информация о белке | |||
|---|---|---|---|
| Раздел UniProtKB | Swiss-Prot | ||
| UniProt ID | DISA_THEMA | ||
| UniProt AC | Q9WY43 | ||
| EMBL AC | AE000512; AAD35292.1 | ||
| PDB ID | 3C1Y;
3C1Z;
3C21;
3C23; 4YVZ; 4YXJ; 4YXM |
||
| Длина | 357 AA | ||
| Молекулярная масса | 40540 Da | ||
| Рекомендуемое название (RecName) |
DNA integrity scanning protein DisA | ||
| Sequence status | Complete | ||
3. Результаты поисковых запросов в UniProt
В таблице 2 содержится информация, полученная с использованием расширенного поиска в UniProt.
| Таблица 2. Сеансы поиска в UniProt | |||
|---|---|---|---|
| Поисковый запрос | Результат поиска | ||
| (1)name:"dna integrity scanning protein disa" (2)name:"dna integrity scanning protein disa" taxonomy:bacteria (3)name:"dna integrity scanning protein disa" taxonomy:thermotogae (4)name:"dna integrity scanning protein disa" taxonomy:archaea |
Списки белков с такой же функцией среди всех организмов(1)-5327 результатов (среди них 65 Swiss-Prot) среди бактерий(2)-5242 результата (65 Swiss-Prot) внутри типа Термотоги(3)-67 результатов (3 Swiss-Prot) среди архей(4)-61 результат, все TrEMBL |
||
| cluster:(uniprot:q9wy43* identity:0.9) NOT id:Q9WY43 | Список белков с похожими аминокислотными последовательностями, порог идентичности 90% 13 результатов |
||
| cluster:(uniprot:q9wy43* identity:0.5) NOT id:Q9WY43 | Список белков с похожими аминокислотными последовательностями, порог идентичности 50%. Белки: DNA integrity scanning protein DisA и DAC domain-containing protein 28 результатов |
||
4. Изучение кластеров UniRef.
В кластерах UniRef50, UniRef100 и UniRef90 мой белок является репрезентативной последовательностью, но не является сидом. Это значит, что мой белок является наиболее изученным из белков данных кластеров, но его последовательность не самая длинная. Сид относится к базе UniParc, находится в кластере UniRef100 и также принадлежит бактерии Thermotoga maritima.
Как видно из таблицы 3, кластеры данного белка содержат сравнительно небольшое число белков, что говорит о низкой распространенности подобных белков среди бактерий. В кластере UniRef50 присутствуют белки, выделенные из организмов порядка Thermotogales, большинство организмов относятся к роду Thermotoga. Из этого можно сделать вывод, что белок является достаточно специфичным.
| Таблица 3. Кластеры UniRef белка DisA | |||
|---|---|---|---|
| UniRef Cluster | Количество белков из базы UniProtKB | Количество белков из базы UniParc | |
| UniRef50_Q9WY43 | 29, 3 reviewed | 11 | |
| UniRef90_Q9WY43 | 14, 3 reviewed | 3 | |
| UniRef100_Q9WY43 | 1, 1 reviewed | 1 | |
5. Сравнение протеомов.
В таблице 4 представлено сравнение протеомов двух близкородственных бактерий: Thermotoga maritima и Pseudothermotoga lettingae. Бактерии относятся к одному семейству Thermotogaceae.
Как видно из таблицы, у бактерии Pseudothermotoga lettingae протеом содержит большее количество белков, но при этом аннотированных последовательностей значительно меньше. У этой бактерии также число трансмембранных белков больше, чем у бактерии Thermotoga maritima, но аннотированных последовательностей совсем мало, что говорит о том, что трансмембранные белки данной бактерии не представляют особого интереса. У моей бактерии также аннотировано небольшое число трансмембранных белков.
В протеом бактерии Thermotoga maritima входит большее число ферментов, и большинство из них проверены, в тот время как у второго протеома аннотировано только 34% белковых последовательностей.
В качестве третьей функциональной группы были выбраны белки, устойчивые при высоких температурах и/или обладающих ферментативной активностью при высоких температурах.
Подобные белки интересны в контексте изучения моей бактерии, так как она обитает при высоких температурах.
Команда entret -filter sw:*_THEMA stdout|grep -A 1 'Temperature dependence' &> tmaritima.txt записывает в файл строчки из записей Swiss-Prot, содержащих информацию о теипературных особенностях функционирования белков бактерии. Из файла видно, что большая часть белков активна при 80, 90, 60-70°С, что соответствует температурному диапозону жизни Thermotoga maritima.
В базе данных UniProt не нашлось записей о термоустойчивых белках протеома бактерии Pseudothermotoga lettingae, несмотря на то, что данная бактерия также является гипертермофильной.6
Небольшое количество аннотированных последовательностей в протеоме Pseudothermotoga lettingae можно объяснить тем, что бактерия была открыта намного позже моей бактерии6, и она в большей степени интересна с точки зрения эволюционных исследований, филогенетических и геномных анализов.
6. Список литературы
- Bejerano-Sagie, M., Oppenheimer-Shaanan, Y., Berlatzky, I., Rouvinski, A., Meyerovich, M. and Ben-Yehuda, S. (2006) A checkpoint protein that scans the chromosome for damage at the start of sporulation in Bacillus subtilis. Cell 125, 679–690
- Braun, F; Thomalla, L; van der Does, C; Quax, TEF; Allers, T; Kaever, V; Albers, SV (September 2019). "Cyclic nucleotides in archaea: Cyclic di-AMP in the archaeon Haloferax volcanii and its putative role". MicrobiologyOpen. 8 (9): e00829. doi:10.1002/mbo3.829
- Witte, G., Hartung, S., Buttner, K. and Hopfner, K.P. (2008) Structural biochemistry of a bacterial checkpoint protein reveals diadenylate cyclase activity regulated by DNA recombination intermediates. Mol. Cell 30, 167–178
- Müller, M., Deimling, T., Hopfner, K.-P., & Witte, G. (2015). Structural analysis of the diadenylate cyclase reaction of DNA-integrity scanning protein A (DisA) and its inhibition by 3′-dATP. Biochemical Journal, 469(3), 367–374. doi:10.1042/bj20150373
- Huber, R., Langworthy, T. A., König, H., Thomm, M., Woese, C. R., Sleytr, U. B., & Stetter, K. O. (1986). Thermotoga maritima sp. nov. represents a new genus of unique extremely thermophilic eubacteria growing up to 90°C. Archives of Microbiology, 144(4), 324–333. doi:10.1007/bf00409880
- Balk M, Weijma J, Stams AJM. Thermotoga lettingae sp. nov., a novel thermophilic, methanol-degrading bacterium isolated from a thermophilic anaerobic reactor. Int J Syst Evol Microbiol. 2002 Jul;52(Pt 4):1361-1368. doi: 10.1099/00207713-52-4-1361. PMID: 12148651.