Практикум 15. Сборка de novo
1. В качестве материала для сборки мне были предложены одиночные риды из проекта SRR4240359, получившиеся в результате секвенирования бактерии Buchnera aphidicola. Первым шагом был скачан файл с чтениями при помощи команды wget.
Затем было произведено удаление остатков адаптеров и триммирование чтений программой trimmomatic. Адаптеры были собраны в файле afapters.fasta. Были удалены правоконцевые нуклеотиды с качеством ниже 20, чтения с длиной меньше 32 нуклеотидов были отсеяны. Команда имела следующий вид:
➙ java -jar /usr/share/java/trimmomatic.jar SE -phred33 -trimlog log_trim SRR4240381.fastq.gz SRR4240381_trim.fastq.gz ILLUMINACLIP:adapters.fasta:2:7:7 TRAILING:20 MINLEN:32Для того, чтобы понять, какой процент остатков адаптеров содержится в чтениях, trimmomatic был запущен отдельно со "step"ом ILLUMINACLIP. Оказалось, что адарптеров в чтениях было всего 5508 (0.04%).
Изначальное количество ридов: 13710994. Было удалено 2486475 (18.13%) ридов, вес файла уменьшился с 533 461 KB до 414 692 KB.
2. Следующим шагом требовалось хэшировать полученные триммированные чтения программой velveth. Данная программа на основе этих чтений подготовила k-меры длинной 31 нуклеотид. Была создана директория velveth, которая затем использовалась как рабочая директория для программы velvetg.
➙ velveth velveth 31 -fastq.gz -short SRR4240381_trim.fastq.gz &> log_velvhПосле этого была произведена сборка генома на основе k-меров при помощи программы velvetg.
➙ velvetg velveth &> log_velvg
- Значение N50 для сборки: 6376;
- Самые длинные контиги: 69671, 27474 и 26062;
- Покрытия контигов: 33.072769, 38.784962 и 35.125393 соответственно.
Источники: stats.txt – информация о длинах контигов и покрытиях, contigs.fa – покрытия, лог-файл – N50.
3. Три самых длинных контига сравнили с хромосомой Buchnera aphidicola (AC CP009253) для анализа правильности сборки. Для этого была использована программа megablast. Значение wordsize по умолчанию 28, все остальные параметры также по умолчанию.
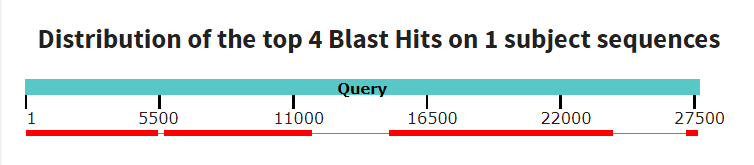
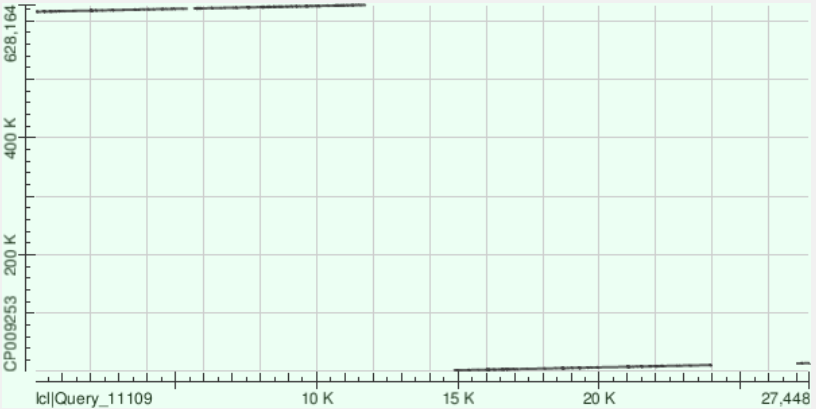
✶ Результат бласта первого (самого длинного) контига с хромосомой. Покрытие 78%, процент идентичности 77,03%, что является неплохим результатом при таком покрытии. Всего получилось 13 выравниваний. Контиг соответствует участку хромосомы от 462496 до 529211 позиции. Число гэпов (суммарно по всем выравниваниям) 1995, число однонуклеотидных замен 11178. На рис. 1 и 2 видно, что контиг хорошо ложится на геном, можно наблюдать 6 крупных делеций.
✶ Результат бласта второго по длине контига с хромосомой. Всего получилось 4 выравнивания. Покрытие 76%, процент идентичности 78,4%. Координаты участка хромосомы, на который ложится контиг: от 2004 до 627104 позиции. Число гэпов 1089, число однонуклеотидных замен 4154.
✶ Результат бласта третьего по длине контига с хромосомой. Получилось только одно выравнивание. Покрытие меньше, чем у предыдущих контигов – 27%, идентичность 76,59%.