Нуклеотидный BLAST
1. Определение функции и таксономии нуклеотидной последовательности
Консенсусная последовательность, полученная при расшифровке хроматограммы.
Для определения функции последовательности и таксономического положения организма был использован алгоритм blastn. Про последовательность ничего не известно, поэтому лучше использовать blastn вместо megablast, т.к. megablast может пропустить хорошие находки и не дает выставить параметр wordsize меньше 16.
Параметры blastn:
- Длина слова: 11 (при указании длины слова 7 blast не выдал результатов)
- Expect threshold: 0.05
- Чилсо находок: 100
- Параметры подсчета веса: по умолчанию
- Database: Nucleotide collection (nt)
Ссылка на выдачу
Лучшая находка – Polycirrus medusa voucher BIOUG
Вывод о таксономии
По результатам выдачи программы можго сделать вывод, что моя последовательность ближе всего к последовательностям из организма Polycirrus medusa (4 находки с самым высоким percent identity и весом). Большая часть находок также принадлежат организмам из рода Polycirrus (первые 20, а также некоторые другие находки), поэтому вероятнее всего консенсусная последовательность действительно была получена из организма Polycirrus medusa Прилагаю для наглядности проект Jаlview выравнивания первых 10 последовательностей, где четко видно, что все последовательности выровнлись хорошо, но первые две все же чуть лучше остальных, что логично, потому что они обе из одного места (kandalaksha bay, velikaya salma strait).
Вывод о функции
На основе выдачи можно сделать вывод, что исследуемая последовательность вероятнее всего представляет собой митохондриальный ген, кодирующий первый сабюнит цитохромоксидазы - фермента, который катализирует перенос электронов с цитохрома с на кислород в ходе электрон-транспортной цепи в митохондриях. Важный фермент, присутствующий у всех эукариот.
2. Поиск генов белков в неаннотированной нуклеотидной последовательности
Был взят контиг из сборки генома рыбы фугу Takifugu rubripes с длиной 64808 пар нуклеотидов. В качестве алгоритма поиска белков был выбран blastx, поиск производился по базе swissprot с целью получения проверенных находок.
Параметры blastx:
- Длина слова: 6
- Expect threshold: 0.05
- Чилсо находок: 100
- Параметры подсчета веса, матрица весов: по умолчанию
- Database: swissprot
ссылка на выдачу
Всего blast нашел 77 последовательностей. Большая часть находок, в том числе находок с лучшим весом и E value относится к различным изоморфам синаптотагмина – трансмембранного кальций-связывающего белка, участвующего в регуляции выброса нейромедиаторов (всего 17 изоморф, хотя связывать кальций способны только 8 из них). За связывания кальция отвечают два C2 домена.
В выдаче также присутствуют белки Double C2-like domain-containing protein alpha и Synaptotagmin-like proteins, что также отсылает нас к синаптотагминам, и белок рабфилин 3А, который также является кальций-связывающим и выполняет функцию регуляции экзоцитоза. Логично предположить, что белки со схожими функциями могут содержать гомологичные участкию Но все эти находки обладают достаточно высоким E value и сравнительно более низким процентом идентичности, чем синаптотагмин-9, поэтому я могу сделать вывод, что вероятнее всего контиг соджержит ген, кодирующий синаптотагмин-9.
3. Интепретация карты локального сходства гомологичных хромосом двух бактерий
В качестве объектов для выполнения данного задания были выбраны геномы 2 бактерий из рода стрептомицеты: Streptomyces venezuelae (AC NZ_CP029196.1) и Streptomyces olivaceus (AC NZ_CP016795.1). Карта локального сходства была построена при помощи алгоритма blast2seq, длина слова 28 и порог E value 9е-200 (такой низкий порог был использован для уменьшения шума).
По оси абсцисс показана последовательность S. venezuelae, по оси ординат – геном S. olivaceus.
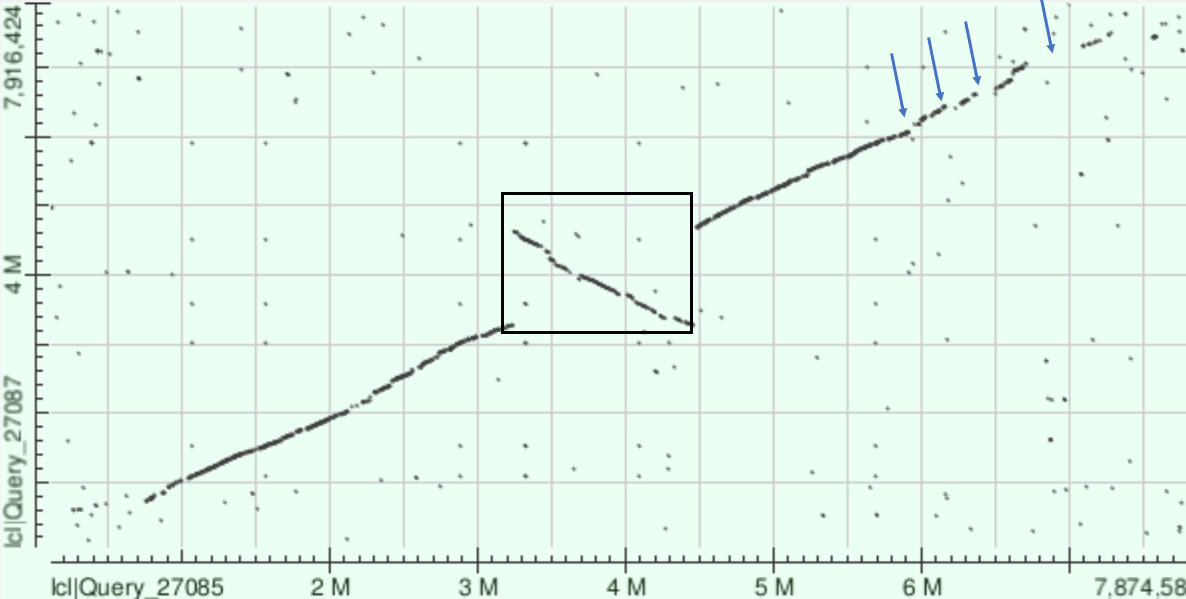
Ось ОХ - S. venezuelae, ось ОY - S. olivaceus
Четко видна инверсия в районе 3.3М - 4.5М (черный квадрат), а также многочисленные индели. Наиболее значительные индели наблюдаются в конце геномов, они отмечены синими стрелками.