База данных STRING
✶ База данных STRING позволяет найти различные взаимодействия (совместное присутствия, коэкспрессия и т.д.) между генами и белками и провести функциональное обогащение по разным параметрам (метаболические пути, внутриклеточная локализация и т.д). В качестве материала для анализа мне был предложен следующий список из 27 генов, поиск велся по организму Homo sapiens.
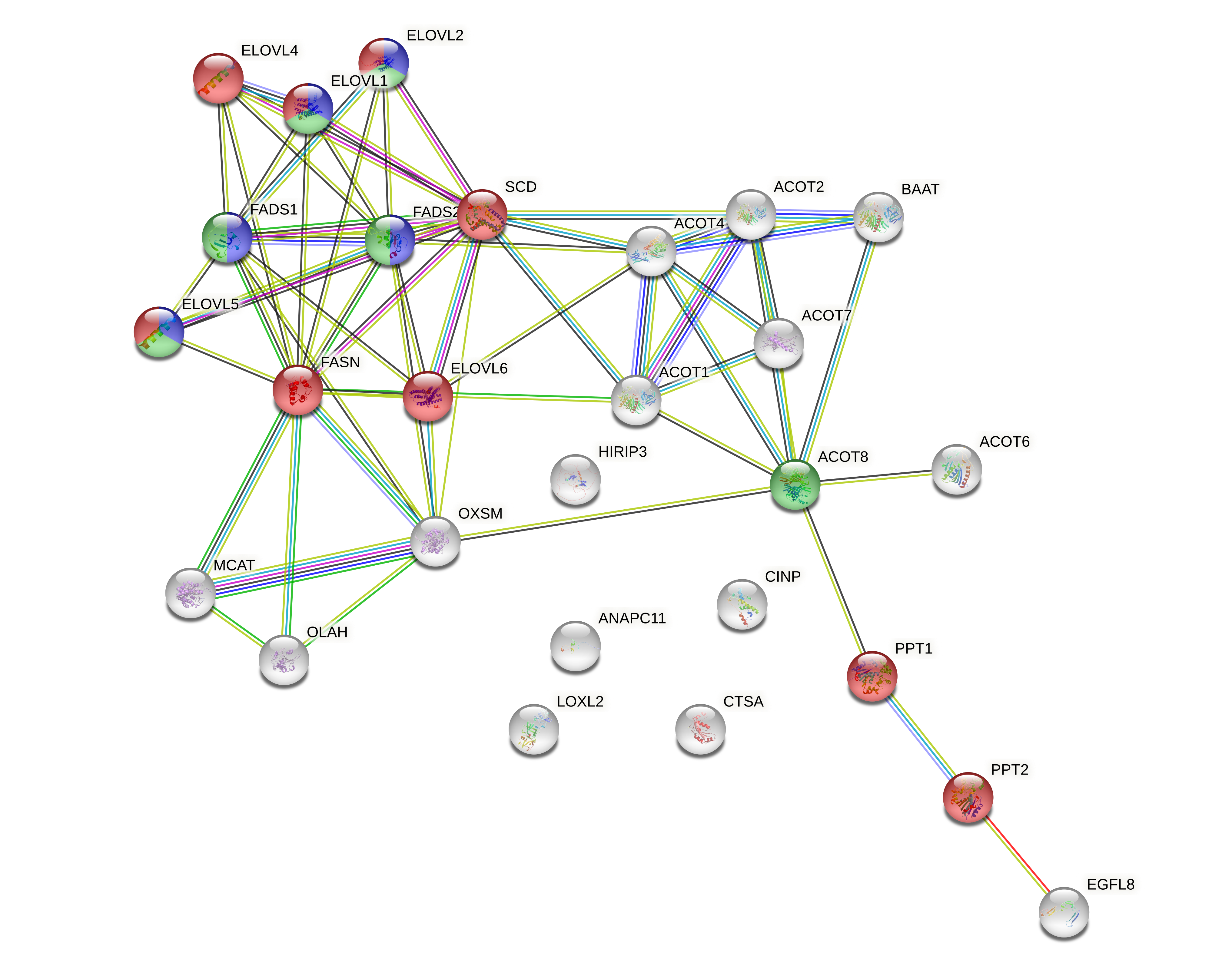
Схема всех взаимодейтсвий представлена на рис. 1. Данная схема иллюстрирует взаимодействия средней достоверности (т.е. взаимодействия, для которых оценка достоверности (confidence score) больше 0.4). 3D структура присутствует у всех 27 белков. Для 5 генов (LOXL2, CINP, CTSA, ANAPC11, HIRIP3) видим отсутствие каких-либо взаимодействий с другими генами.
✶ Цвета ребер отвечают за какой-то определенный тип взаимодействий. На рисунке 1 довольно много ребер желтого цвета, они показывают совместные упоминания генов в абстрактаз статей. Малиновые ребра символизируют экспериментально подтвержденные взаимодействия. Для данного набора генов можем наблюдать эксперементально подтвержденные взаимодействия между парами ACOT1 и ACOT2 (ссылка) и FASN и SCD (ссылка), для остальных взаимодейстивий есть экспериментальные подтверждения только у близких гомологов.
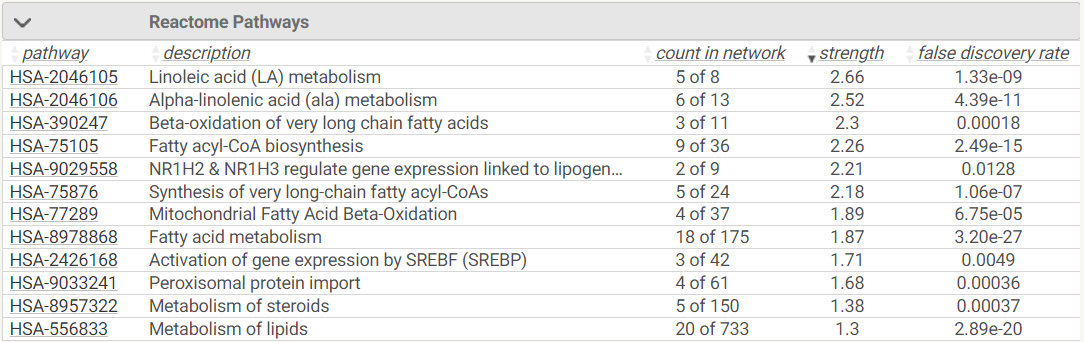
✶ В разделе Analysis можно посмотреть результаты функционального обогащения генов по разным базам данных. Давайте посмотрим на выдачу БД Reactome (рис. 2). Судя по данным из Reactome, белки, кодируемые генами, задействованы в метаболизме линолиевой и альфа-линолиевой кислот, синтезе ацил-Коа и бета-оксидации жирных кислот. В общем данные белки участвуют в метаболических путях, связанных с модификациями и синтезами жирных кислот и их производных. STRING позволяет также отобразить различные функциональные группы на схеме. На рисунке 2 красные узлы соответствуют белкам, участвующим в синтезе ацил-КоА, синие – белкам, участвующим в синтезе линолиевой кислоты, а зеленые – в синтезе альфа-линолиевой. По данным из Reactome, белки ELOVL1, ELOVL2, ELOVL5 учатсвуют во всех трех процессах.
✶ На рисунке 3 представлено совместное присутсвие генов у разных организмов. Наличие белка у организма, кодируемого каким-то геном, отмечается красным квадратом, а отсутствие - белым пробелом. Интенсивность цвета красного квадрата отражает степень консервативности гомологичного белка в данном организме[1]. На каждый квадратик можно нажать и посмореть выравнивание нашего белка с гомологичным ему у другого организма, и чем лучше выравнивание, тем интенсивнее цвет квадрата. Как мы видим, близжайшие гомологи белков из списка присутствуют у приматов, и впринципе их гомологи лучше всего представлены у млекопитающих. Стоит отметить, что у Gorilla gorilla отсутствует белок OLAH (тиоэстераза), а у Pongo abeli – ANAPC11 (Anaphase-promoting complex subunit 11). У гориллы также отсутствует тиоэстераза PPT2, вместо нее присутствует PPT1, которая не очень хорошо выравнивается по последовательности с PPT2 (ссылка на выравнивание).
Наибольшим числом гомологов обладает белок OXSM, являющийся митохондриальной 3-оксиацил-синтазой, относится к семейству бета-кетоацил-ACP синтаз. Данный митохондриальный белок имеет большое число гомологов среди бактерий. Его гомолог также присутствует у нескольких видов эуархеот.
Рис. 3. Совместное присутствие генов (наведите курсор на картинку чтобы увеличить)
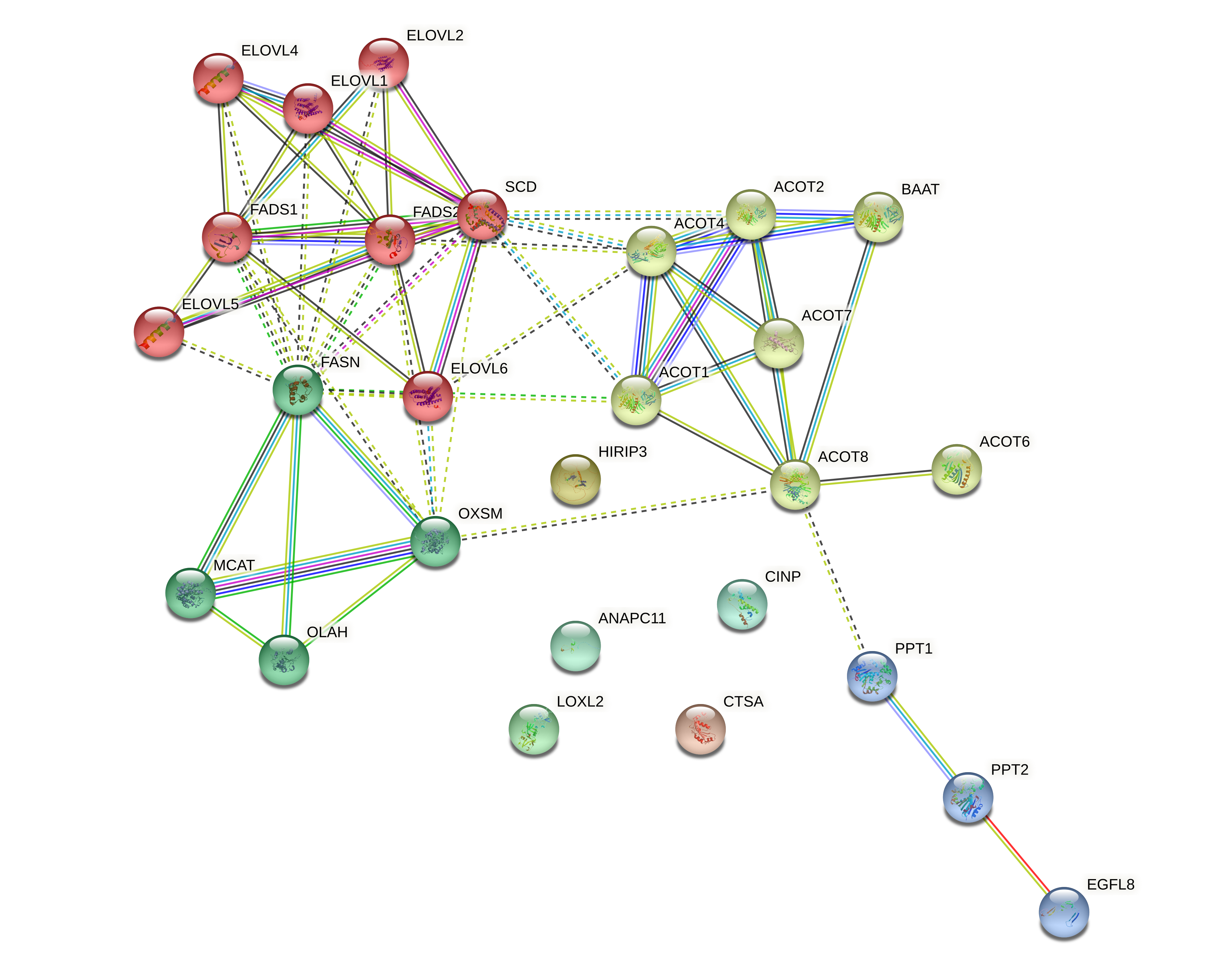
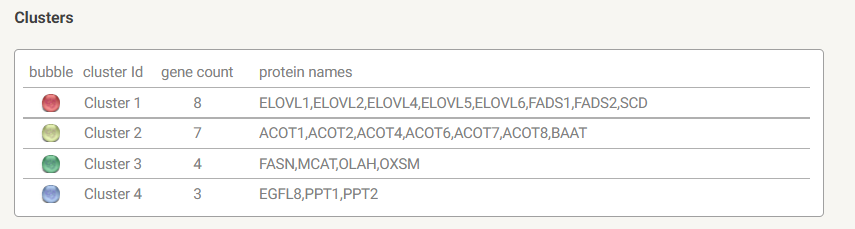
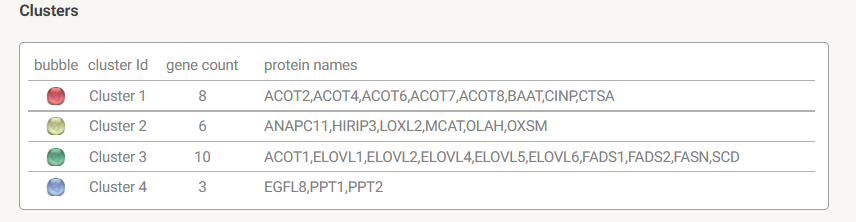
✶ Далее была проведена кластеризация сета белков. Поскольку я заведомо не знаю, на сколько групп можно поделить сет белков, я сначала провела кластеризацию методом MCL (Markov Cluster Algorithm). На рисунке 4 представлены результаты кластеризации. Параметр инфляции был выбран равным 3, потому что при увеличении данного параметра количество кластеров не увеличивалось, и я решила, что данное значение является наиболее оптимальным. Всего получилось 4 кластера, они представлены на рисунке разными цветами. Пунктирными линиями отмечены взаимодействия между кластерами.
Следующим шагом я решила провести кластеризацию методом k-средних чтобы посмотреть, будет ли отличаться результат кластеризации при использовании другого метода. Как мы видим, результаты имеют небольшие отличия. Например, метод k-средних вкдючил ACOT1 в кластер с белками ELOVL (это нельзя назвать грубым расхождением, поскольку по результатам MCL между белком и тем кластером есть взаимодействие). В отличие от MCL, в кластере k-means белки CINP и CTSA принадлежат к кластеру с ACOT, а ANAPC11, HIRIP3, LOXL2 в кластере с MCAT.
Рис. 4. Кластеризация генов. Слева MCL, справа метод k-средних.