Про трансмембранные белки
1. Описание белка из базы данных OPM
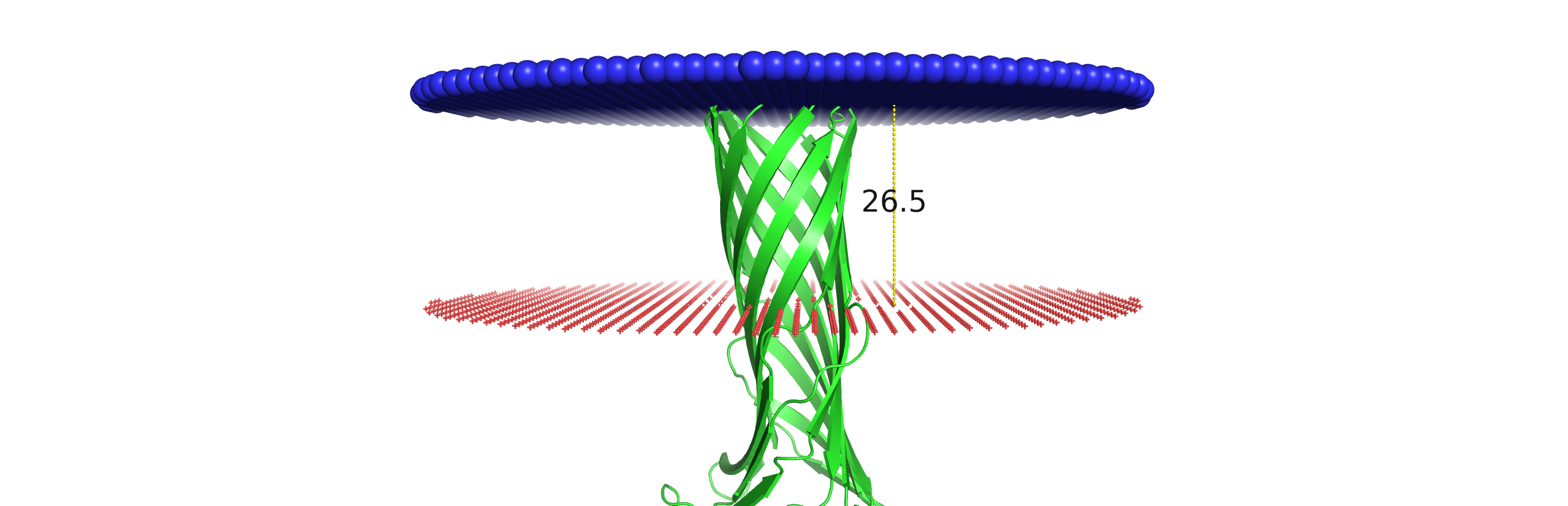
В качестве объекта для выполнения данного задания была выбрана протеаза OmpT E. coli, локализованная во внешней мембране бактерии. Сылка на БД OPM. PDB ID: 1i78, Uniprot: OMPT_ECOLI.
| Таблица 1. Информация о белке из базы OPM | |
|---|---|
| Название белка | Outer membrane protease OmpT |
| Класс белка | Beta-barrel transmembrane |
| Семейство | OM protease omptin, OMPT |
| Организм | Escherichia coli |
| Локализация | Внешняя мембрана грамотрицательных бактерий |
| Толщина гидрофобной части | 26.5 Å |
| Координаты трансмембранных участков | 1(11-20),2(50-61),3(64-72),4(111-121),5(126-136),6(178-187),7(192-201),8(230-240),9(245-254),10(287-296) |
| Среднее количество остатков в β-тяже | 9 |
2. DeepTMHMM: Предсказание трансмембранных элементов по последовательности белка
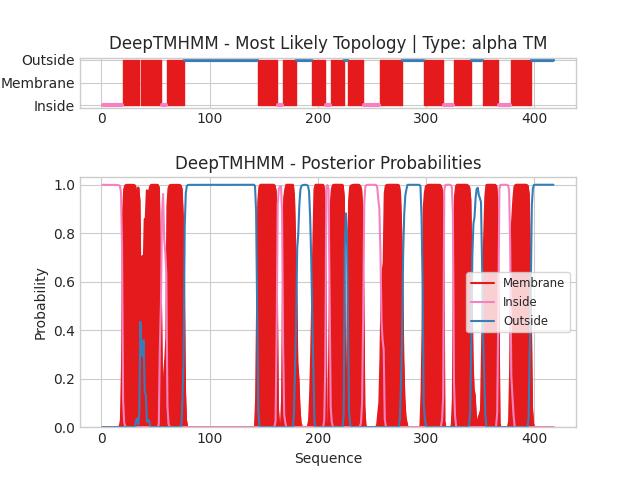
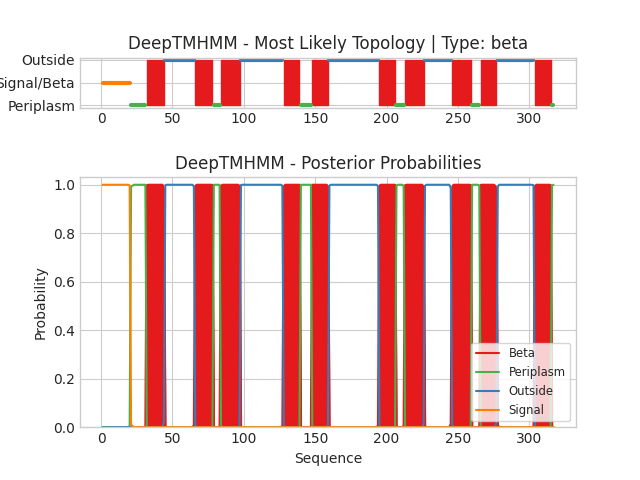
Было произведено предсказание трансмембранных участков двух белков: бета-бочонка из первого задания и альфа-спирального белка EPS I polysaccharide export inner membrane protein EpsF (EPSF_RALSL) при помощи сервиса DeepTMHMM. Данный белок предположительно участвует в экспорте через внутреннюю мембрану экзополисахаридов EPS I. На вход программе подается последовательность белка в fasta формате. Получили следующие результаты: альфа-спиральный белок и бета-бочонок. Графическая визуализация результатов представлена на рис. 2 и 3. По оси ОX представлены позиции аминокислот в последовательности, по оси OY – вероятность принадлежности определенного остатка к какой-то топологии.
У альфа-спирального белка N-конец находится внутри клетки (розовый цвет), С-конец снаружи (синий). Всего программа нашла 13 трансмембранных альфа-спиральных участков (показаны красным цветом).
У моего бета-листового белка наблюдается 10 трансмембранных участков (как и должно быть), на N-коне расположен сигнальный пептид (желтый). Имеются участке в периплазме – мемжмембранном пространстве у грамотрицательных бактерий.
3. Предсказание положения белка в мембране с помощью PPM
Входной файл, взят с сайта Uniprot: EPSF_RALSL. Результат работы PPM: pdb-структура
и html страница с результатами.
Параметры:
- Тип мембраны: внутренняя мембрана грамотрицательных бактерий (локализация следует из названия белка)
- Положение N-конца: внутри (из предсказания DeepTMHMM)
- Разрешить искривлять белок: нет (поскольку белок бактериальный деформация мембраны не предусматривается)
- Последний параметр (включать гетероатомы, исключать воду и детергенты) нам не принципиален, потому что на вход подается структура, полученная не из рентгенструктурного анализа, и в PDB файле нет полей HETATM.
| Таблица 2. Предсказания о расположении альфа-спирального белка | |
|---|---|
| Локализация | Внутренняя мембрана грамотрицательных бактерий |
| Толщина гидрофобной части | 29.9 ± 0.7 Å |
| Координаты трансмембранных участков | 1(9-31), 2(34-52), 3(56-74), 4(144-163), 5(167-188), 6(189-206), 7(211-226), 8(227-240), 9(259-282), 10(297-317), 11(323-345), 12(346-366), 13(377-397) |
| Среднее количество остатков в α-спирали | 19-20 |
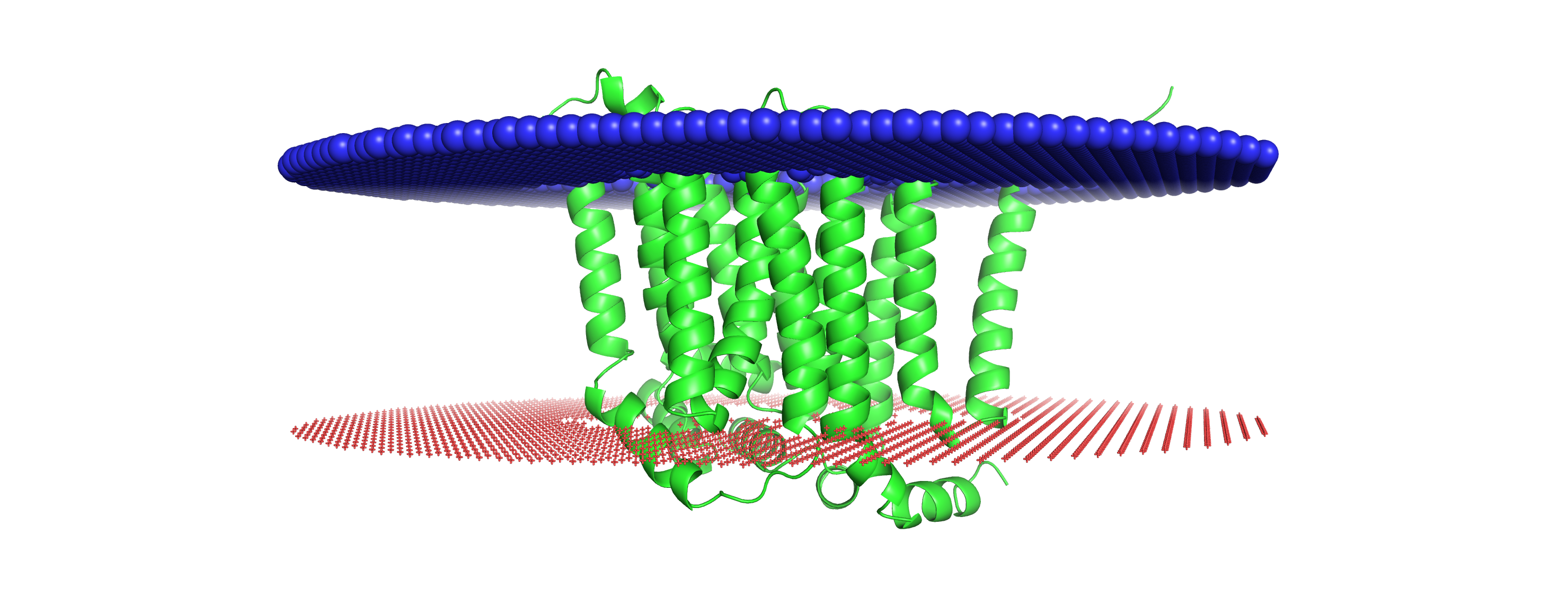
4. Сравнение алгоритмов предсказания трансмембранных спиралей
✶ Для выданного альфа-спирального белка EPSF_RALS сервисы DeepTMHMM и PPM предсказали одинаковое количество элементов (13). Сравним координаты трансмембранных альфа-спиралей:
DeepTMHMM: 1(20-35), 2(37-55), 3(61-76), 4(145-162), 5(168-180), 6(195-207), 7(212-224), 8(228-242), 9(258-278), 10(298-316), 11(326-342), 12(353-367), 13(379-397)
PPM: 1(9-31), 2(34-52), 3(56-74), 4(144-163), 5(167-188), 6(189-206), 7(211-226), 8(227-240), 9(259-282), 10(297-317), 11(323-345), 12(346-366), 13(377-397)
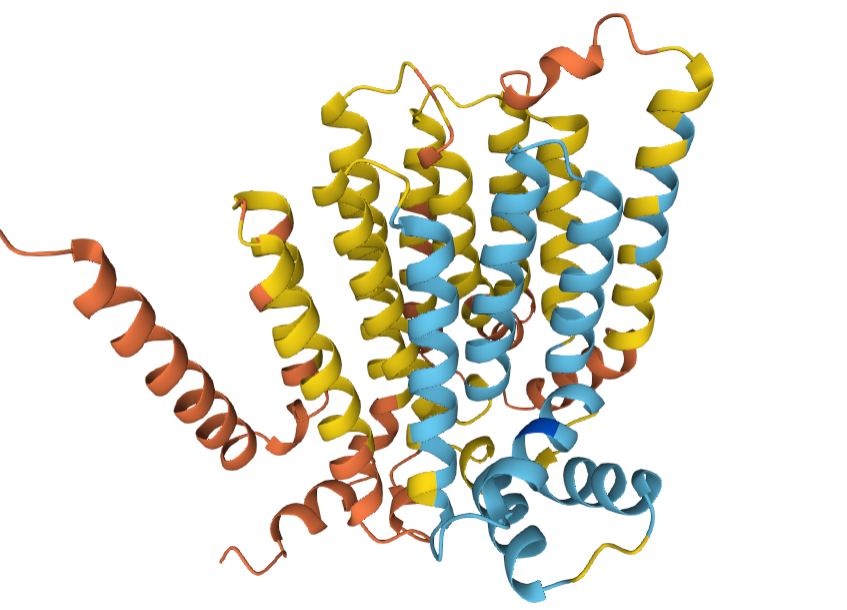
Видим расхождения в координатах, особенно для первого элемента. Полных совпадений не наблюдается, однако участки находятся примерно в одних регионах. На рисунке представлена 3D структура белка, предсказанная программой AlphaFold. Большая часть белка предсказана недостоверно. Альфа-спираль на N конце недостоверна полностью судя по pLDDt-скору, низкую достоверность также имеет C-концевой участок. Низкая достоврность элементов вторичной структуры указывает на то, что вероятность того, что какие-то остатки формируют именно этот элемент вторичной структуры низка. Это значит, что качество предсказания расположения белка в мембране, выполненное на основе вторичной структуры, должно напрямую зависить от качества самой изначальной пространственной структуры, ведь для достоверного предсказания важно иметь правильно расположенные аминокислотные остатки внутри мембраны.
✶ Для бета-бочонка DeepTMHMM предсказал правильное число бета-листов (10)
DeepTMHMM: 1(32-44), 2(66-78), 3(84-97), 4(128-139), 5(148-159), 6(195-206), 7(213-226), 8(246-259), 9(266-277), 10(304-315)
OPM: 1(11-20),2(50-61),3(64-72),4(111-121),5(126-136),6(178-187),7(192-201),8(230-240),9(245-254),10(287-296)
Здесь опять видны расхождения, даже более существенные, чем в случае с альфа-спиральным белком.
5. База данных TCDB
Поиск по Uniprot AC альфа-спирального белка в базе данных TCDB не дал никаких резултатов, страница есть только для бета-бочонка (ссылка на выдачу). По классификации TC (transport classification) белок имеет код 9.B.50.1.1, здесь 9 – это предполагаемык транспортные белки, 9.B.50 – бета-листовые эндопротеазы внешней мембраны, семейство Омптин.
Мне удалось найти альфа-спиральный белок, похожий на выданный. В поиске TCDB я вбила название бактерии, и среди предложенных результатов была ссылка на белок EPS I polysaccharide export inner membrane protein EpsE. Судя по записи в Uniprot, предполагаемые функции у этих двух белков одинаковые. Его вторичная структурв также была предсказана с помощью AlphaFold, и это предсказание намного более достоверное. По классификации ТС данный белок имеет следующий код: 2.A.66.2.11, где 2 – это электрохимические потенциал-зависимые транспортеры, 2.A – портеры, 2.A.66.2 – семейство транспортеров полисахаридов.