Выравнивание геномов
Для выравнивания 2 последовательностей был выбраны сборки геномов рода Bacillus, а именно Bacillus smithii (AC: NZ_CP012024.1) и Bacillus rugosus (AC: NZ_CP096590.1). У обоих сборка complete genome, референсные.
Геномы искались при помощи "Browse by Organism" в NCBI с параметрами: сборка хромосомная и выше, референсный геном.
Было проведено 2 выравнивания: blastn и megablast. Результаты выравнивания можно увидеть на DotPlot (рис. 1 и рис. 2):
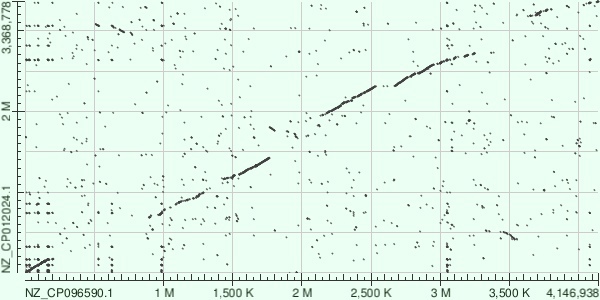
Рисунок 1. DotPlot blastn
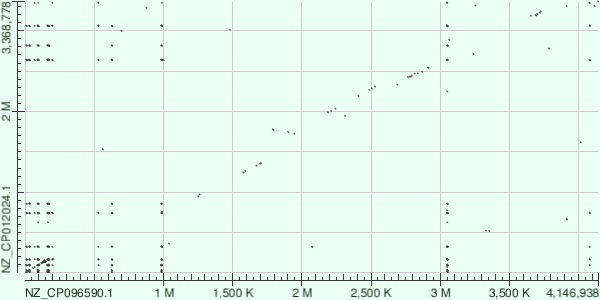
Рисунок 2. DotPlot megablast
Можно заметить, что в целом blastn-ом они выровнялись лучше (что логично, например, из-за различия в параметре wordsize). В целом на обоих дотплотах хорошо заметна общая линия выравнивания. Обе последовательности берут начало в нулевых координатах, что говорит о правильном выборе цепи ДНК и точки начала кольцевой хромосомы бактерии.
На некоторых участках произошла инверсия (по оси абсцисс: 0,5 - 0,75 М, несколько небольших последовательностей; 1,75 - 1,8; 3,45 - 3,55). Также виден участок делеции или инсерции (2,55 - 2,70). Большое количество просто негомологичных, вариабельных участков (например, самый большой из них: 0,2 - 0,95, а также 1,3- 1,4; 1,8 - 2,15; 3,3 - 3,4; 3,6 - 3,7). Можно заметить, особенно на выравнивании blastn, очень много случайных совпадений, которые выражаются в точках, расположенных в рандомных координатах. Однако есть участок 0 - 0,2 (а также очень маленькие на 0,6; 1,0; 3,05), в котором, видимо, много повторяющихся участков. В геноме на вертикальной оси их больше, и они все сгруппированны. Вероятно, это нужно для регуляции экспрессии генов.