Сборка de novo
1
Данный код доступа проекта по секвенированию бактерии Buchnera aphidicola: SRR4240360.
Архив с чтениями был скачан командой:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/000/SRR4240360/SRR4240360.fastq.gz
Файлы с адаптерами в папке /mnt/scratch/NGS/adapters были объединены в один:
cat ../../adapters/* > adapters.fasta
Далее с помощью программы trimmomatic удаляем остатки адаптеров в ридах:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240360.fastq.gz SRR4240360_out.fastq.gz ILLUMINACLIP:adapters.fasta:2:7:7
После удаления адаптеров количество чтений уменьшилось с 8254632 до 8212774 (99.49%). Удалилось 41858 чтений (0.51%).
Следующий шаг: удаление с правых концов чтений нуклеотидов с качеством ниже 20, а также чтений длиной меньше 32 нуклеотидов:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240360_out.fastq.gz SRR4240360_last.fastq.gz TRAILING:20 MINLEN:32
От 8212774 ридов осталось 7915474 (96.38%), удалено 297300 (3.62%). Изначальный размер файла с 194М на первом шаге уменьшился до 193М, на последнем шаге - до 184М.
2-3
Далее программа velveth формирует k-меры длиной 31 на основе оставшихся ридов (в созданном подкаталоге ./velveth):
velveth ./velveth/ 31 -fastq.gz -short SRR4240360_last.fastq.gz
Программа velvetg выполняет сборку на основе работы velveth:
velvetg ./velveth
Программа выдала значение N50 = 43070. Информация о длинах контигов и их покрытии получена в файле stats.txt. Найти самые длинные из них можно с помощью команды:
sort -k 2 -n -r stats.txt
Результаты приведены в таблице 1.
| ID | length | coverage |
|---|---|---|
| 1 | 113474 | 33.525460 |
| 5 | 83603 | 33.646065 |
| 4 | 64155 | 35.847323 |
При поиске аномально больших/малых покрытий по файлу stats.txt была использована команда:
sort -k 6 -n -r stats.txt
Из аномально больших покрытий были значения 134953, 590, однако их длины равны 1, соответственно в файле contigs.fa они будут отсутствовать. При поиске наименьших покрытий было множество покрытий со значением 1, длины этих контигов были равны 1.
Среди них был найден один контиг, который был длиннее 31 нуклеотида (73) с покрытием 2.671233. Этот контиг в файле contigs.fa: NODE_358_length_73_cov_2.671233. Megablast выровнял последовательность этого контига с АТФ-зависимой ДНК-хеликазой Acyrthosiphon pisum (гороховой тли, в которой живет изучаемая бактерия).
Также был найден контиг NODE_565_length_31_cov_1.612903, однако выравнивание не вывело никаких находок.
4
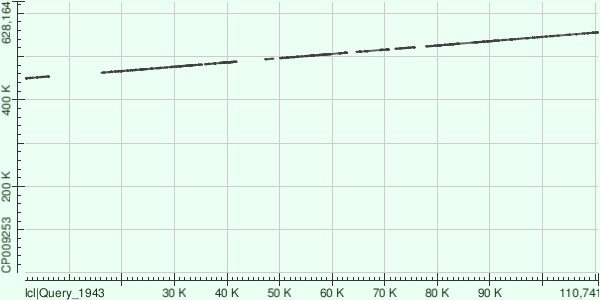
Каждый из трех самых длинных контигов был выравнен с помощью megablast с хромосомой изучаемой бактерии (AC = CP009253). Были получены следующие DotPlot'ы (рис. 1-3).
Выравнивание контига 1 с хромосомой бактерии дало 15 фрагментов (начало и конец фрагмента на хромосоме, процент совпадений, процент гэпов):
449411-454069, 75, 3
462496-467421, 77, 3
467412-474667, 77, 2
474844-480660, 74, 4
480874-481545, 82, 2
481997-488106, 74, 4
493487-494864, 80, 0 (всего 13)
495033-495148, 89, 4
496111-500325, 75, 3
500370-508806, 76, 4
510438-516539, 78, 3
517766-521500, 77, 2
523105-528679, 77, 3
528794-550219, 81, 2
550361-555905, 81, 2.
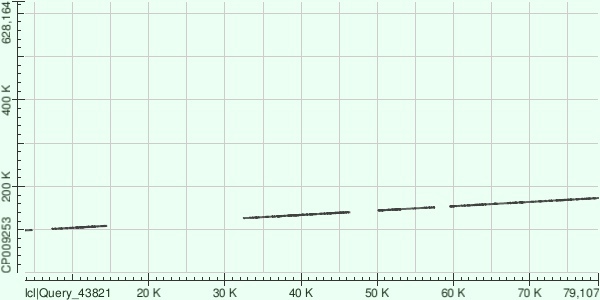
Для контига 5 было найдено 8 фрагментов:
98408-99303, 81, 0 (всего 9)
101712-108876, 77, 2
126623-127815, 84, 0 (всего 11)
127825-140555, 75, 4
144368-151796, 78, 3
153752-161738, 78, 3
161898-166752, 80, 2
166750-173180, 76, 2.
Для контига 4 выровнялось 12 фрагментов.
2004-11103, 78, 2
13994-14465, 82, 1
14727-17919, 76, 2
17962-20182, 85, 1
20358-22183, 82, 2
23067-28363, 77, 4
30013-32745, 77, 3
599832-604795, 78, 3
611229-611524, 79, 0 (всего 2)
611633-613671, 78, 3
613658-620926, 79, 2
621055-627104, 76, 4.
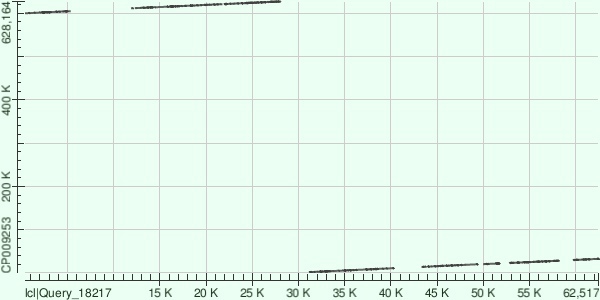
Контиг 1 имеет меньший объем негомологичных участков, несмотря на то, что их количество больше (судя по количеству выровненных фрагментов). Также можно отметить, что в ситуации с контигом 4 на него пришелся ориджин хромосомы (соответственно первая половина контига выровнена с концом хромосомы, вторая половина - с началом). В первых двух случаях с этим проблем нет.