Нуклеотидный BLAST
1
В ходе поиска нужного нам контига в сборке генома Sus scrofa (см. предыдущий практикум) с длиной менее 10000 bp и по крайней мере с одним CDS, выяснилось, что таковых нет. Минимальная длина контига - 15000 bp. В итоге был найден контиг длиной 17924.
Характеристика контига 1960:
идентификатор: NW_018084991.1
длина контига: 17924
координаты mRNA: 12048..17856
координаты CDS: 13,268..17,741
длина CDS: 696 nt
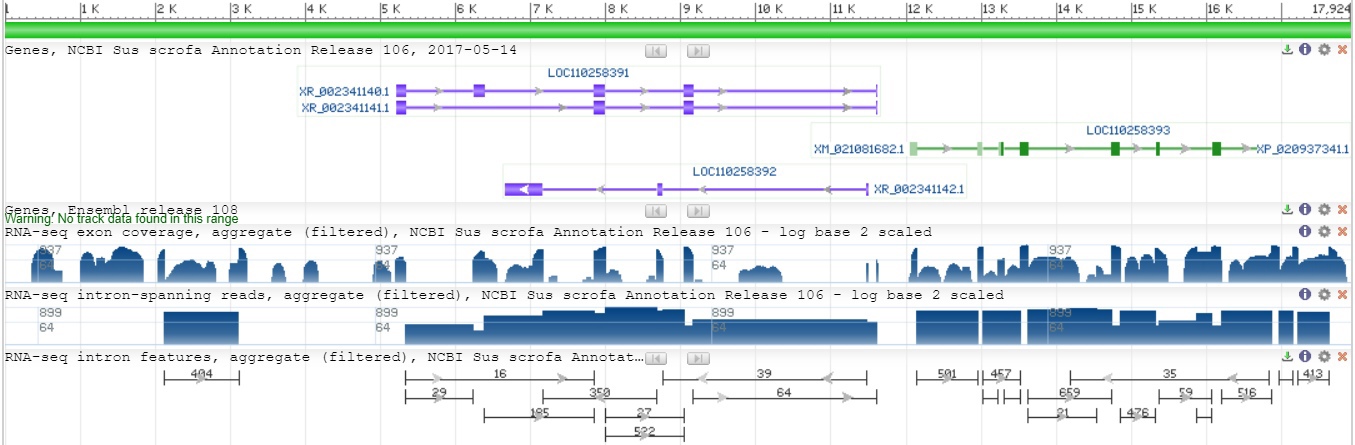
Рис. 1. Расположение генов в контиге
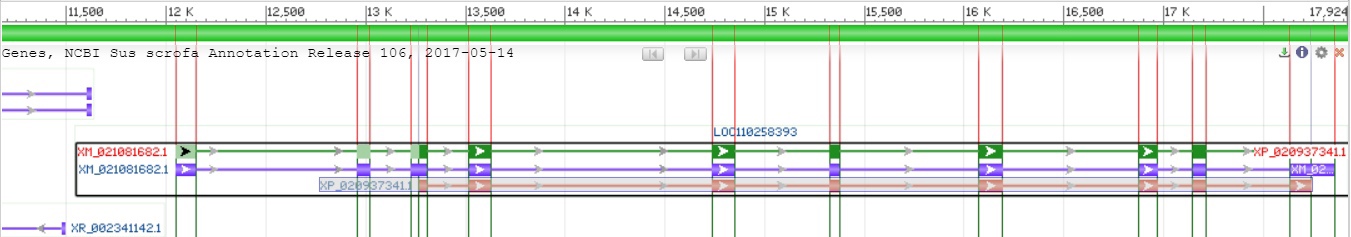
Рис 2. Более подробная схема выбранного фрагмента mRNA
Можно заметить, что контиг содержит 3 участка генов: 2 из них uncharacterized (ncRNA), отмечены на графике фиолетовым, последний (выбранный) кодирует USP6 N-terminal-like protein, выделен зеленым цветом.
Более детальная схема №2 показывает, что CDS начинается на 1220 нуклеотидов позже mRNA (нетранслируемая область). Темно-зеленым отмечены кодирующие экзоны, светло-зеленым - некодирующие. В нетранслируемую область попадают 3 экзона, таким образом, в мРНК видно 10 экзонов, а в CDS уже 8.
Ссылки на последовательности в формате fasta контига и CDS.
Далее был проведен следующий поиск с включением таксона Eukaryota и исключением такона Chordata:
blastn: 157 находок, большинство predicted, max E-value = 4e-15 (параметры: wordsize = 11, max target seqs = 250)
megablast: 48 находок, max E-value = 5e-80 (в эукариотах вне хордовых находок не было; во всех эукариотах находит только в самом роде Sus и родственном Phacochoerus (бородавочник) и, удивительно, у синего кита и тибетского макака, но у последних двух самый высокий показатель E-value)
blastx: 76 находок, max E-value = 2e-09 (постоянно было очень много находок, какие бы параметры не изменялись, причем большинство predicted, low quality, uncharacterized; wordsize = 3, E-value = 5*10-8)
tblastx: ни при каких изменениях любых параметров (woedsie, E-value, таксон, база данных и пр.) не дает адекватных результатов, их или очень много, или результат вообще не выводится.
Таким образом, blastn для нас полезен, когда последовательность белок-некодирующая или когда надо найти не в родственных видах, megablast - для поиска гомологичных последовательностей в близких родах (очень схожих), blastx - когда последовательность кодирует белок.
2
Создание локальной базы данных по геному Sus scrofa производилось с помощью команды:
makeblastdb -in ../db/GCF_000003025.6_Sscrofa11.1_genomic.fna -dbtype nucl -out db.fasta
Далее с помощью команды blastn был произведен поиск гомологичных участков на rRNA E.coli по созданной базе данных. 16S rRNA - малая субъединица рибосомы прокариот, 23S - большая. Команды для 23S и 16S rRNA:
blastn -task blastn -query ../db/16srRNA_ecoli.txt -db db.fasta -out result16.fasta
blastn -task blastn -query ../db/23srRNA_ecoli.txt -db db.fasta -out result23.fasta
Ссылки на файлы с выводом для 23S и 16S.
Использовался blastn из-за того, что нуклеотидные последовательности рРНК бактерии короткие и, возможно, не имеющие сильное сходство с последовательностью кабана + некодирующая последовательность.
Для 23S рРНК выдалось 9 находок, из них с более-менее адекватным E-value (от 0.007) - 6. Самая лучшая находка имеет E-value 5e-17, эта последовательность находится в 7 хромосоме. Этот участок кодирует ncRNA (что может подтверждать то, что этот участок хромосомы кодирует рРНК, однако большей информации нет.
Для 16S также найдено 9 последовательностей, однако лишь 2 из них имеют нормальный E-value (8e-05), оставшиеся больше 0.5. Одна из последовательностей находилась в скэффолде и кодировала 18S рРНК (гомологичны, тоже малая субъединица).