Трансмембранные белки
OPM
Для выполнения данного практикума был выдан идентификатор трансмембранного белка: KBAA_BACSU. В Uniprot в разделе Structure было найдено предсказание трехмерной структуры белка.
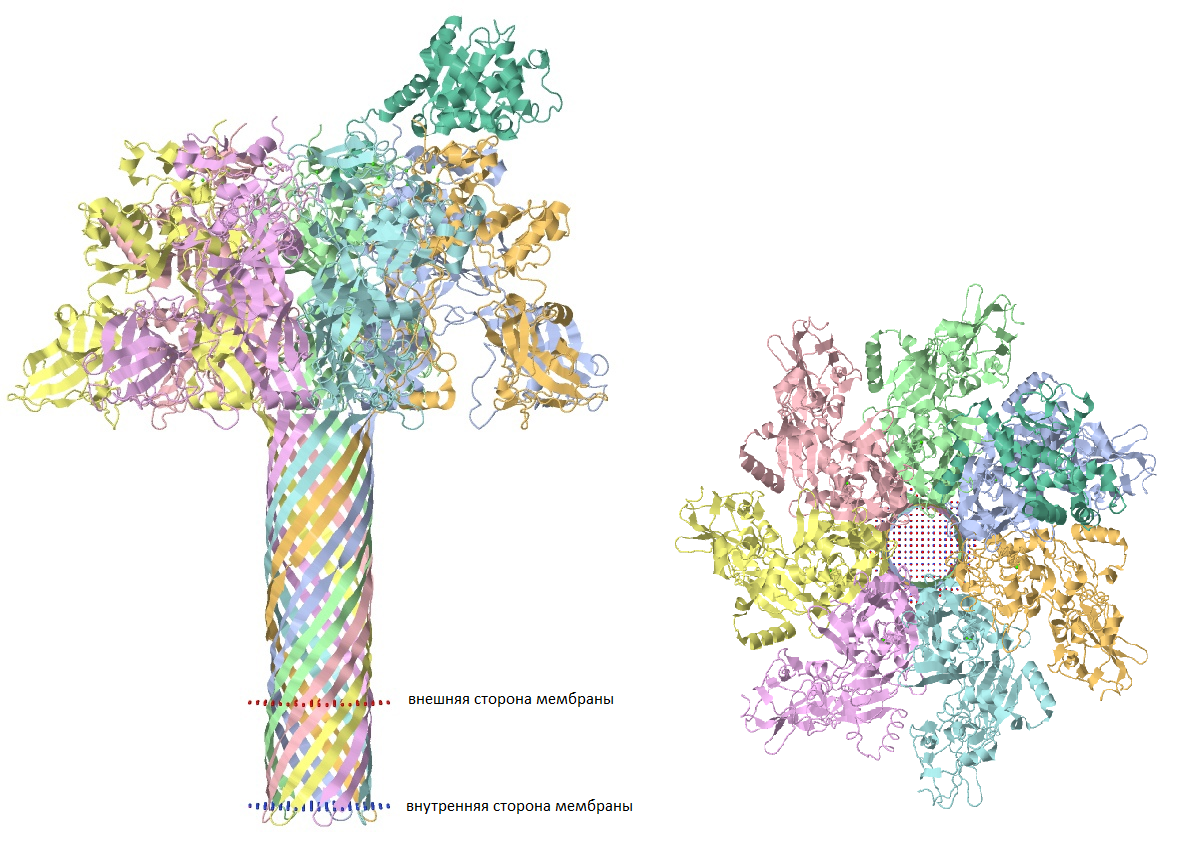
В базе данных OPM (Orientations of Proteins in Membranes database) была найдена структура с β-листами в трансмембранной части с PDB ID 6PSN (рис.1). Эта структура является комплексом летального фактора с антигеном токсина сибирской язвы (Bacillus anthracis) (вызывает клеточную гибель макрофагов). β-бочонок образован антигеном (PAG_BACAN). С помощью OPM были найдены следующие характеристики:
Толщина гидрофобной части белка в мембране: 27.0 Å;
Координаты трансмембранных участков: 302-312, 316-325;
Среднее количество остатков в одном β-тяже белка: 36;
Белок находится на различных мембранах клетки хозяина.
DeepTMHMM
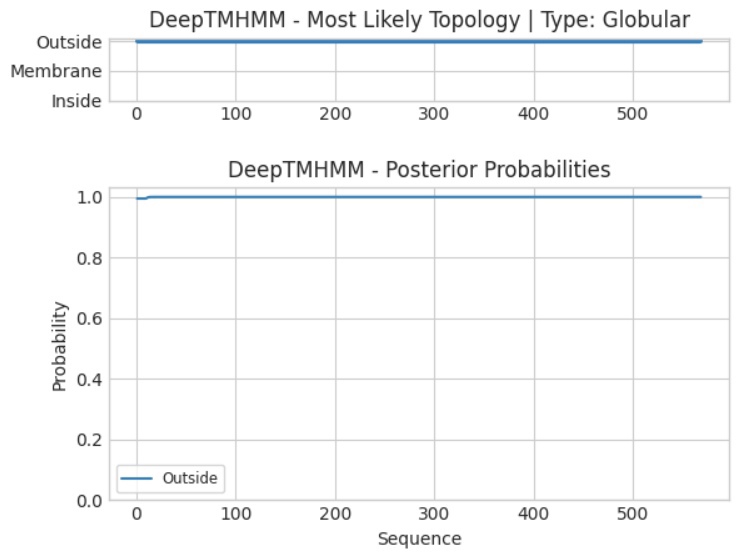
Далее белок KBAA_BACSU и часть структуры 6PSN были проанализированы в сервисе DeepTMHMM, предсказывающем трансмембранные элементы по последовательности белка. Выдача в текстовом формате для KBAA_BACSU и 6PSN (в графическом виде - рис.2, рис.3). Сервис, очевидно, плохо предугадал строение второго белка (на вход подавалась последовательность одной субъединицы антигена), он не "угадал" β-складки (глобулярное строение по всей посдедовательности, но она только вне мембраны). Результат у первого белка оказался лучше, заметно выделяются α-спирали (например, AlphaFold предугадал спираль на 5-28 остатках, на графике примерно те же остатки находятся в мембране). α-спиралей шесть, N- и C-концы находятся по внутреннюю сторону мембраны.
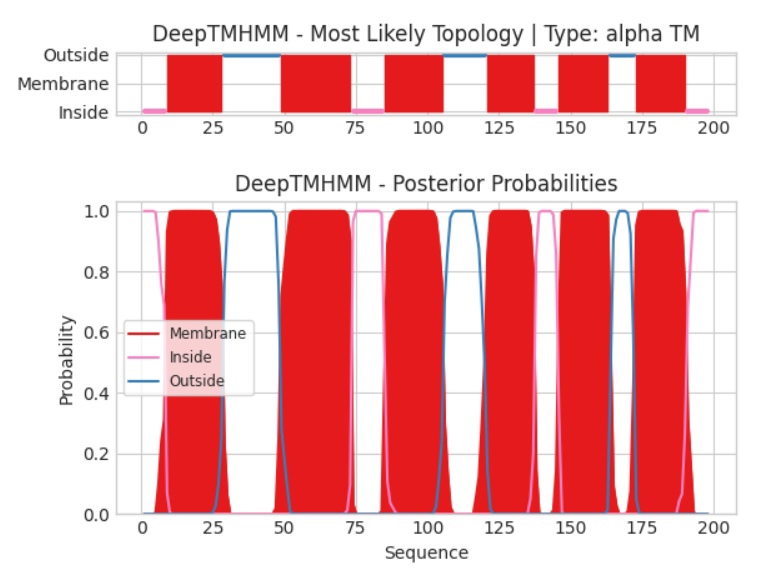
Пояснение к графикам: на оси Y показана вероятность остатка быть на внутренней/внешней стороне мембраны или внутри. На оси X - позиции аминокислотных остатков. Розовая линия показывает веротность нахождения на внутренней стороне, синяя - на внещней, красным обозначено расположение внутри мембраны/
PPM
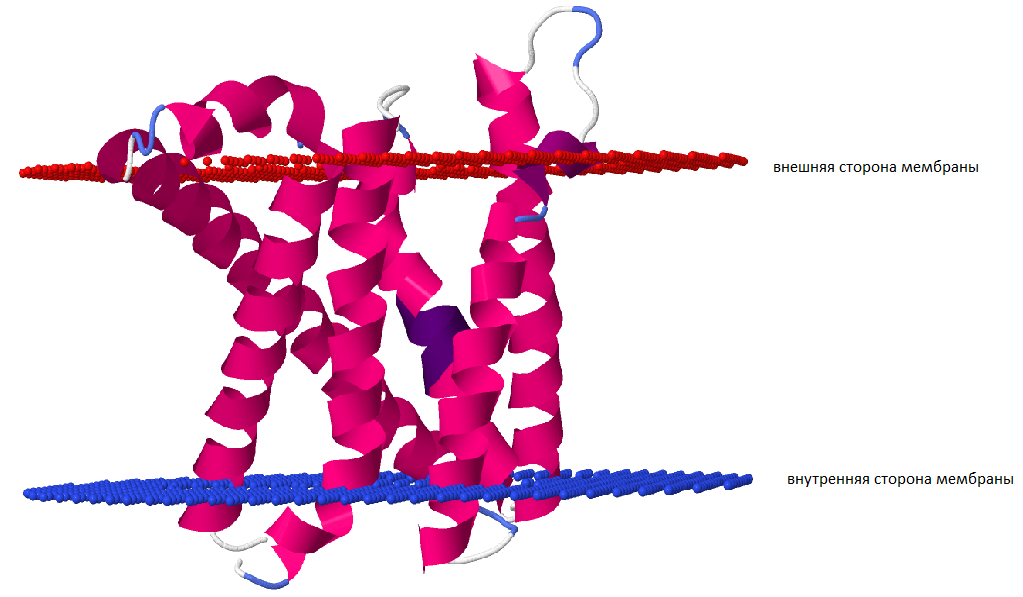
В помощью PPM анализируется активирующий KinB-сигналинг белок (KBAA_BACSU, см. выше). Для него не существует достоверной структуры, однако существует ее модель, которая предсказана с помощью AlphaFold. Оценка достоверности (pLDDT) у этой модели высокая (>70, только 1 остаток на C-конце имеет оценку 70 > pLDDT > 50). С помощью сервиса PPM было предсказано положение этого белка относительно мембраны (рис.4).
Критерии: одна мембрана, мембрана грамположительной бактерии, N-конец на внутренней стороне мембраны (из графика прошлого задания). Результаты работы сервиса:
Толщина гидрофобной части белка в мембране: 26.8 ± 1.7 Å;
Координаты трансмембранных участков: 5-28, 45-77, 86-105, 120-138, 146-166, 173-192;
Среднее количество остатков в одном β-тяже белка: 25 ;
Клеточная мембрана бактерии.
Сравнение алгоритмов предсказания трансмембранных спиралей
KBAA_BACSU: сравнивая результаты работы PPM и DeepTMHMM, можно заметить, что оба сервиса примерно одинаково предсказали строение белка и положение его относительно мембраны. N- и C-концы расположены к внутренней стороне мембраны, 6 трансмембранных элементов (альфа-спиралей), примерно те же номера остатков, которые находятся в этих альфа-спиралях:
DeepTMHMM: 9-28, 49-73, 85-105, 121-137, 146-163, 173-190;
PMM: 5-28, 45-77, 86-105, 120-138, 146-166, 173-192 (различие в несколько остатков).
То же самое можно сказать и про модель, построенную с помощью AlphaFold.
PAG_BACAN: результаты работы DeepTMHMM и данные из OPM не совпадают. DeepTMHMM предсказал расположение исключительно на внешней стороне мембраны, пересечения отсуствуют. Структура 6PSN при этом имеет определенно бета-бочонок. Возможно, из-за того, что очень малая часть бочонка находится внутри мембраны, такую последовательность можно по-другому свернуть (около 10 остатков из 36 в бета-листе находятся в мембране, наверное, это не много).
TCDB
В базе данных TCDB производился поиск двух выше анализирлванных белков. Был найден белок антигена (PAG_BACAN, найден по прямой ссылке из OPM), но белок KBAA_BACSU (по AC P16449) найден не был.
TC-код белка: 1.C.42.1.1. Код означает следующее:
1 - канал;
C - токсины, образующие каналы/поры;
42 - семейство BAPA (защитный антиген Bacillus anthracis).
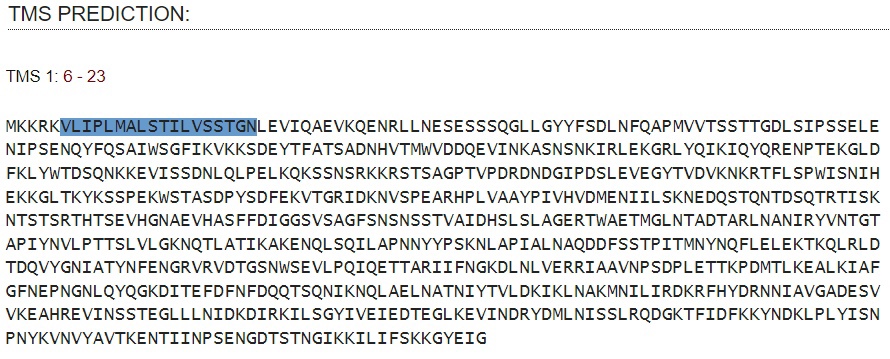
TMS может предсказать трансмембранные участки в последовательности (Predict TMSs)(рис.5). Сервис предсказал один участок из 18 остатков. Это вполне могут быть те 20 остатков в двух бета-листах, расположенных внутри мембраны (что еще раз доказывает неправоту DeepTMHMM).