1. Составление списка гомологичных белков, включающих паралоги
С помощью blastp были найдены гомологи белка CLPX_ECOLI в протеомах бактерий из предыдущих практикумов. 8 протеомов были объединены в файл all.fasta.
makeblastdb -dbtype prot -in all.fasta -out dbase
blastp -query ECOLI.fasta -num_threads 4 -db dbase -evalue 0.001 -out results.txtфайл с результатом
Найдена 21 находка. Находки с наименьшим E-Value - АТФ-зависимые Clp протеазы всех восьми бактерий (ATP-dependent Clp protease).
2. Реконструкция и визуализация
Было реконструировано дерево найденных гомологов программой FastME со следующими параметрами:
Gamma distributed rates across sites — No (чтобы программа не работала медленнее)
Starting tree (начальное дерево, алгоритм) — BIONJ
100 бутстреп реплик
Результат в файле (newick)
Можно выделить пары (допустим, три) паралогов:
CLPX STRAW и Q82EB8 STRAW
FTSH MYCTU и CLPX MYCTU
Q8G3S2 BIFLO и CLPX BIFLO
и ортологов:
CLPX MYCLE и CLPX MYCTU
CLPX LEIXX и CLPX CLAMS
Q6ACQ0 LEIXX и B0RHW4 CLAMS
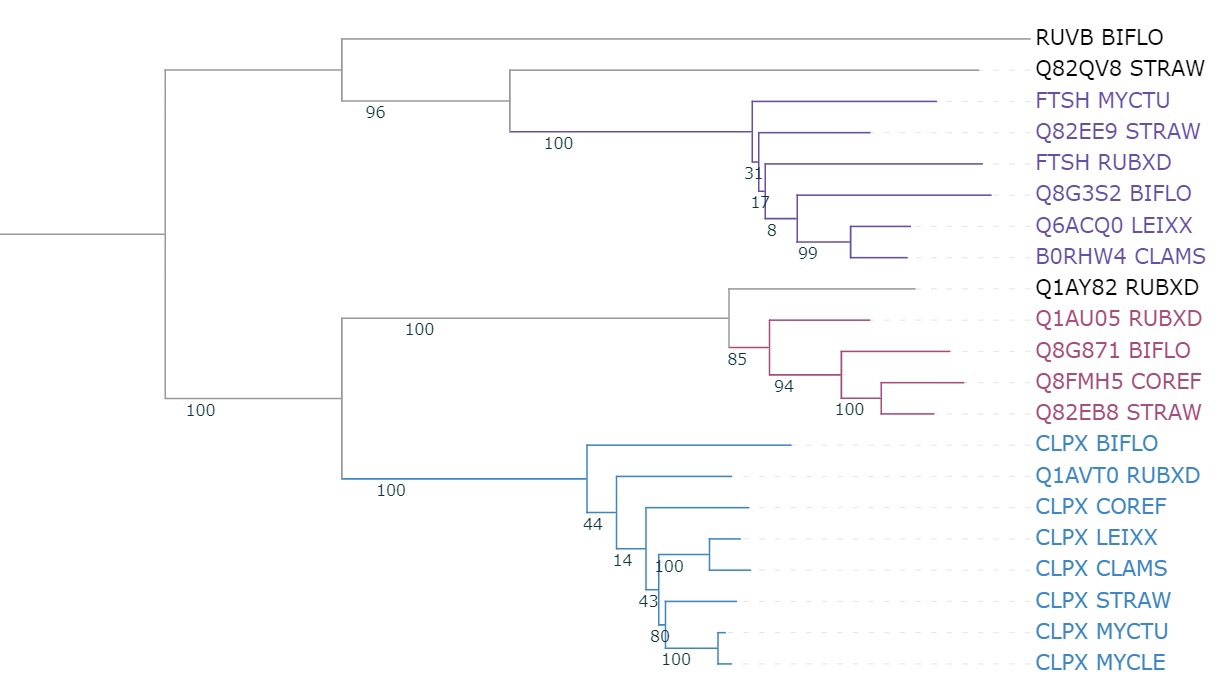
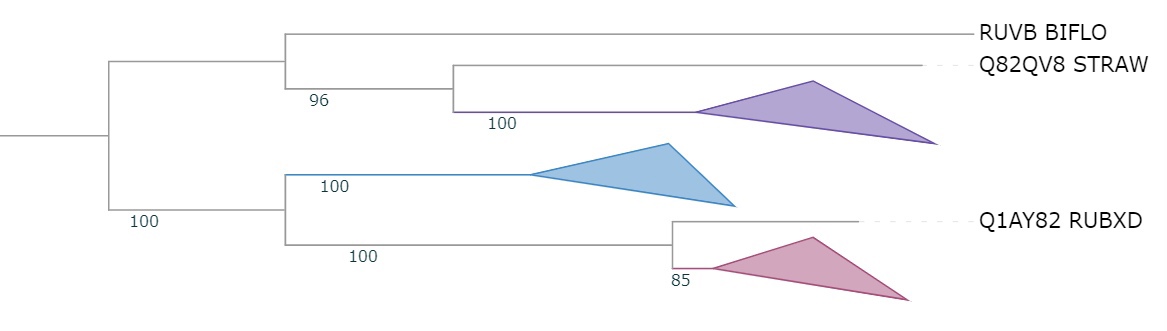
Пояснение к рисунку 2.
Группа, выделенная голубым цветом: АТФ-зависимые Clp протеазы. В группе находятся все белки, филогения белков не полностью совпадает с филогенией бактерий (BIFLO отделяется раньше всех, изначально был в ветви с CLAMS, LEIXX; STRAW относится к ветви MYCLE, MYCTU, изначально к CLAMS, LEIXX, BIFLO; COREF не входит в ветвь MYCLE, MYCTU)
Группа, выделенная розовым цветом: АТФаза ААА-2 (RUBXD), протеаза (BIFLO), АТФ-зависимая Clp протеаза (STRAW), эндопептидаза Clp (COREF). В группу вошло 4 белка. Филогения не совсем совпадает с филогенией организмов, но RUBXD в обоих деревьях выделяется раньше всех. Здесь также переместился BIFLO.
Группа, выделенная фиолетовым цветом: АТФ-зависимые цинковые металлопротеазы FtsH. Группа представлена 6 белками (нет MYCLE, COREF). Здесь тоже филогения не совпадает полностью. Совпадает ветвь с BIFLO, LEIXX, CLAMS, но самый близкий лист к ней - RUBXD, а не STRAW. MYCTU стал отходить самым первым.