Сигналы и мотивы
1 - IRES (Internal Ribosome Entry Site) - сайт внутренней посадки рибосомы
IRES - регуляторный участок мРНК, обеспечивающий кэп-независимую инициацию трансляции. Большинство молекул мРНК, которые используют IRES для трансляции, кодируют белки, участвующие в транскрипции, развитии клеточного цикла, апоптозе и выживании клеток. Встречается у эукариот и вирусов.
Структуры эукариотических IRES очень разнообразны, и в них не выявлено никаких консервативных последовательностей и мотивов. Вторичная структура IRES, образующая шпильку, может как активировать, так и ингибировать трансляцию.
Источники:
Lewis, S M; Holcik, M (2007). For IRES trans-acting factors, it is all about location. , 27(8), 1033–1035. doi:10.1038/sj.onc.1210777
https://en.wikipedia.org/wiki/Internal_ribosome_entry_site
2 - Построение PWM для последовательности Козак Homo sapiens
Для выполнения задания был использован код Георгия Муравьева, ссылка на скрипт. На вход подается таблица генов человека, выводит kozak-learn.fasta, kozak-test.fasta и pseudokozak1.fasta – файлы для обучения, тестирования и негативного контроля.
По выравниваниям без гэпов из тестовой выборки была построена PWM:
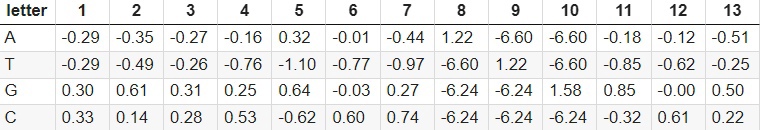
Таблица 1. PWM для последовательности Козак Homo sapiens
На основе данной таблицы были посчитаны веса всех последовательностей и построена гистограмма распределения (рис.2).
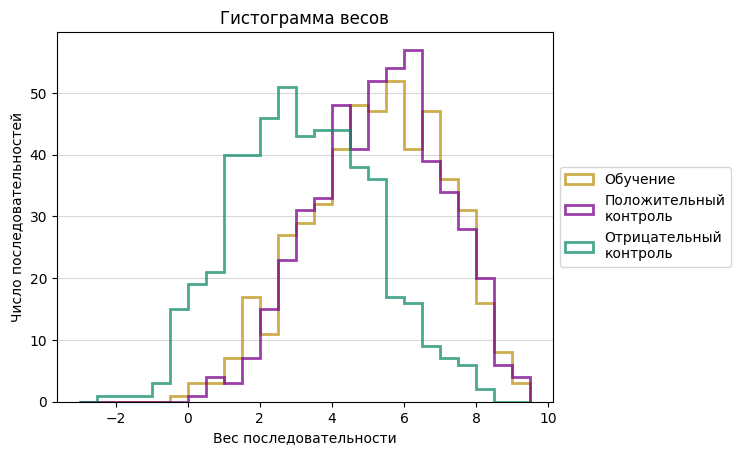
Рис. 2. Гистограмма распределения весов последовательностей
Весы последовательностей отрицательного контроля сдвинуты влево относительно весов последовательностей обучения и положительного контроля. Порог для "отделения" отрицательного контроля от остальных последовательнстей был взят за значение 4 (на гистограмме на этом значении вес отриц. контроля значения пересекаются, при значении выше вес отриц. контроля явно меньше веса остальных последовательностей). На основании этого строилась матрица результатов проверки (таблица 2).

Таблица 2. Матрица результатов проверки
При пороге, равном 4, большая часть последовательностей отрицательного контроля попала в сигнал(-), а последовательности обучения и положительного контроля - в сигнал(+).
3 - Информационное содержание последовательности Козак H.sapiens
Для вычисления информационного содержания последовательности Козак строилась матрица информационного содержания:
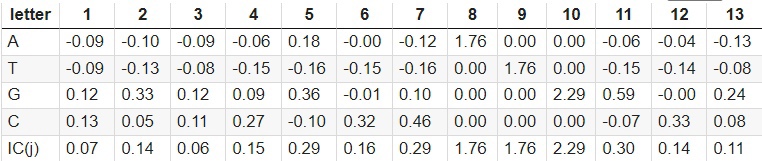
Таблица 2. Матрица IC для последовательности Козак Homo sapiens
С помощью сервиса WebLOGO 3 было визуализировано ИС последовательности. Последовательности длиной 7 нуклеотидов до ATG и 3 нуклеотида после имеет значимый информационный вес. Однако они не так значимы, как старт-кодон.
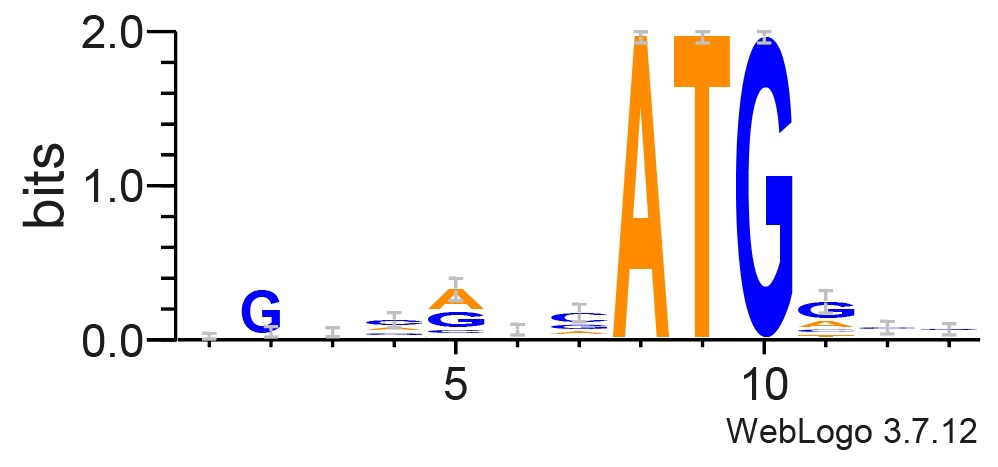
Рис 2. Визуализация ИС последовательности Козак Homo sapiens
4 - Подсчет числа сайтов GAATTC в полном геноме одного штамма E.coli
С помощью скрипта было посчитано количество GAATTC-сайтов в Escherichia coli str. K-12 substr. MG1655. Их оказалось 646. Ожидаемое число GAATTC было подсчитано как произведение частот соответствующих оснований, умноженное на длину генома (4675188) = 1141, что является намного больше полученного числа сайтов (почти в два раза). Для оценки статистической значимости использовался Хи-квадрат тест, полученное pvalue=1.13784066848064e-31, отличие статистически значимое.