Сигналы и мотивы - 3
Консервативный мотив в выравнивании последовательностей гомологичных белков
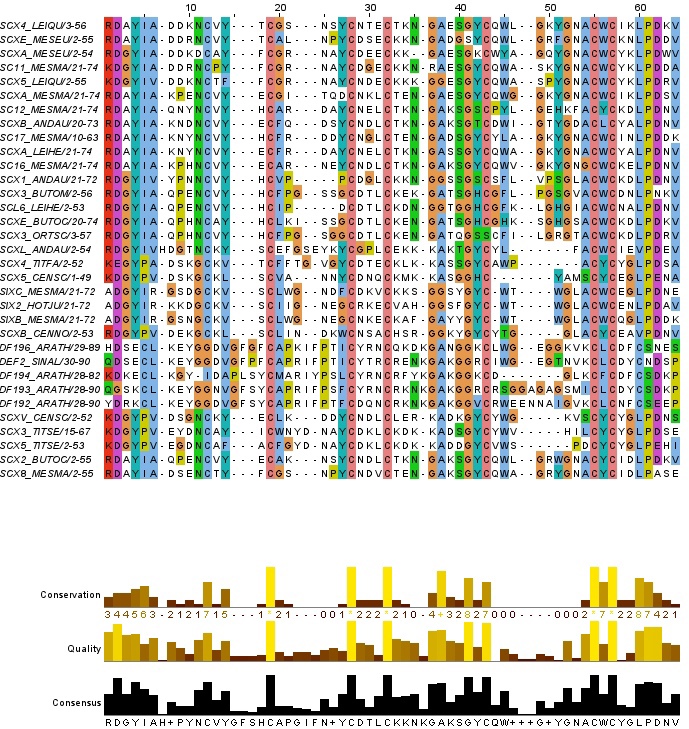
Для выполнения задания было выбрано семейство Toxin_3 (PF00537) (токсины скорпиона). Выравнивание seed содержит 33 последовательности. Далее выравнивание было открыто в JalView (рис.1).
Были найдены консервативные мотивы
[RAK][D].[Y][IPC][AVR] (27 находок в выравнивании)
[GK][GA]..[G][YSH][CS] (24 находки в выравнивании)
[C][WLY][C]..[LC][PS][DE] (26 находок).
С помощью сервиса MyHits производился поиск мотивов по базе данных SwissProt.
Результаты по первому (1837 находок), второму (3966 находок), третьему (196 находок) мотивам. Очевидно, что самым удачным найденным мотивом является третий. 188 из них имеют в своем названии "toxin", что говорит о том, что этот мотив часто встречается в белках-токсинах.
Мотив, специфичный для одной клады филогенетического дерева
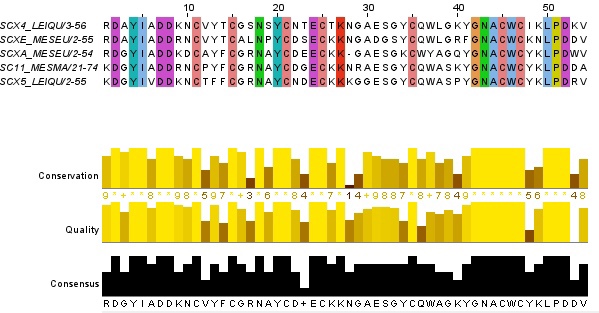
В JalView было построено филогенетическое дерево с помощью NJ. Была выделена клада из 5 последовательностей (много единичных клад, в которых есть мотив, но они отходят раньше, чем клада, в которой мотив менее консервативен)(рис.2). Был выделен еще более длинный мотив [G][N][A][C][W][C][YIK][KN][L][P][D], который встречается во всех последовательностях клады (рис.3). При этом при поиске по всем последовательностям нашлось 7 таковых с данным мотивом, то есть в двух последовательностях вне клады (рис.4). Можно допустить, что этот мотив присущ для этой клады.


PSI-BLAST
Для выполнения задания был выбран белок P19954 (Spinacia oleracea, участвует в ингибировании трансляции за счет связывания с A и P-сайтами рибосомы).
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 17 | P30334.1 | 0.004 | - | - |
| 2 | 28 | P9WMA8.1 | 0.003 | Q0C0T0.1 | 0.027 |
| 3 | 28 | P9WMA8.1 | 7,00E-13 | - | - |
| 4 | 28 | P9WMA8.1 | 8,00E-13 | - | - |
Проверка гипотезы о том, что число TA в геноме меньше ожидаемого по статистике
Для выполнения задания был выбран геном бактерии Thermogutta terrifontis (штамм R1) из практикума 7.
Всего нуклеотидов: 4810751
Число A: 1024594
Число T: 1027685
Таким образом, ожидаемое число TA в геноме = 218877. С помощью wordcount программы EMBOSS было посчитано наблюдаемое количество TA, равное 107278. Таким образом, ожидаемое значение в два раза больше, чем наблюдаемое.