Домены и профили
Выбор домена и доменной архитектуры
Для данного практикума был выбран домен Head_binding (PF09008). Его характеристики:
средняя длина: 108.5
full: 71
seed: 4
%id: 51
average coverage: 17.86
число доменных архитектур: 8

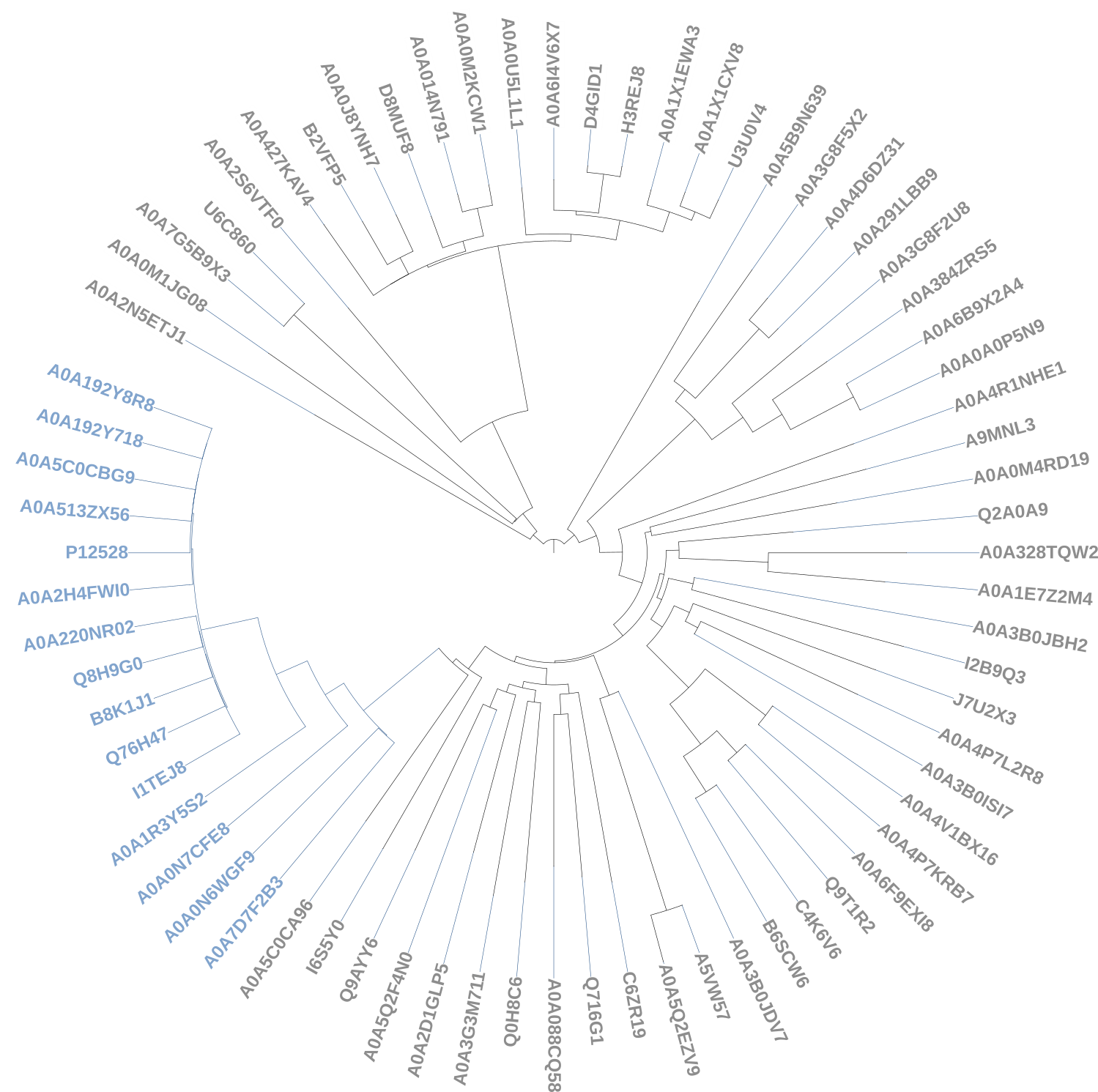
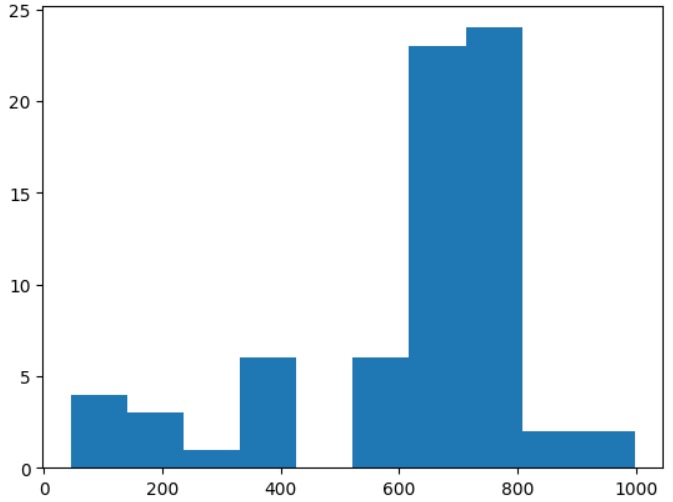
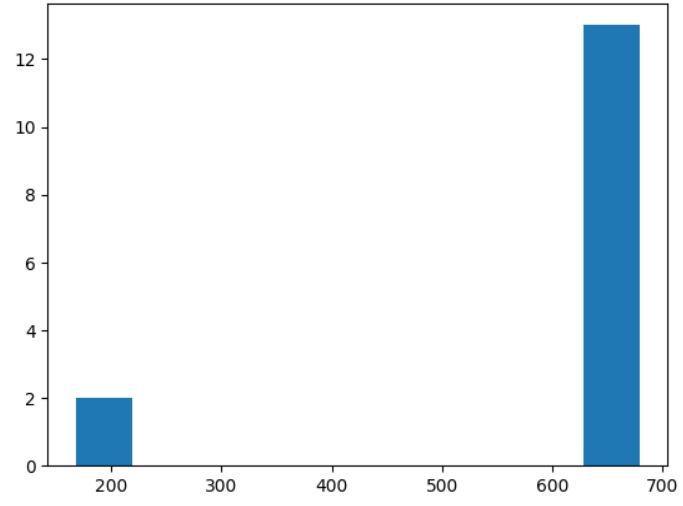
Также была выбрана двухдоменная архитектура Q8H9G0 (рис.1), которая содержится в 15 белках. Распределение длин последовательностей семейства и выбранной архитектуры отображены на рис. 2 и рис. 3.
Построение HMM-профиля
Далее был скачан файл с последовательностями выборки full в формате fasta и создан файл с последовательностями белков с выбранной архитектурой. С помощью JalView последовательности с архитектурой были выравнены. Были удалены лишние участки (второй домен), а также удалены последовательности, совпадающие на 100%. Осталось 6 последовательностей из 15 (рис. 4).

Для получения HMM-профиля был использован пакет HMMER. Команды:
hmm2build HMM ./pr9/6seq.fa
hmm2calibrate HMM
hmm2search --cpu=1 HMM ./pr9/korostina-full-71.fasta > output.txt
Программа выдала HMM-профиль, имеющий длину 115, а также таблицу с находками.
В результате была получена таблица, содержащая сведения о всех последовательностях, содержащих домен Head_binding (и об их вхождениях в выбранную архитектуру, в выборку для HMM-профиля и в находки).
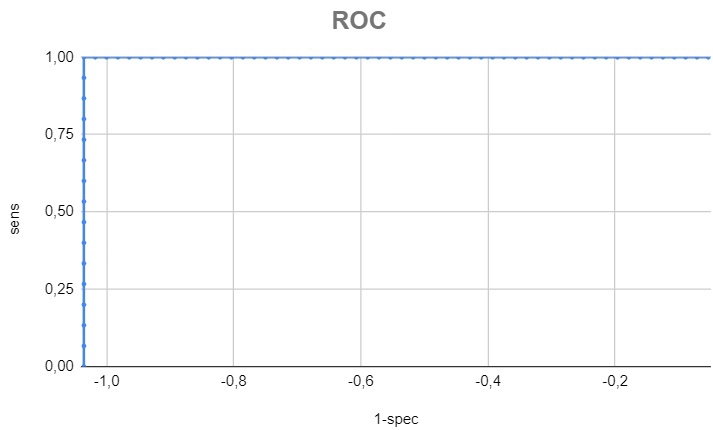
Далее исходя из данных были посчитаны sensitivity, 1-specificity, F1 и построен график ROC (рис. 5)
Построение филогенетического дерева
На основе выравнивания последовательностей, содержащих этот домен, было построено филогенетическое дерево (рис. 6). Голубым отмечены последовательности, в которых содержится выбранная архитектура. Можно заметить, что все они находятся в одной кладе, а других последовательносей в ней нет.