ColabFold (AlphaFold)
Задание B4
Для выполнения практикума были даны три последовательности (seqA, seqB, seqC). Отличие последовательностей seqB и seqC от seqA заключается в замене нескольких аминокислот на пролин (в частности, в seqB – 4 а.о, в seqC – 17). Пролин обладает конформационно жесткой структурой и сильно изгибает пептидную цепь. Также пролин не может быть донором водорода при формировании водородной связи. Участки белков с высоким содержанием пролина могут образовывать вторичную структуру полипролиновой спирали.
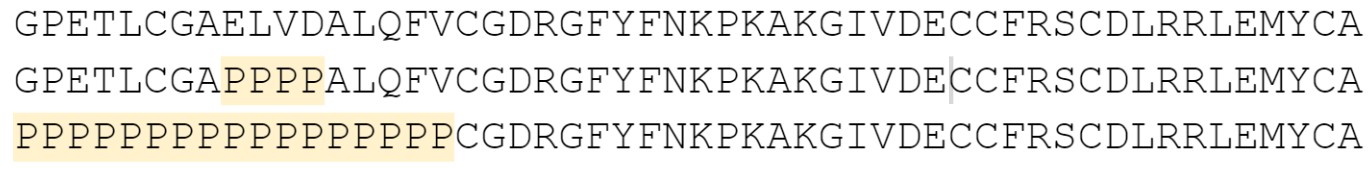
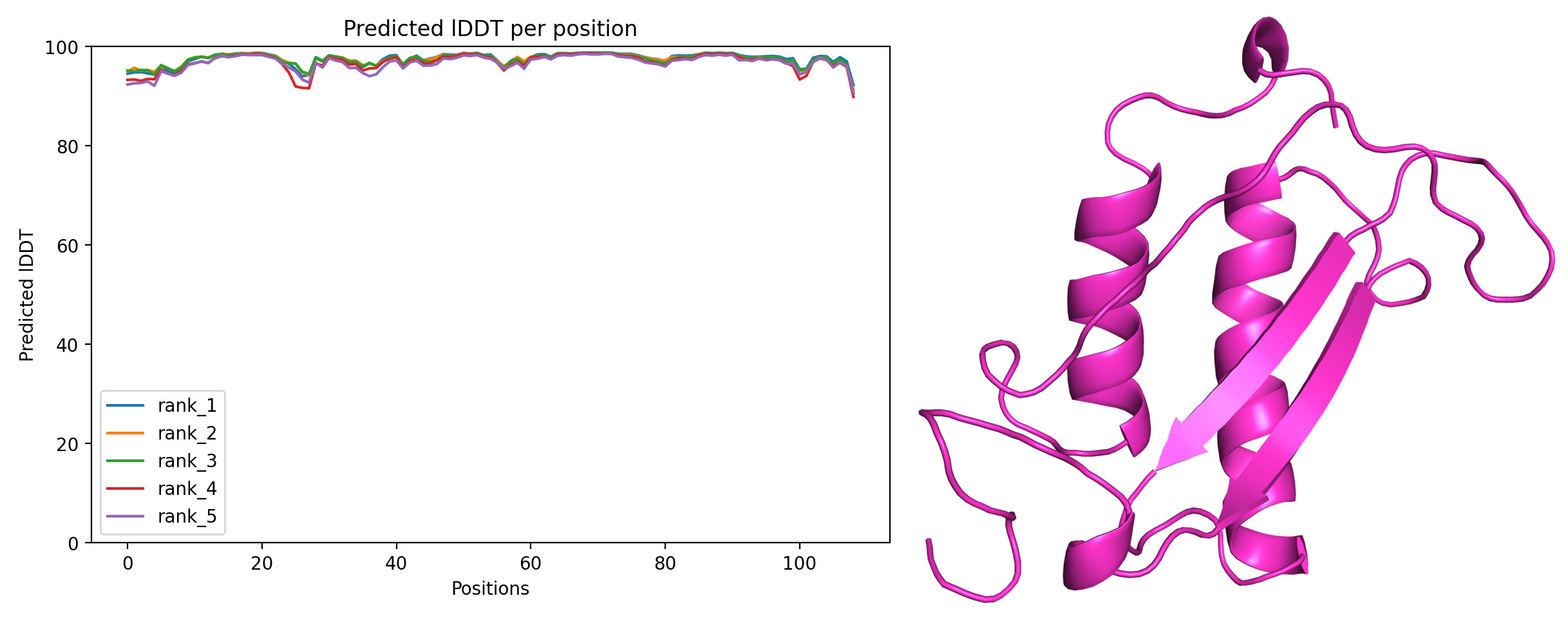
Была предсказана структура для seqA с помощью ColabFold. Результат на рисунке 1. Можно предположить, что изменится первая (синяя) альфа-спираль. В seqB этот участок не сложится в спираль (пролинов мало + не с самого начала последовательности), а в seqC пролины могут образовать свою вторичную структуру (их достаточное количество + в начале).
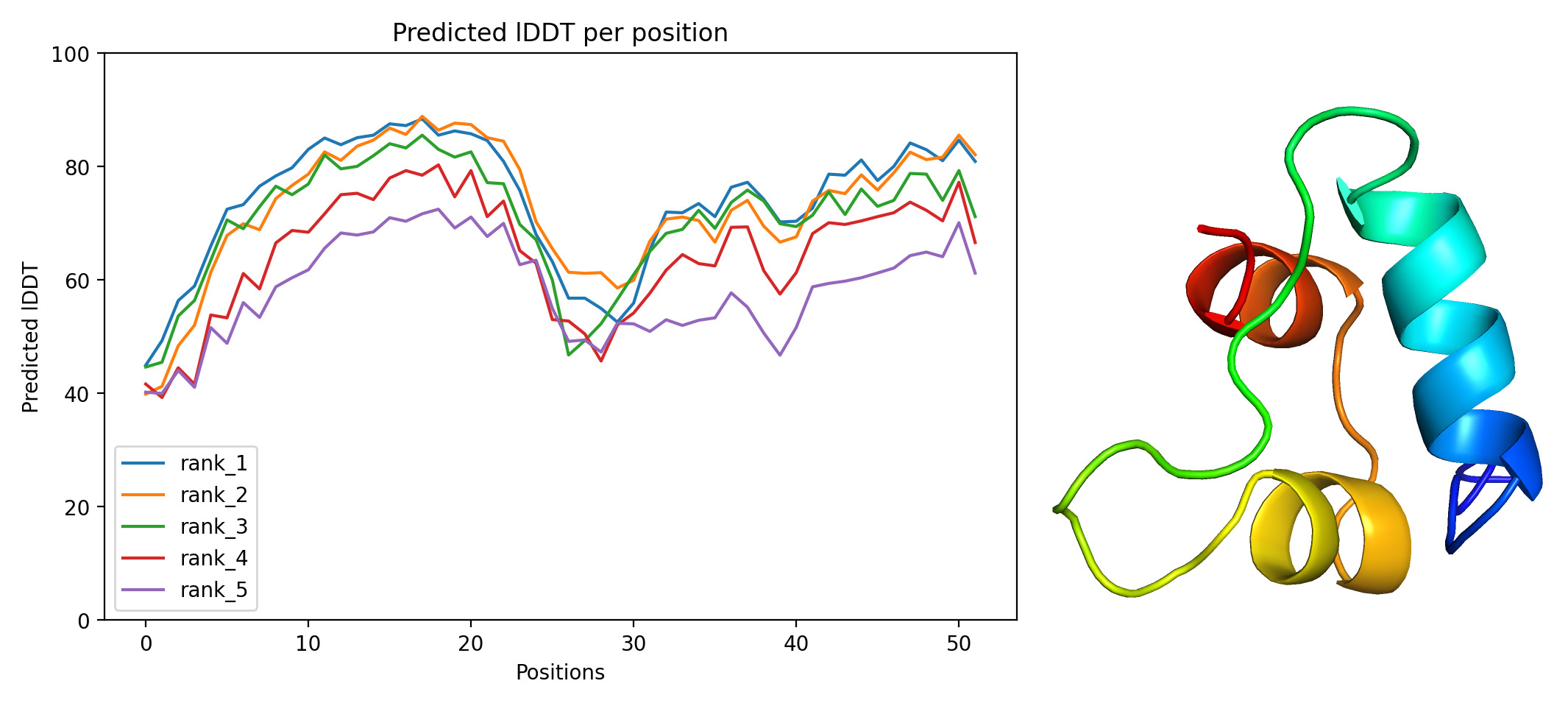
Лучшая находка blastp по seqA соответствует цепи А инсулиноподобного фактора роста человека (PDB: 1TGR).
Выравнивание структуры 1TGR и seqA (рис. 2) показало, что общее предсказание верно. Некоторые альфа-спирали недопредсказаны, а также не были выявлены бета-слои.
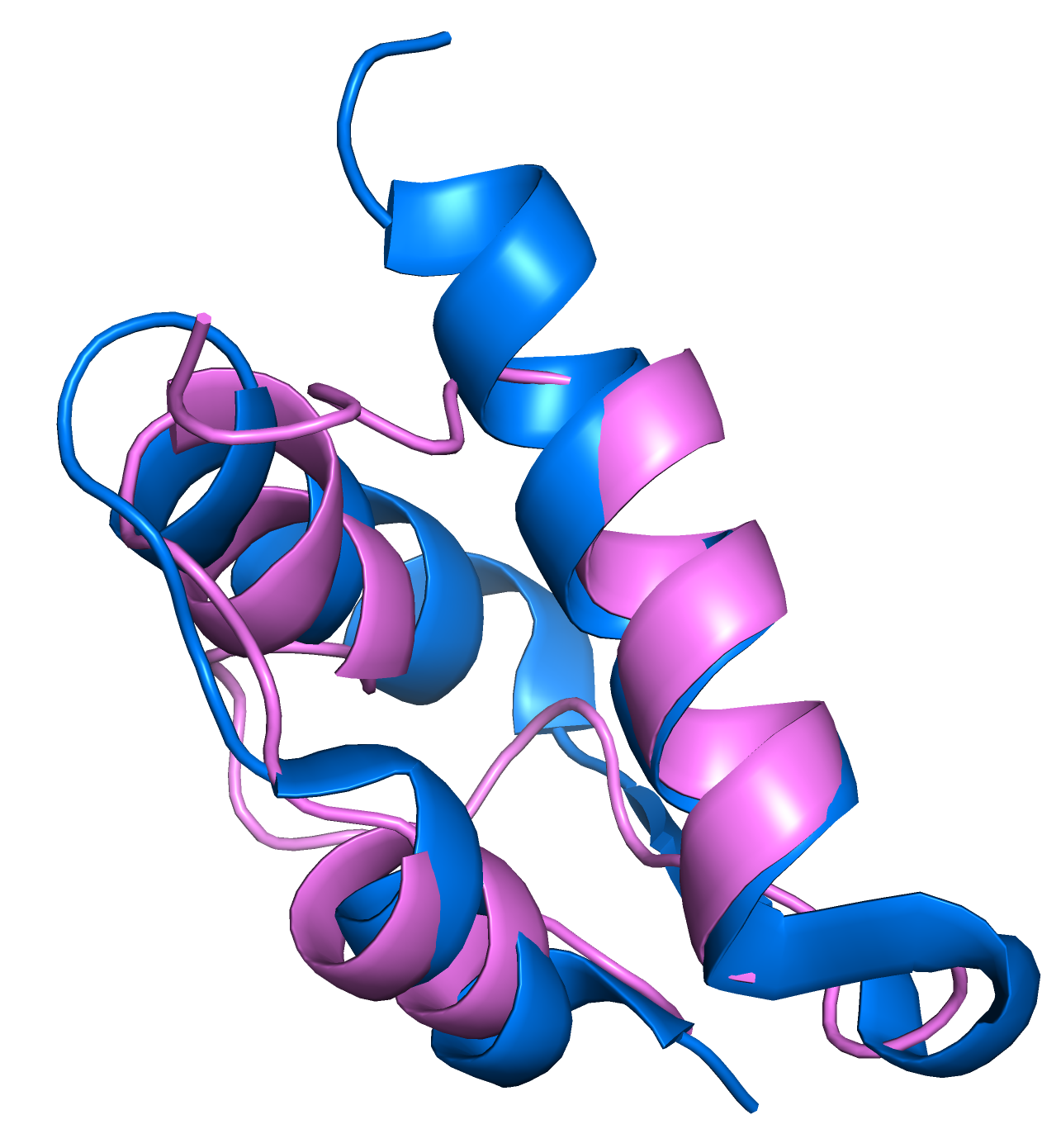
Далее были рассмотрены предсказания структур seqB и seqC. В предсказании seqB (рис. 3), как и следовало ожидать, частично “разрушена” структура альфа-спирали, она стала короче, а N-конец имеет несколько разные положения в пространстве. Никакой дополнительной стабильной структуры не наблюдается. Однако далее по ходу последовательности структуры практически идентичны. По графику можно заметить, что значение model confidence на участке около 10 позиции (где находятся пролины) сильно снизилось.

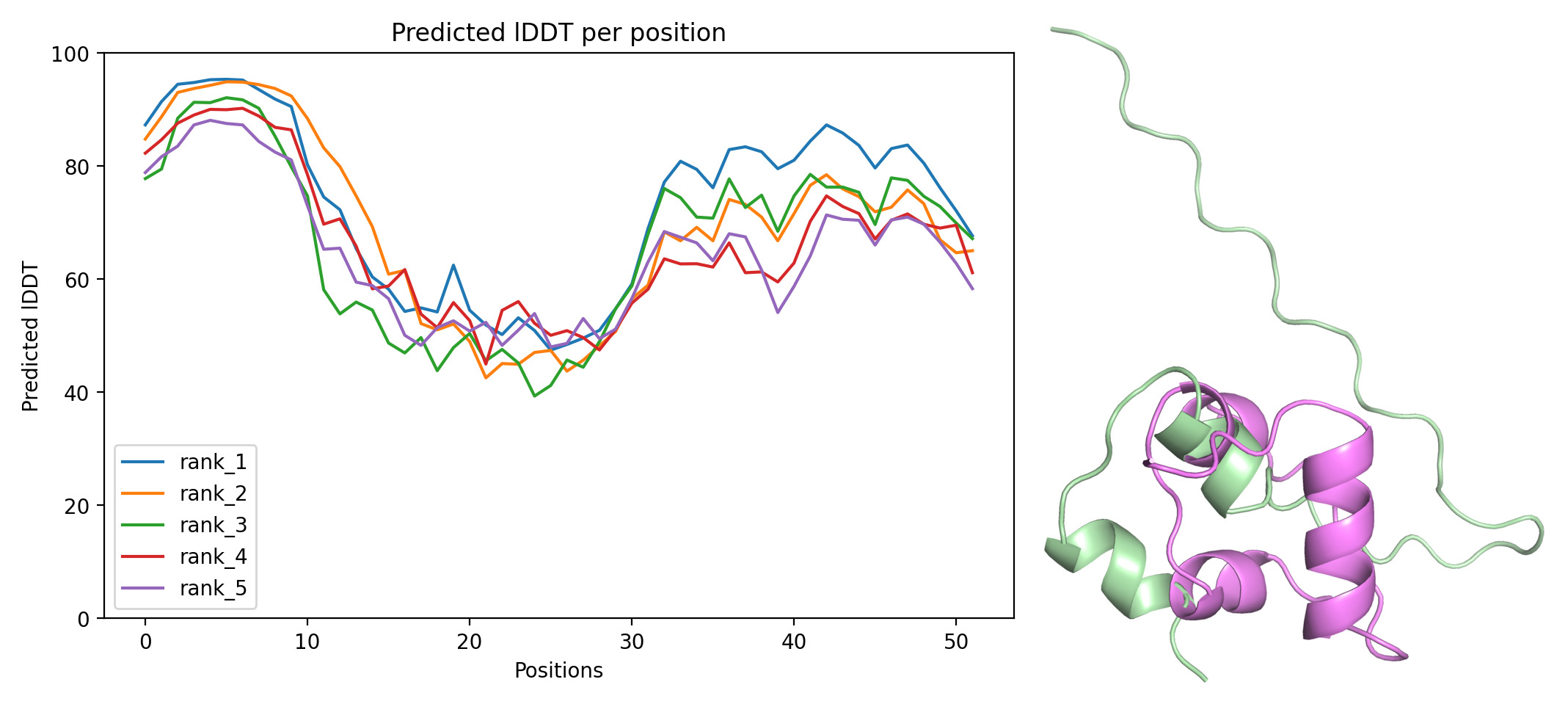
При рассмотрении предсказания структуры seqC и ее последующего выравнивания с seqA (рис. 4) видно образование полипролиновой спирали, последующий участок последовательности очень плохо выровнялся и предсказался. По графику видно очень высокое значение model confidence на пролиновом участке и очень низкое после него.
Ссылка на сессию PyMOL здесь.
Задание D4
Дано две последовательности, seqA и seqB. seqB отличается от seqA N-концевой делецией.

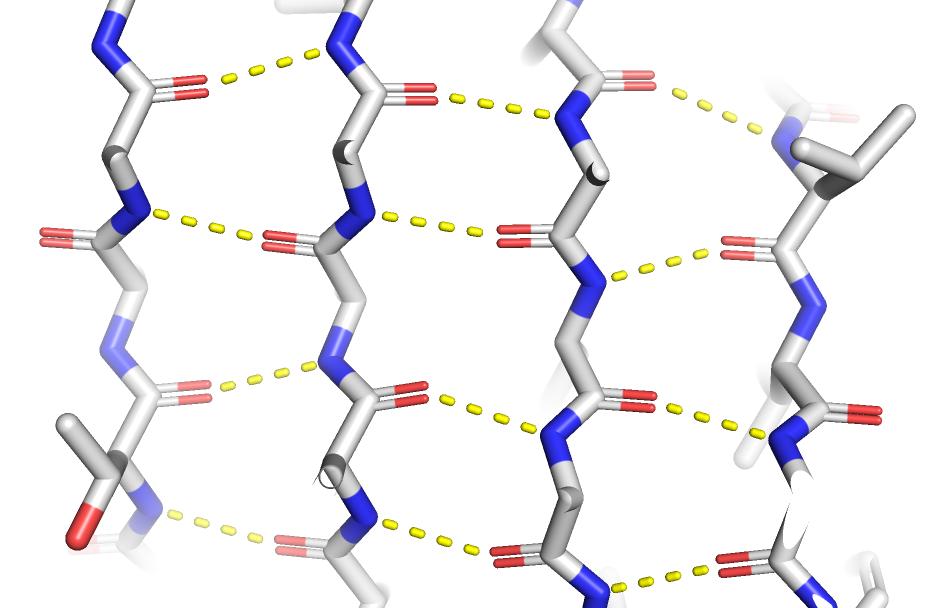
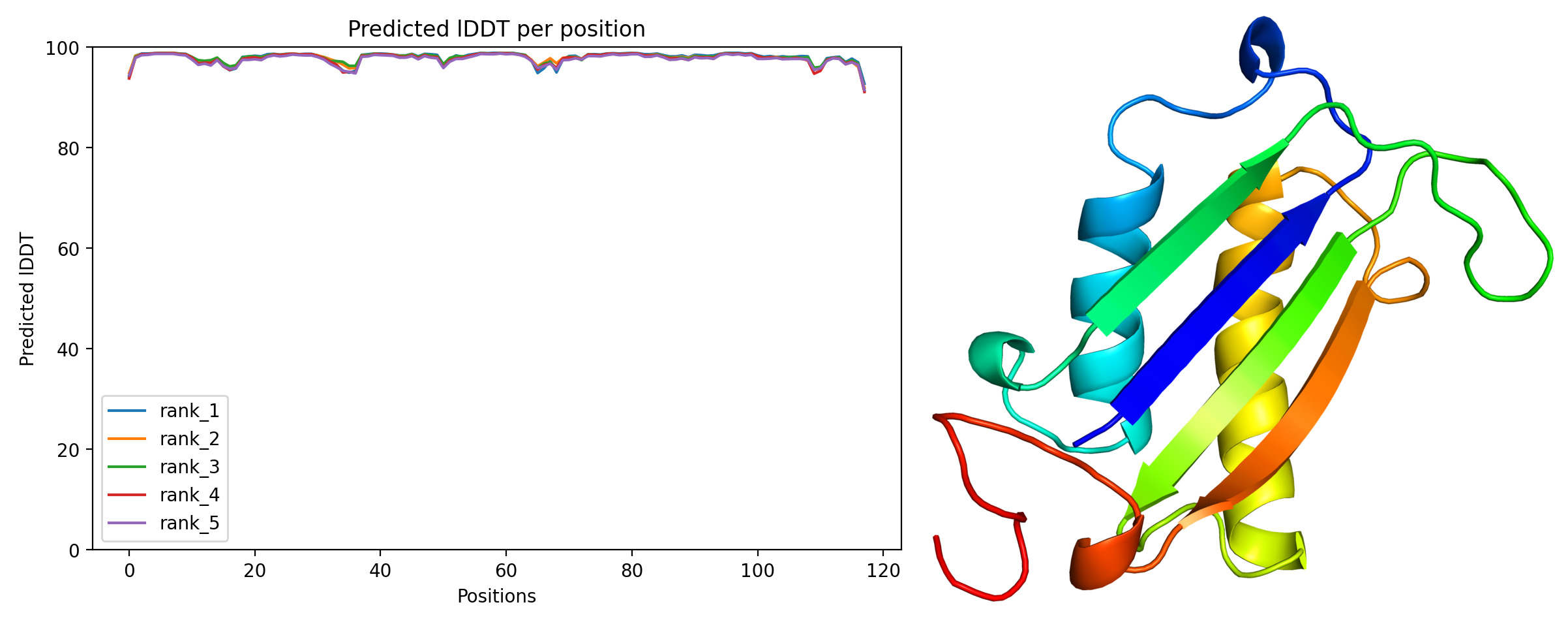
Была предсказана структура последовательности seqA (рис. 6). График показывает очень высокие значения model confidence, практически во всех участках оно близко к 100. Структура образует 4 бета-цепи, которые взаимодействуют друг с другом за счет образования водородных связей (рис. 5), и 2 альфа-спирали.
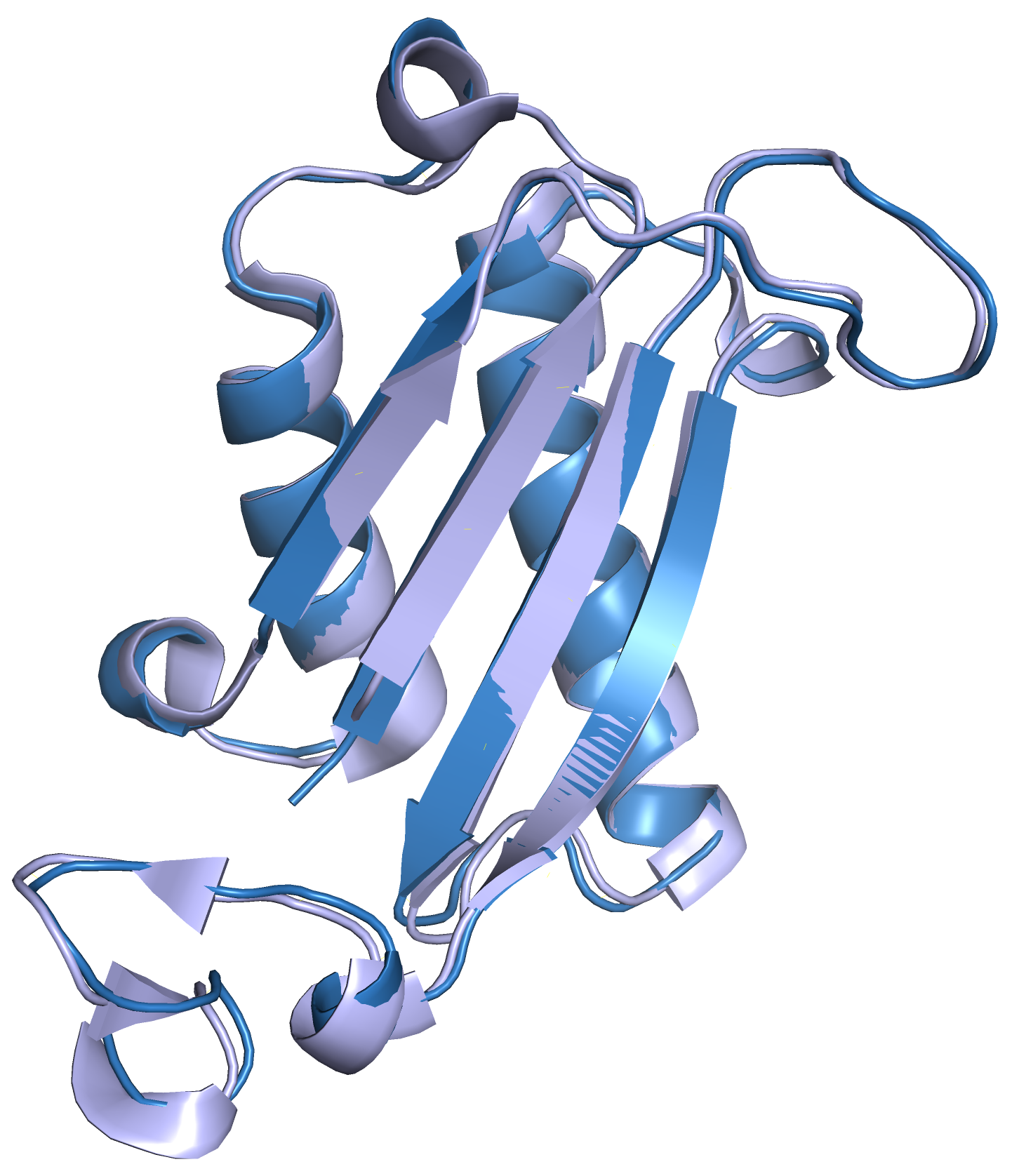
Лучшая находка blastp по seqA соответствует D-дофахром декарбоксилазе человека, цепи А (PDB: 8DBB). Она была выровнена по seqA (рис. 7). Структура очень хорошо предсказана, различия единичны в длине альфа-спиралей или бета-слоев, положение цепи в пространстве идентично.
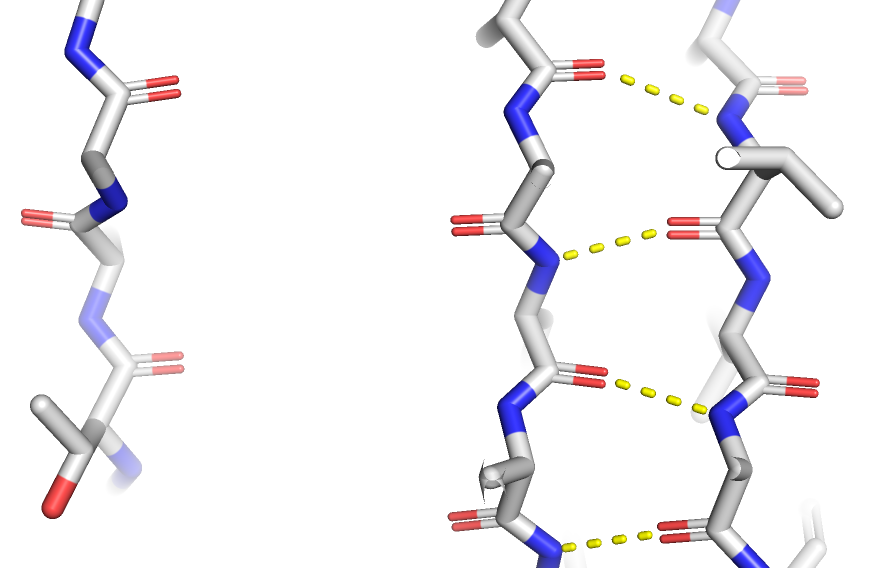
Далее была предсказана структуры seqB с делецией на N-конце (рис. 9). В ней отсутствует одна из бета-цепей (вторая по порядку слева), соответственно, водородные связи образуются между 3 и 4 цепями, а первая не формируется в устойчивую структуру (рис. 8). На графике значение model confidence практически не изменилось. Из этого можно сделать вывод, что предсказание структуры сильно опирается на уже известные. Делеция одной из бета-цепей делает невозможным сворачивание белка именно таким способом, так как такая структура не будет поддерживаться достаточным количеством водородных связей.
Ссылка на сессию PyMOL здесь.