DOMAK
Задание 1
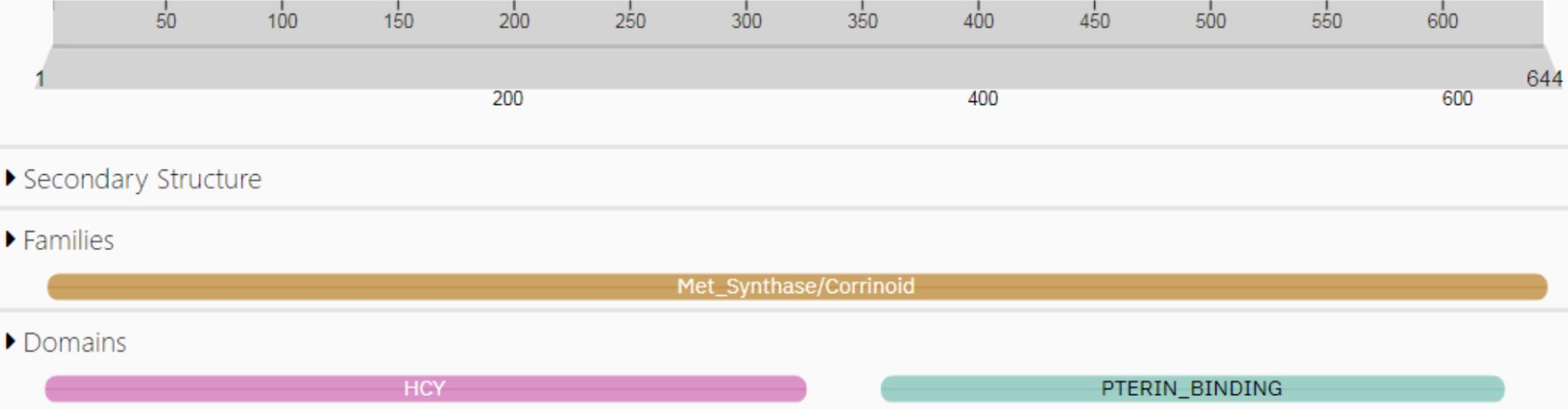
Для выполнения практикума была дана структура 4CCZ 5-метилтетрагидрофолат-гомоцистеин метилтрансферазы человека, участвующая в биосинтезе метионина. Структура содержит домены, связывающие гомоцистеин и фолат.
С помощью алгоритма DOMAK можно предсказать наличие доменов в структуре. Он использует метод SplitValue (индекс разделенности), который предполагает, что остатки внутри домена будут образовывать много контактов, тогда как между доменов взаимодействий быть практически не должно.
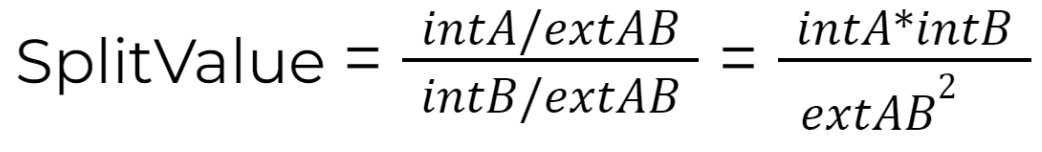
(где intA – число пар контактирующих остатков из участка A (до некоего остатка), intB – число пар контактирующих остатков из участка B (после некоего остатка), extAB – число пар контактирующих остатков из двух участков)
На рисунке 1 изображен график, отображающий значение SplitValue для каждого остатка структуры (ссылка на ноутбук с кодом для реализации). На графике можно выделить три основных пика: на 28 остатке, на 349 и 637. Пики отображают линкеры между доменами, поэтому можно “разделить” структуру по этим пикам.
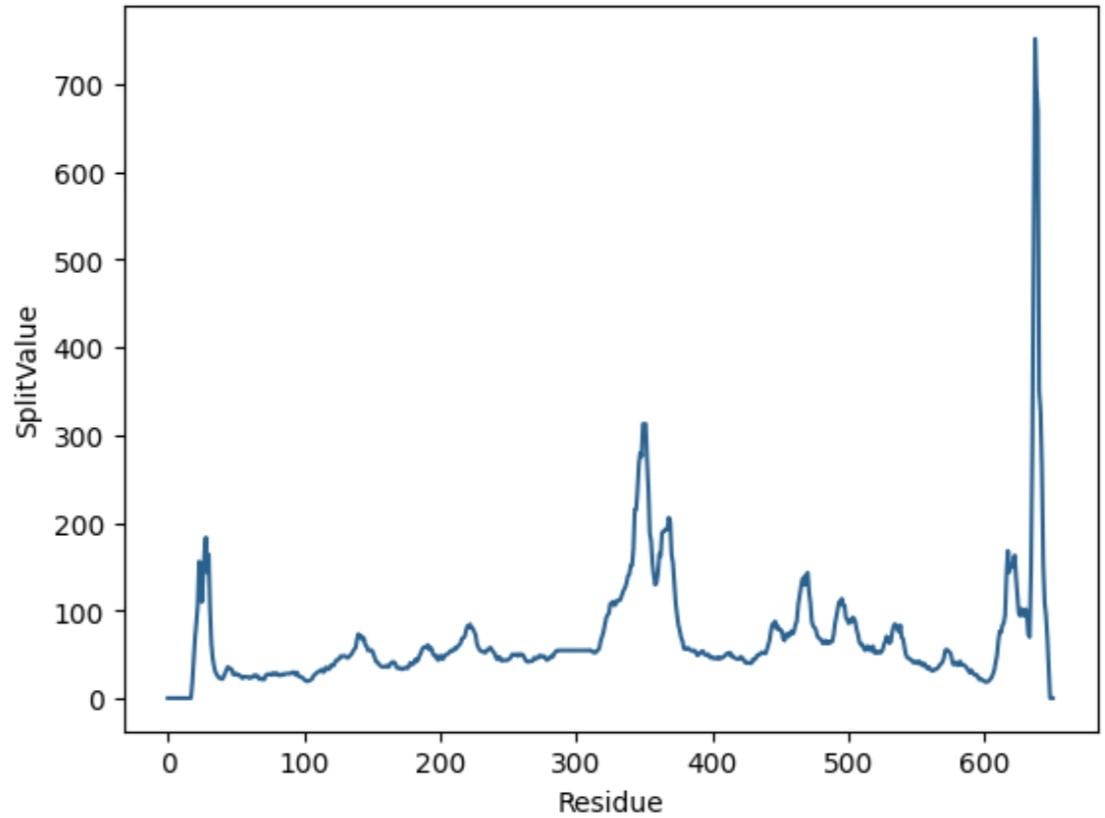
В структуре есть проблемы с нумерацией: в PDB говорится о 644 остатках, тогда как на деле их меньше, и отсчет идет с 17 по 651. Соответственно, если судить по пикам, то на концах последовательности остаются небольшие, длиной в 10 нуклеотидов последовательности, которые якобы отделены от домена, но, кажется, их лучше отнести к одному домену (это небольшие альфа-спирали). Но пока оставим их отдельно. Итого было предсказано два домена + два кусочка по краям (рис.2).
Задание 2
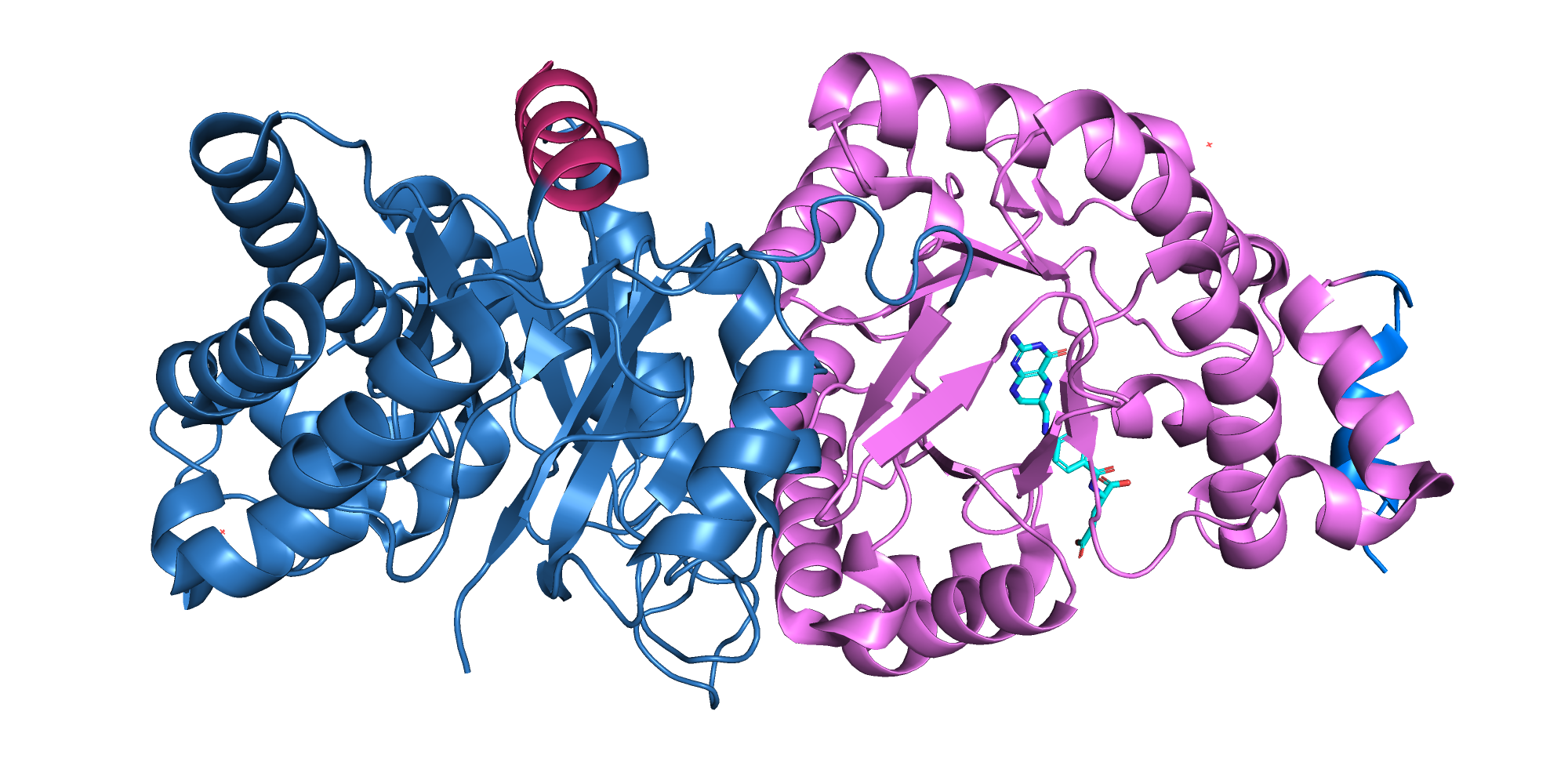
Далее было предложено рассмотреть домены структуры в базах данных SCOP и CATH. В SCOP данная структура не была найдена. В CATH содержатся данные о том, что структура (TIM-бочонок) содержит два домена:
• 17-354 - гомоцистеин связывающий домен
• 355-651 - дигидроптероат синтаза
Разбиение находится примерно в тех же остатках (между доменами длинный линкер, в предсказании ровно посередине линкера, в CATH - перед бета-листом другого домена), а последовательности на краях отнесены к основным доменам (рис.3).
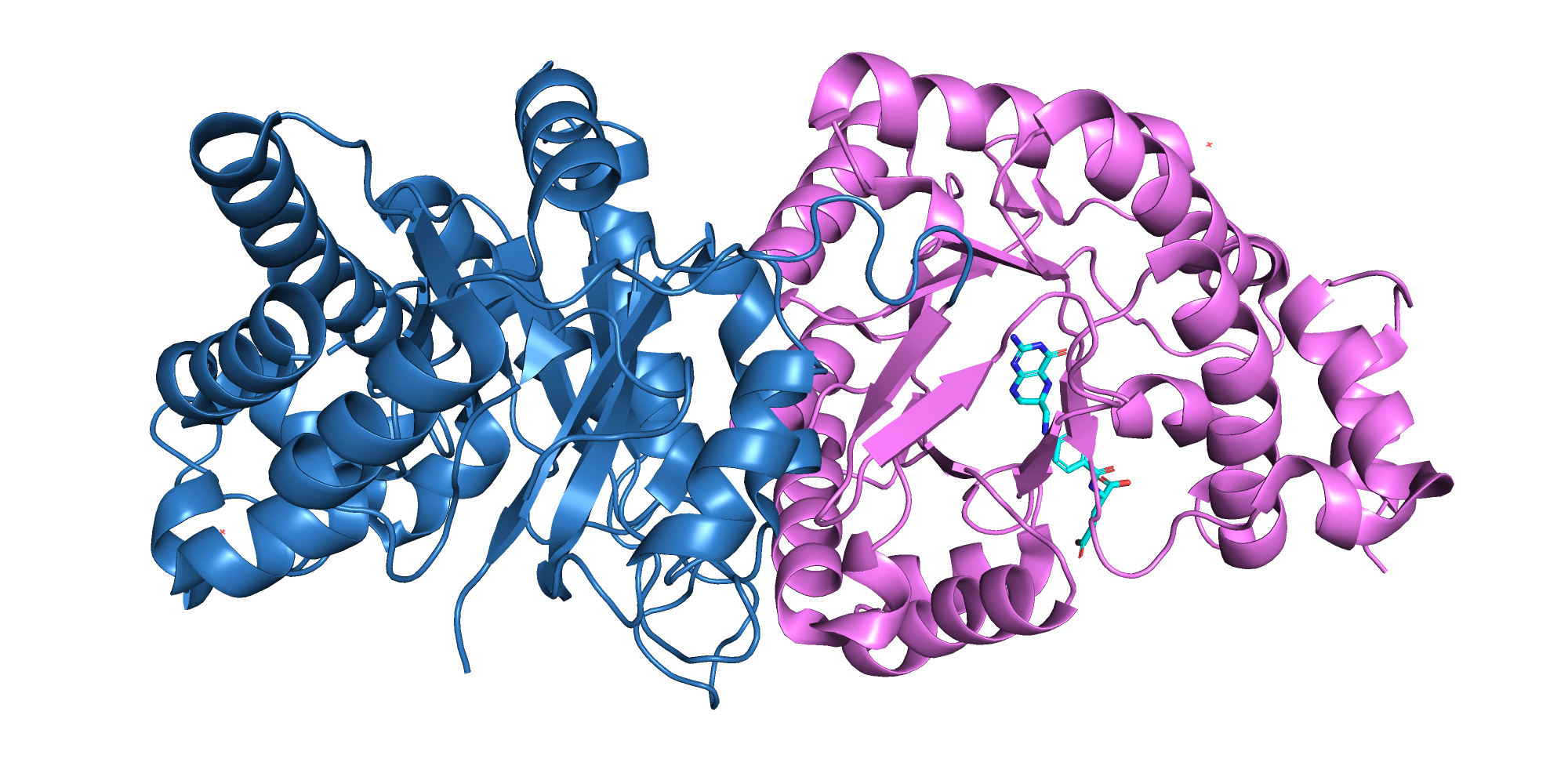
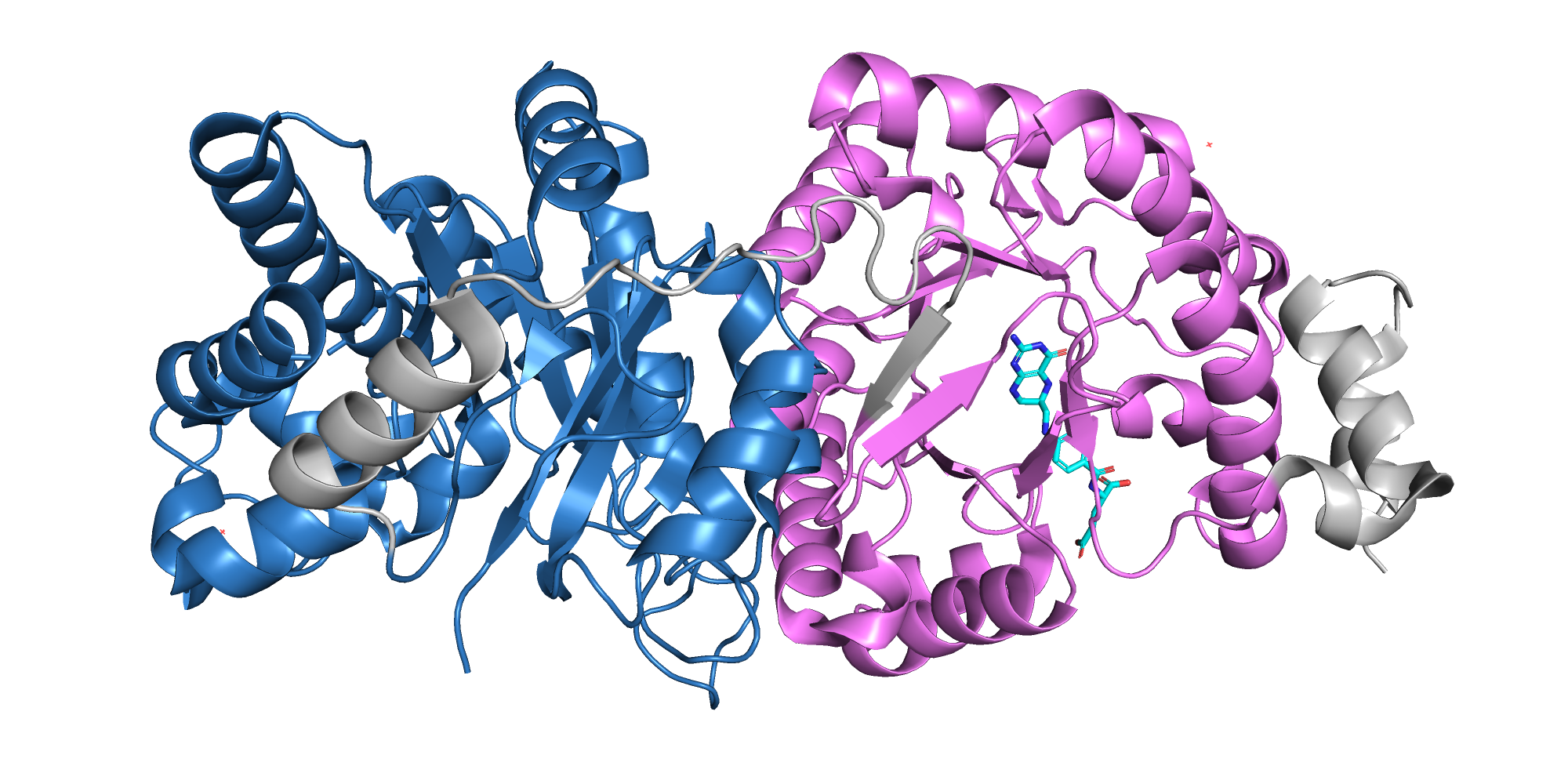
Еще структура была найдена в InterPro по PDB. Там тоже говорится о двух доменах, но их границы немного другие (рис.4):
• 6-325 - гомоцистеин связывающий домен
• 358-619 - птерин связывающий домен
Некоторые несоответствия (рис.5):
• между доменами линкерная часть и даже как будто бы уже части доменов (одна альфа-спираль и один бета-лист) не относятся ни к одному из них.
• в конце последовательности 2.5 альфа-спирали не относятся к домену, хотя, казалось бы, почему бы и нет
Задание 3
Далее в базе эволюционных доменов InterPro производился поиск по последовательности. Были найдены те же домены, что и в предыдущем задании в обеих базах данных (рис.6). Разбиение такое же, как и в найденной записи в InterPro.
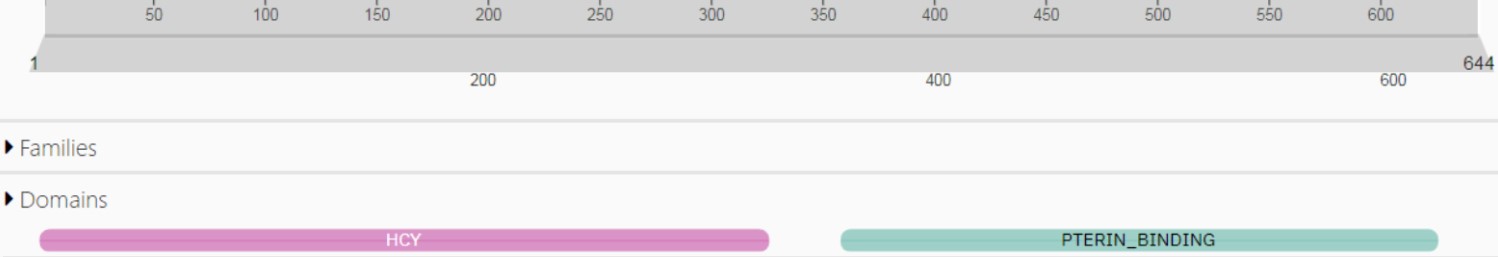
Найденные домены:
• HCY (Homocysteine-binding domain profile, PS50970) - этот домен есть во всех белках, которые переносят метильную группу с различных соединений на гомоцистеин.
• PTERIN_BINDING (Pterin-binding domain profile, PS50972) - домен есть в тех белках, где в реакции участвует производное птерина (как в метионинсинтазе, так и в некоторых других белках).
Функция белка - перенос метильной группы 5-метилтетрагиброфолата на гомоцистеин, образуя метионин. Поэтому все домены в структуре несут те функции, что и должны (связывание нужных соединений для реакции…)
Задание 4
Далее было предложено воспользоваться сервисами DSSP и STRIDE для сравнения сгенерированных аннотаций вторичной структуры. Выдача STRIDE показана на рисунке 7, выдачу DSSP, к сожалению, никак не получилось визуализировать, выдача программы лежит в файле.

В целом, оба сервиса примерно так же определяют укладку последовательности белка, в основном различия в границах типов укладки (плюс-минус пара аминокислот), однако несколько отличий были найдены.
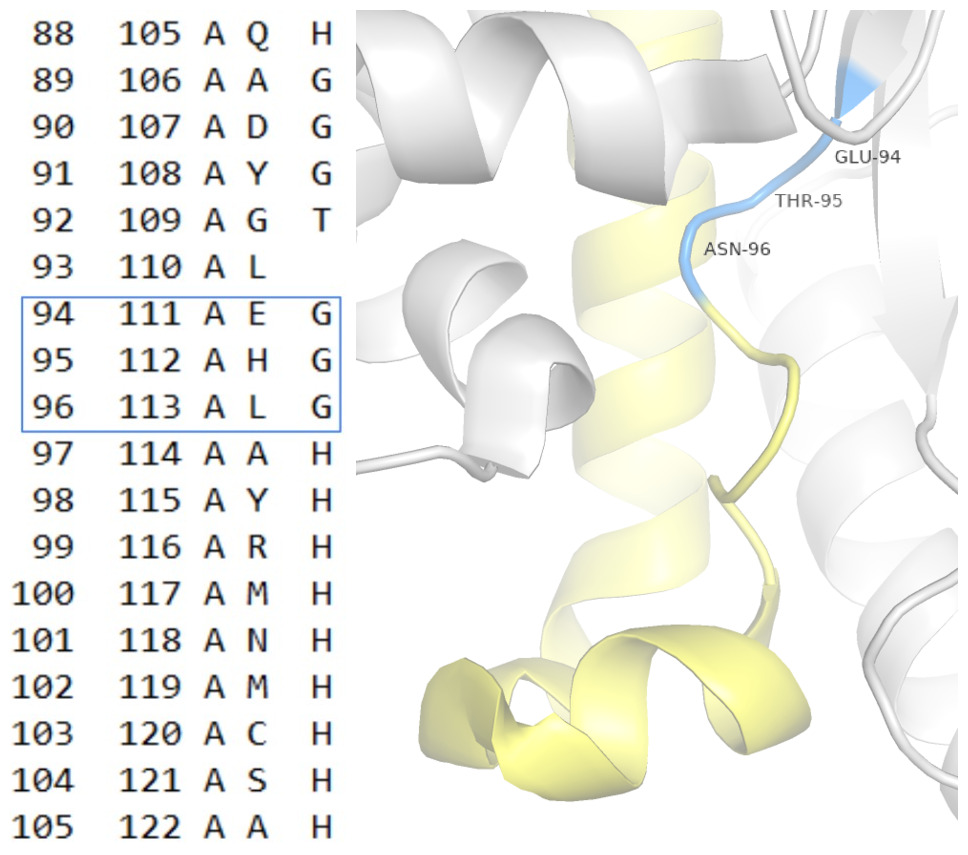
Позиции 94-96: по STRIDE – это поворот и начало альфа-спирали (в синем квадрате), в то время как в DSSP – 310-спираль, сразу переходящая в альфа-спираль (рис. 8). Посмотрев структуры в PyMOL, можно сказать, что эти три остатка точно не образуют никакую спираль, также есть сомнения насчет последующих остатков. Их положение слишком нестабильное для образования спирали. Здесь обе выдачи неверные.
Позиции 211-218: согласно DSSP, здесь нет никакой четкой укладки (рис. 9), а по STRIDE – два бета-листа (розовый квадрат). Структура в PyMOL в данном участке точно не образует (и не сможет) никаких бета-листов, скорее права выдача DSSP, где некоторые остатки указаны как поворот.
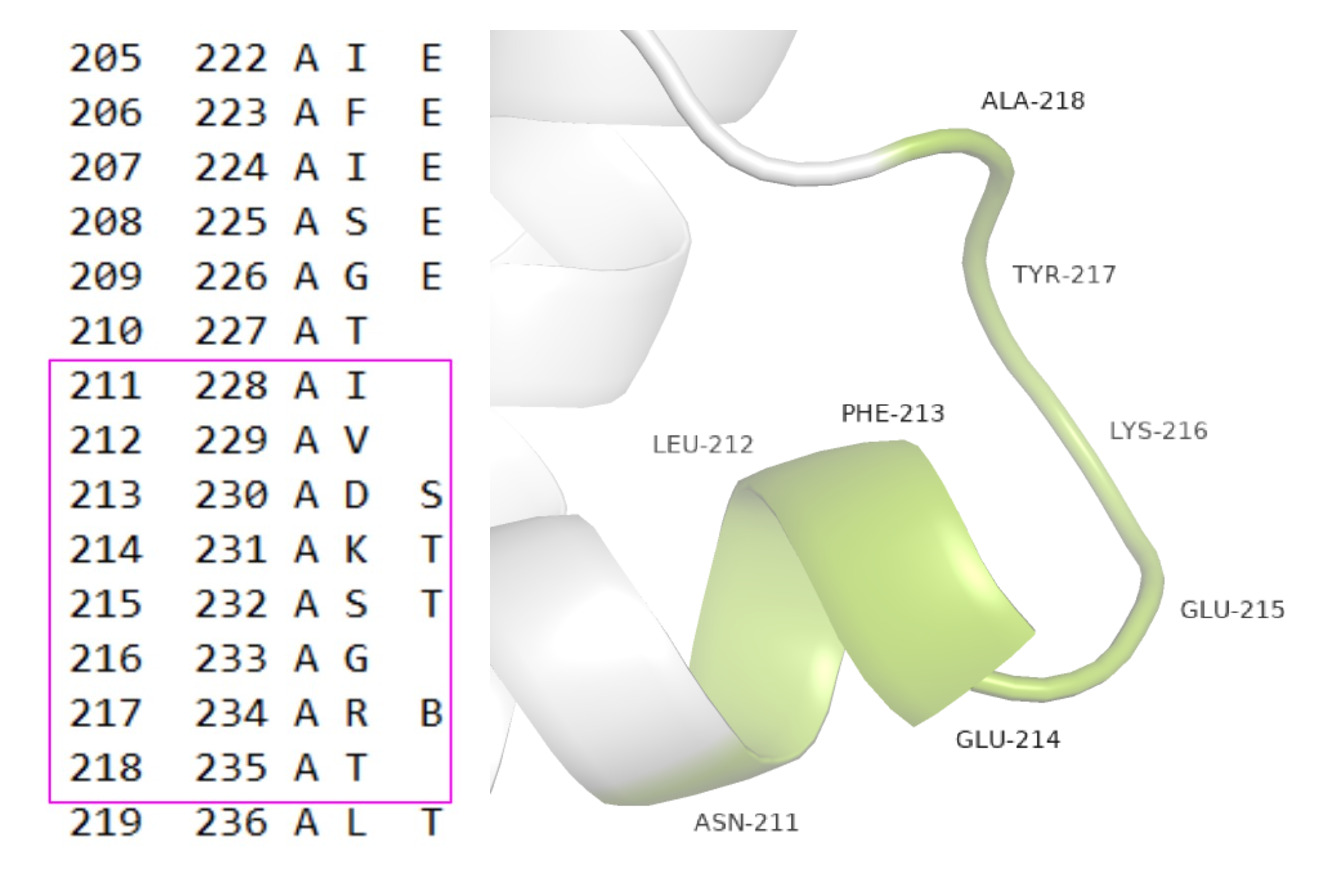
Таким образом, сложно сказать, какой сервис лучше аннотирует вторичную структуру. В данном случае более реалистично предсказал, наверное, DSSP, потому что STRIDE в обоих случаях выдал крайне малвероятные укладки...