Галоархеи - на данный момент относительно малоизученная группа организмов. Тем не менее, её представители обладают рядом интересных особенностей. И потому целью исследования, которому посвящена эта статья, стало изучить некоторые особенности генома одного из представителей галоархей - Halobaculum salinum. В данной статье описаны методы и результаты исследований функционального состава генов, GC-состава CDS, некоторые особенности TATA-бокса и длины закодированных в геноме археи белков.
Для изучаемого организма установлено следующее систематическое положение (данные взяты с сайта NCBI Taxonomy Browser):
Суперцарство: Archaea
Царство: Methanobacteriati
Филум: Methanobacteriota
Клада: Stenosarchaea group
Класс: Halobacteria
Порядок: Halobacteriales
Семейство: Haloferacaceae
Род: Halorarum
Вид: Halorarum salinum
Первоначальное название вида (базионим) - Halobaculum salinum.
Halobaculum salinum - грам-отрицательная, галофильная (минимальная концентрация соли в среде для предотвращения лизиса клеток - 4% (вес/объем)), нейтрофильная (оптимальный для роста pH - 7.0, рост возможен и при значениях pH в пределах от 5.5 до 8.5), термотолерантная (оптимальная для роста температура - 40°C, клетки выживают при значениях температуры в пределах от 25 до 55°C) архея. Способна к анаэробному росту. Клетки подвижные, палочковидные. Были выделены из соленых почв Таримской долины (Китай)[1].По данным таблицы особенностей генома Halobaculum salinum (см. таблицу Т2 сопроводительных материалов) с помощью электронных таблиц отдельно для хромосомы и плазмиды “unnamed1” были рассчитаны:
1. количество генов, кодирующих белки;
2. количество псевдогенов;
3. количество РНК, выполняющих разные функции.
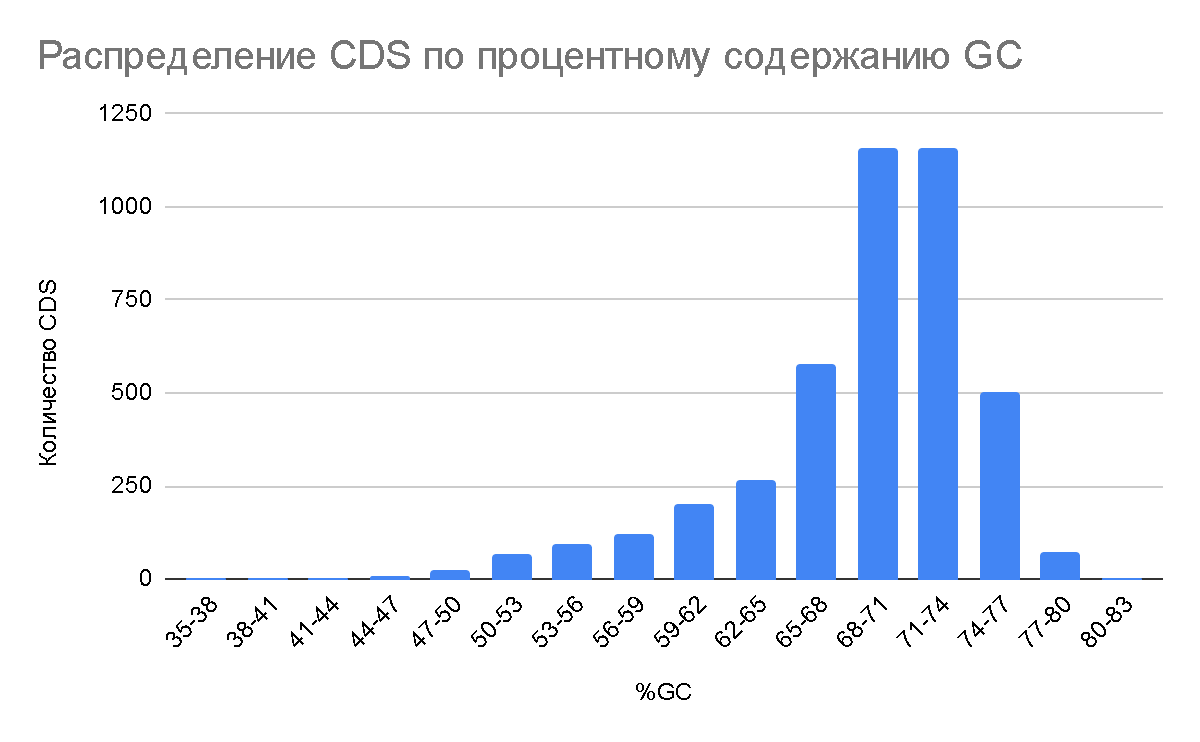
В ходе исследования были использованы данные из таблицы свойств последовательностей (см. таблицу Т1 сопроводительных материалов). С помощью электронных таблиц данные о GC-составе были соотнесены с соответствующими CDS (по их идентификаторам). Затем были построены гистограммы распределения CDS по содержанию в них GC (шаг кармана равен 3)(рис. 1) и расположения их в геноме (рис. 2). Были определены значения, в пределах которых варьируется GC-состав CDS генома, а также среднее по всем значениям GC-состава CDS (таблица 1).
| Минимальный GC% | Максимальный GC% | Средний GC% по всем CDS |
| 36,14 | 81,73 | 68,78 |
Гистограмма, иллюстрирующая GC состав CDS Halobaculum salinum и их расположение на хромосоме позволила определить в геноме археи 4 области, в которых расположены CDS с GC-составом значительно ниже среднего. Эти области были дополнительно исследованы с применением программ BLASTn и BLASTx на предмет наличия последовательностей, схожих с участками геномов других организмов. При выполнении операций в BLAST были установлены параметры ‘по умолчанию’.
Для изучения нуклеотидного состава TATA-боксов в геноме Halobaculum salinum были написаны коды на языке программирования Python (см. коды TATA1 и TATA2 сопроводительных материалов). Первый позволил вывести последовательности 50 нуклеотидов, предшествующих каждой CDS (учитывались только CDS, отделенные от других не менее чем на 50 нуклеотидов, т.к. 50 нуклеотидов - минимальное расстояние между оперонами) (координаты старта CDS взяты из таблицы особенностей генома Halobaculum salinum, см. таблицу Т2 сопроводительных материалов), и записать их в файл 'final.txt'. Каждая новая строка соответствует следующей CDS. В 89,68% из полученных последовательностей присутствовали фрагменты “AT” и “TA”. По этим динуклеотидам осуществлялся последующий поиск TATA-боксов.
Полученный в результате работы первой программы файл передавался на вход второй программе, которая, в свою очередь, в каждой строке искала координату co первого входа динуклеотида, указанного пользователем (на участке от -40-го до -20-го НТ от старта CDS. Такие координаты были выбраны, т.к. это наиболее вероятное место расположения TATA-бокса у архей[2][3]). При чём отдельно были изучены участки, на которых:
1. есть динуклеотид ‘AT’, но отсутствует ‘TA’,
2. есть ‘TA’, но отсутствует ‘AT’,
3. есть и ‘TA’, и ‘AT’, расчет по координатам относительно ‘AT‘,
4. есть и ‘TA’, и ‘AT’, расчет по координатам относительно ‘TA‘.
Эта программа рассчитывала количество последовательностей из файла 'final.txt', соответствующих заданным условиям, частоту нахождения нуклеотидов на позициях от co - 5 до co + 5 и среднее значение координаты co относительно координаты старта CDS. Полученные значения были записаны в электронную таблицу (см. таблицу Т3 сопроводительных материалов).
В ходе исследования были использованы данные из таблицы свойств последовательностей (см. таблицу Т1 сопроводительных материалов). С помощью электронных таблиц по значениям длин CDS были рассчитаны длины соответствующих им белков. По полученным данным была составлена гистограмма длин белков (шаг кармана равен 70; количество белков длины более 1280 аминокислотных остатков незначительно, поэтому белки соответствующей длины объединены в один карман)(рис.7).
| Хромосома | Плазмида “unnamed1” | ||
|---|---|---|---|
| Категория | Количество | Категория | Количество |
| Белок-кодирующие гены | 3984 | Белок-кодирующие гены | 220 |
| Псевдогены | 58 | Псевдогены | 3 |
| РНК РНКазы P | 1 | РНК РНКазы P | 0 |
| рРНК | 3 | рРНК | 0 |
| СР РНК | 1 | СР РНК | 0 |
| тРНК | 48 | тРНК | 0 |
Построена таблица (таблица 2), содержащая информацию о количестве белок-кодирующих генов, псевдогенов, и разных РНК (РНК рибонуклеазы P, рибосомальные РНК, сигнал-распознающая РНК, транспортные РНК).

По гистограмме распределения CDS по содержанию в них GC видно, что в геноме археи преобладают CDS, GC-состав которых лежит в области 68-74%. Такое высокое значение содержания GC обеспечивает термотолерантность археи: пары G-C в ДНК обладают большей устойчивостью, чем пары A-T, т.к. образуют три водородные связи (а не две, как пары A-T), и потому среди организмов, обитающих в экстремальных условиях, наблюдается тенденция к увеличению процента содержания GC в геноме[4].
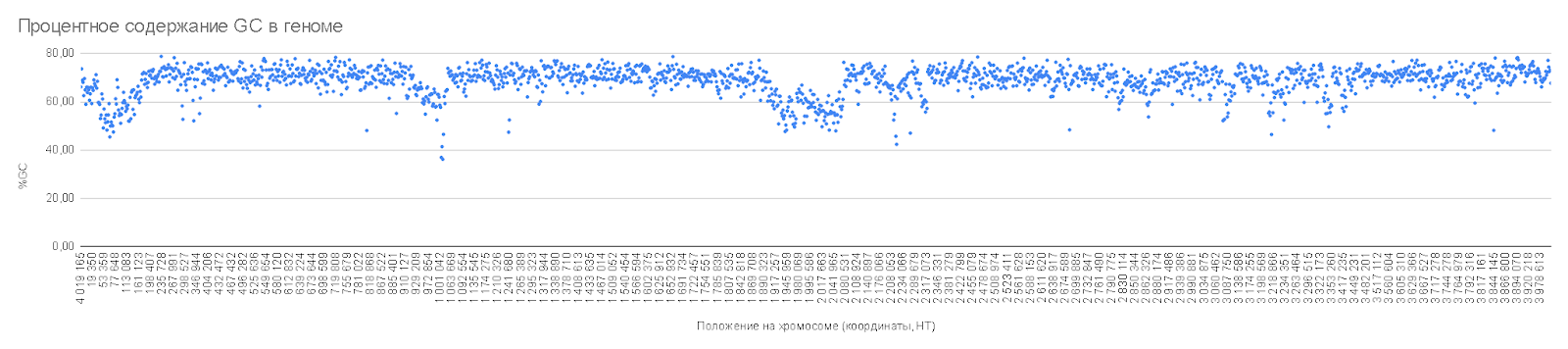
Также по результатам исследования GC состава генома бактерии была составлена гистограмма, иллюстрирующая расположение CDS на хромосоме и их GC-состав (рис.2). С помощью сайта NCBI Blast были исследованы участки с CDS с низким относительно среднего (менее 60%) содержанием GC: 45000-150000 НТ, 965000-1009000 НТ, 1944000-2044000 НТ, 3082000-3132000 НТ. По результатам BLAST’а данных фрагментов были выделены участки, найденные в плазмидах родственных Halobaculum salinum архей:
1. 76132-79604 НТ - найден в плазмидах Halorientalis marina, Halobaculum magnesiiphilum. Включает 4 CDS (таблица 3).
| Координата начала - координата конца CDS, НТ |
Белок, закодированный в данной CDS | GC% |
|---|---|---|
| 76146 - 76571 | ДНК-связывающий белок из семейства Tfx |
52,35 |
| 76634 - 77647 | Внеклеточный белок, связывающий растворенные вещества |
55,03 |
| 77648 - 78463 | Пермеаза ABC-транспортера |
61,27 |
| 78460 - 79515 | ATP-связывающий белок ABC-транспортера | 59,09 |
2. 133996-138436 НТ - найден в плазмидах Haloprofundus salinisoli, Haladaptatus halobius. Содержит 3 CDS (таблица 4).
| 134026 - 134772 | Белок семейства YqcI/YcgG |
51,00 |
|---|---|---|
| 134798 - 135655 | Аденилил-сульфат киназа |
57,46 |
| 135899 - 137122 | Белок, содержащий сульфотрансферазный домен |
57,19 |
3. 983974-985740 НТ - найден в плазмидах Halobacterium noricense, Haloarcula sp. В этом участке не отмечены CDS, но есть одна CDS, лежащая на 983741-985813 НТ и кодирующая трансферазу (содержание GC - 63,92%). Интересно, что похожая последовательность в плазмидах кодирует другой белок: белок семейства VirB4 системы секреции. Это может означать, что данный участок действительно был получен исследуемой бактерией от одной из таких плазмид, но впоследствии в нём произошли мутации, приведшие к изменению функции закодированного белка.
4. 986465-987788 НТ - найден в плазмидах Natrinema sp., Haloterrigena turkmenica. 1 CDS (таблица 5).
| 986580-987695 | Аланин рацемаза | 60,84 |
5. 1967824-1970771, 1972507-1975861 НТ - схожие последовательности обнаружены в плазмидах Halorussus lipolyticus, Haloferax volcanii. Содержит 4 CDS, включая одну, лежащую в промежутке между указанными фрагментами (таблица 6).
| 1967825-1968373 | Фактор транскрипции | 49,54 |
|---|---|---|
| 1968385-1968924 | ТАТА-box-связывающ ий белок |
49,26 |
| 1969143-1972505* | N-6 ДНК-метилаза (относится к системе рестрикции-модифик ации) |
51,09 |
| 1972498-1975860 | Относящийся к хеликазе белок |
52,93 |
*стоит заметить, что в указанных выше плазмидах есть последовательность, кодирующая тот-же по функции белок, но отличающийся по аминокислотному составу от белка, закодированного в геноме исследуемой археи. Это может быть связано с повышенной специфичностью белков системы рестрикции-модификации[6].
6. 3087643-3088168 НТ. Схожие последовательности найдены в плазмидах Halomicrobium urmianum, Haloplanus rubicundus. Включает 1 CDS (таблица 7).
| 3087750-3088082 | Гипотетический белок |
53,75 |
Итак, для указанных последовательностей были найдены схожие в плазмидах архей, родственных Halobaculum salinum, кодирующие те же или схожие белки. GC% CDS, лежащих в этих последовательностях, значительно ниже среднего (68,74%). При этом не было найдено плазмид, принадлежащих виду Halobaculum и содержащих эти последовательности. На основании этих данных можно сделать вывод, что данные последовательности были приобретены H. salinum или её предком в результате горизонтального переноса от родственных архей. Стоит отметить, что некоторые из этих последовательностей встречаются и в хромосомах некоторых архей.
Исследование участка 3082000-3132000 НТ позволило обнаружить в геноме H. salinum фрагменты генома вируса Halorubrum virus Humcor1, паразитирующего на археях Halorubrum, относящихся к тому же семейству, что и исследуемая архея. Они расположены в области 3108540-3117271 НТ(рис.3). Было проведено выравнивание последовательностей генома вируса и соответствующего участка генома бактерии (таблица 8).
| Перекрывание, % | Идентичность, % | Счёт выравнивания (в скобках - нормированный), биты | Гэпы |
|---|---|---|---|
| 86 | 75 | 1716 (929) | 262/4510 |
На данном участке генома изучаемой археи расположено 9 CDS, кодирующих гипотетические белки, 1 CDS, кодирующая бактериоцин, 1 CDS, кодирующая портальный белок фага. На соответствующем участке генома вируса лежат CDS, кодирующие различные белки капсида (главный белок капсида, протеаза головки, морфогены головки, портальный белок, большая протеаза). Кроме того в геноме археи присутствует CDS, кодирующая белок хвоста этого же вируса. Как показало исследование этой CDS, кодируемый ею белок сильно отличается от вирусного (наибольшее сходство - 50.86% при 69% перекрывании; E-value = 1e-104). При чем схожая последовательность была обнаружена у других представителей порядка Halobacteriales: Halorubrum trueperi, Haloparvum sedimenti, Haloferax volcanii, Natrinema sp.

По полученным данным можно сделать предположение, что Halorubrum virus Humcor1 инфицировал общего предка указанных бактерий (или же заражение произошло независимо для разных групп, что менее вероятно), но в связи с неприспособленностью бороться с его системами защиты (системой рестрикции-модификации и т.д.) не смог на нём паразитировать. В результате работы защитных механизмов археи принадлежащие вирусу гены подверглись различным мутациям, что привело к утрате или изменению функций закодированных в них белков. В результате, в геноме археи остались измененные, вероятнее всего, нефункциональные участки генома фага.
На основании полученных в результате исследования данных были построены четыре гистограммы частот нахождения нуклеотидов в заданных участках (см. п. 3.3)(рис.4).
На гистограммах 3 и 4 можно заметить некоторое соответствие между группами столбцов для разных координат (рис.5)(обнаруживается и соответствие между некоторыми группами гистограмм 1 и 2, но оно слишком незначительно, чтобы делать какие-либо выводы). Это может указывать на идентичность соответствующих им координат относительно начала CDS, т.к. для этих двух условий подходит одно и тоже множество последовательностей, предшествующих CDS. Такое соответствие не случайно: оно может быть результатом нахождения в этом участке TATA-бокса (динуклеотиды ТА, и АТ, которые по заданным условиям одновременно находятся в интервале от -40 до -20 нуклеотида (от начала CDS), могут перекрываться, что и отразилось на гистограммах как некоторый “сдвиг значений” ). На этом основании этого было решено рассчитать среднее от значений результатов исследований 3 и 4 (среднее рассчитывалось от соответствующих значений). По полученным данным была построена гистограмма, иллюстрирующая предполагаемый нуклеотидный состав области вокруг TATA-бокса (рис.6).
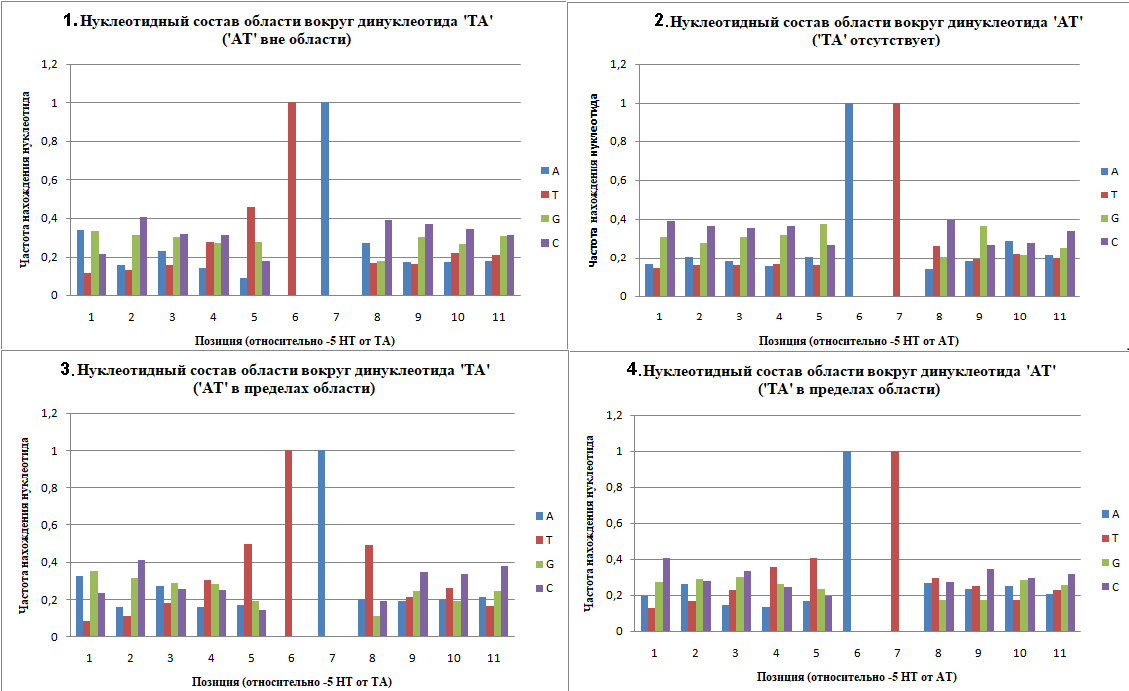
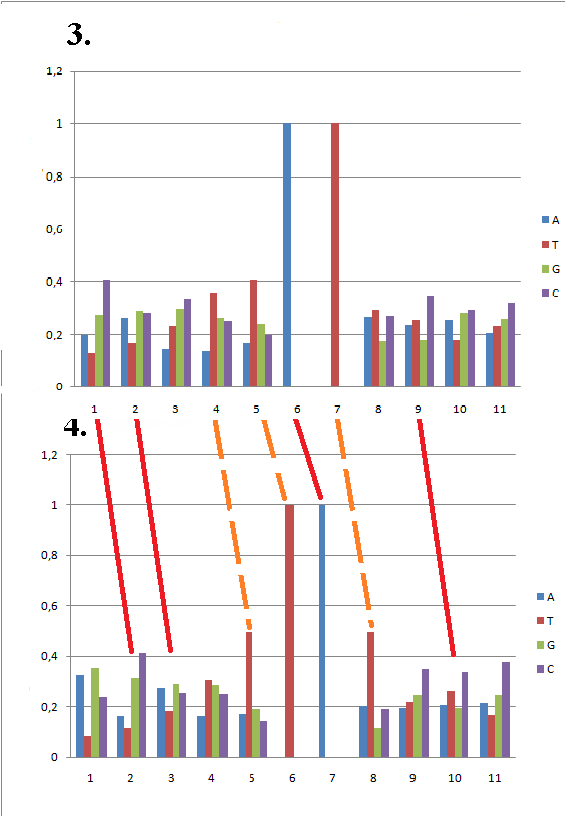
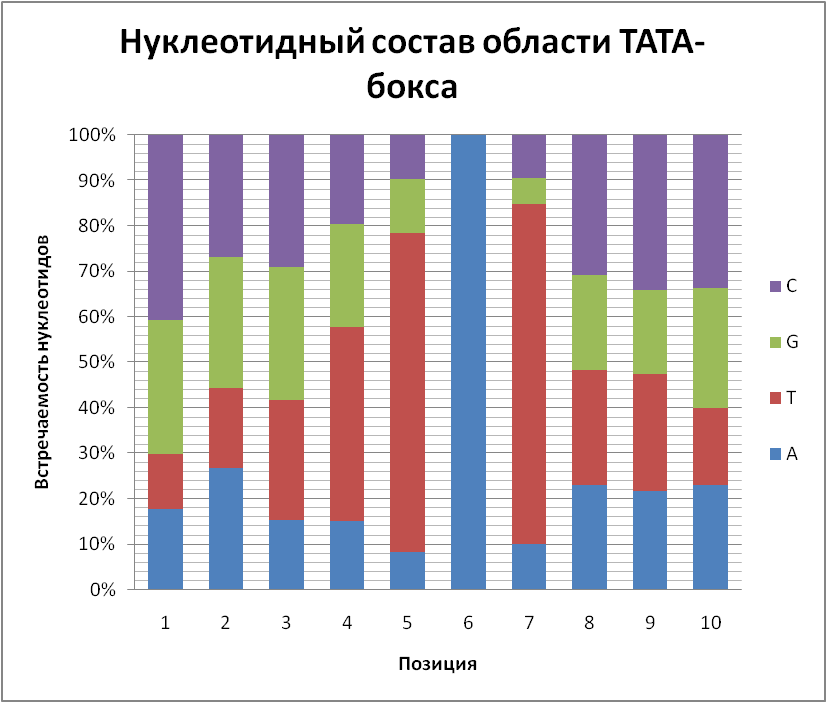
По полученной гистограмме можно сделать вывод, что чаще всего в ТАТА-боксе Halobaculum salinum встречается элемент ‘TAT’. Сам ТАТА-бокс окружён GC-богатыми участками, но встречаемость G и C в изученных позициях ниже, чем в среднем по геному, что не противоречит функции TATA-бокса.
Стоит отметить, что у архей TATA-бокс гораздо менее консервативен, чем у эукариот[2]. Именно поэтому появилась идея исследовать последовательности, предшествующие оперонам. Полученный результат показывает часть вариантов структур TATA-бокса, встречающегося у H. salinum.
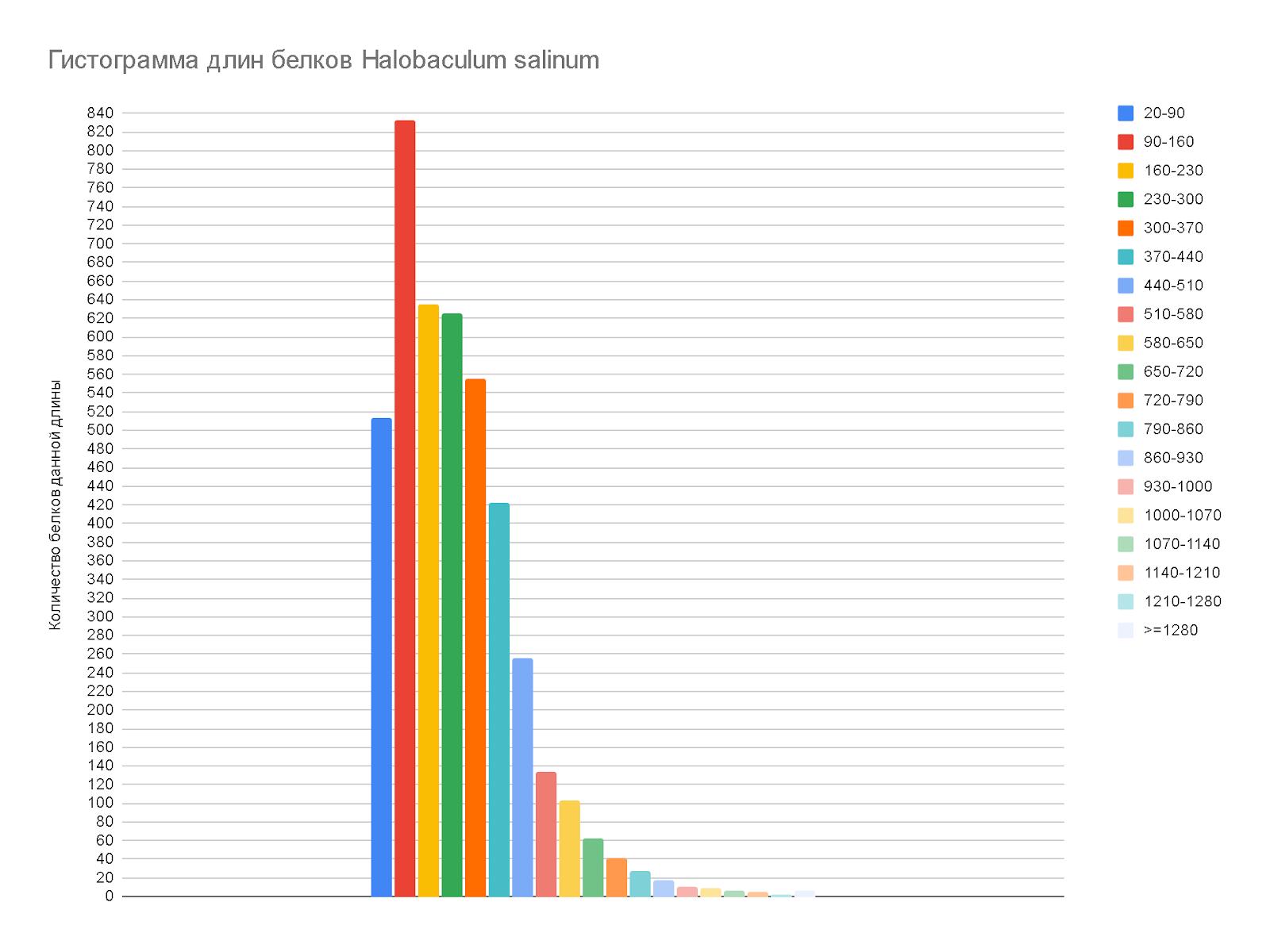
По результатам анализа данных генома археи была создана гистограмма, иллюстрирующая распределение закодированных в нем белков по длине (рис.7). По ней можно сделать выводы о том, что длина большей части белков археи варьируется в пределах от 90 до 370 аминокислот - 2648 из 4265 закодированных в геноме (62,09 %), что в целом соответствует средней длине белков архей[5]. Лишь 29 белков (0,68 %) имеют длину более 1000 аминокислот.
В ходе исследования были изучены некоторые особенности генома археи Halobaculum salinum. Были получены следующие результаты:
● Установлено количество псевдогенов и генов, кодирующих белки РНК отдельно для хромосомы и плазмиды;
● Исследован GC-состав CDS археи. Построены гистограммы распределения CDS по содержанию в них GC и расположения их в геноме. Построенные гистограммы показали участки с особенностями GC-состава, что послужило поводом более подробно исследовать эти участки. В результате были найдены последовательности, вероятно, приобретенные путем горизонтального переноса генов от других архей, и фрагмент генома вируса, вероятно, ставший нефункциональным в результате действия систем защиты археи;
● Установлен возможный нуклеотидный состав области ТАТА-бокса;
● Построена гистограмма длин белков, закодированных в геноме Halobaculum salinum
1.Т1 - Таблица свойств последовательностей генома Halobaculum salinum. Содержит данные о длинах CDS, GC-составе CDS. Была дополнена данными о длинах продуктов CDS, координатами начала каждой CDS и гистограммами, описанными и представленными ранее.
2.T2 - Таблица особенностей генома Halobaculum salinum. Из этой таблицы были взяты все данные о координатах CDS, кодируемых ими продуктах и др. Дополнена другими таблицами для проведения описанных в статье исследований.
3. T3 - Таблицы результатов исследования нуклеотидного состава области около ТАТА-бокса. Содержат выводы программы TATA2 и представленные в пункте 3.3 гистограммы.
4.TATA1 - на вход даётся название файла, содержащего нуклеотидную последовательность генома организма. В одной директории с программой должен находиться файл 'start.txt', содержащий координаты начала CDS, расположенных на ‘+’ цепи ДНК (эти данные можно взять из таблицы особенностей генома соответствующего организма). Вывод программы - файл 'final.txt', содержащий в каждой строке 50 нуклеотидов, предшествующих CDS.
5.TATA2 - программа на вход берёт файл 'final.txt' из той же директории, где расположена она. При запуске программы нужно выбрать режим её работы: если мы хотим проверить последовательности с участками, в которых есть одновременно 2 типа динуклеотидов, то нужно выбрать режим ‘&’. Если же нас интересуют такие участки, что в них есть один динуклеотид, но нет другого, то нужно выбрать режим ‘/’. Затем нужно ввести динуклеотид, в радиусе 5 нуклеотидов от которого будет рассчитан процентный нуклеотидный состав последовательности. В следующей строке вводим динуклеотид, который должен присутствовать/отсутствовать в изучаемом участке. На выход программа дает количество исследованных последовательностей, процент содержания разных нуклеотидов на позициях в радиусе 5 нуклеотидов от первого найденного динуклеотида, заданного условием, и среднее положение этого динуклеотида относительно старта CDS.
Примечание: программа ищет заданные динуклеотиды на участке от -40-го до -20-го нуклеотида относительно старта CDS.
1. Cui HL, Shi XW, Yin XM, Yang XY, Hou J, Zhu L. Halobaculum halophilum sp. nov. and Halobaculum salinum sp. nov., isolated from salt lake and saline soil. Int J Syst Evol Microbiol. 2021 Jul;71(7). doi: 10.1099/ijsem.0.004900. PMID: 34283016.
2. Martinez GS, Sarkar S, Kumar A, Pérez-Rueda E, de Avila E Silva S. Characterization of promoters in archaeal genomes based on DNA structural parameters. Microbiologyopen. 2021 Oct;10(5):e1230. doi: 10.1002/mbo3.1230. PMID: 34713600; PMCID: PMC8553660.
3. Hausner W, Frey G, Thomm M. Control regions of an archaeal gene. A TATA box and an initiator element promote cell-free transcription of the tRNA(Val) gene of Methanococcus vannielii. J Mol Biol. 1991 Dec 5;222(3):495-508. doi: 10.1016/0022-2836(91)90492-o. PMID: 1748992.
4. Hu, EZ., Lan, XR., Liu, ZL. et al. A positive correlation between GC content and growth temperature in prokaryotes. BMC Genomics 23, 110 (2022). doi:10.1186/s12864-022-08353-7
5. Tiessen A, Pérez-Rodríguez P, Delaye-Arredondo LJ. Mathematical modeling and comparison of protein size distribution in different plant, animal, fungal and microbial species reveals a negative correlation between protein size and protein number, thus providing insight into the evolution of proteomes. BMC Res Notes. 2012 Feb 1;5:85. doi: 10.1186/1756-0500-5-85. PMID: 22296664; PMCID: PMC3296660.
6. Janulaitis A, Kazlauskiene R, Lazareviciute L, Gilvonauskaite R, et al. Taxonomic specificity of restriction-modification enzymes. Gene. 1988 Dec 25;74(1):229-32. doi: 10.1016/0378-1119(88)90293-4. PMID: 3074011.