Данный домен входит в структуру белков семейства MutS - белков, участвующих в репарации mismatch-мутаций в ДНК. В это семейство входят в том числе эукариотические белки MSH 1, 2, 3, 4, 5, 6. Они играют различные роли в репарации и рекомбинации ДНК. Для человеческого MSH показана связь с предрасположенностью к раку толстой кишки. Домен MutS I участвует в распознавании мутации и связывании с ДНК и инициации процесса репарации.
Наиболее распространённая архитектура белков, включающих домен MutS I - пять доменов: собственно MutS I, MutS II (распознаёт структуры ДНК, близкие к структуре Холидея), MutS III (центральный домен), MutS IV (зажим, способствующий продвижению комплекса по ДНК), MutS V (ATPазный домен, содержащий мотив Walker A).
Белки, включающие данный домен, представлены у 25491 вида, из них 19843 вида - бактерии, 4880 - эукариоты, 679 - археи, 70 - вирусы. Также домен обнаружен в белках 19 организмов неясного систематического положения.
Более подробная информация представлена в таблице 1.
| Информация | |
|---|---|
| AC pfam | PF01624 |
| ID pfam | MutS domain I |
| #SEED | 50 |
| #All | 54k |
| #SW | 652 |
| #architectures | 437 |
| #3D | 53 |
| Taxonomy | 53666 |
| #eukaryota | 16728 |
| #archaea | 1559 |
| #bacteria | 34793 |
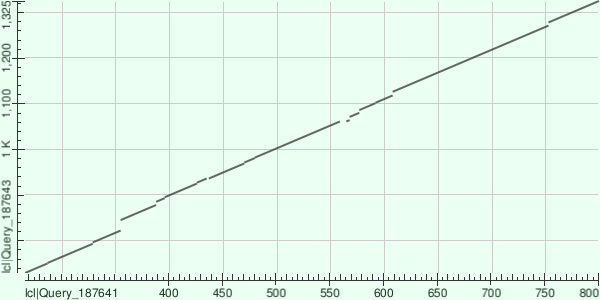
Для построения карты локального сходства были выбраны 2 белка с различной архитектурой: MutS кишечной палочки (Uniprot AC: P23909) и Msh мыши (Uniprot AC: Q8C2N9). Карта локального сходства (см. рис.1) показывает сходства между участками ~0-160, ~260-800 P23909 и ~400-580, ~720-1325 Q8C2N9 соответственно. Участок ~580-720 белка мыши не перекрывается ни с одним участком белка E. coli, как и участок ~160-260 белка кишечной палочки не перекрывается ни с одним участком белка мыши. После сравнения полученных данных со схемами архитектур (см. рис. 2, 3) можно сделать вывод о том, что соответствующий этим участкам фрагмент домена MutS III довольно сильно изменился в процессе эволюции (домен MutS III, представленный в белке мыши, длиннее, чем соответствующий домен белка кишечной палочки, то есть в нём, скорее всего, появились новые участки, которым нельзя найти соответствий в белке E. coli). Участок ~0-400 белка мыши не выравнивается с белком кишечной палочки. В этой области у белка мыши представлен домен PWWP, отсутствующий у бактериального белка и позволяющий белку мыши связываться с метилированным по K20 гистоном 4. Также при проведении выравнивания с пониженным порогом E-value (1*10^-11) на карте локального сходства пропадает участок, соответствующий доменам MutS I и MutS II (см. рис.4), что говорит о том, что данный участок довольно сильно различается у изученных белков.

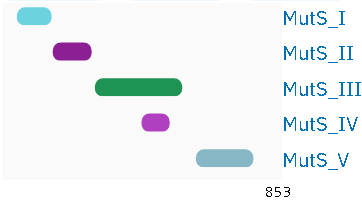
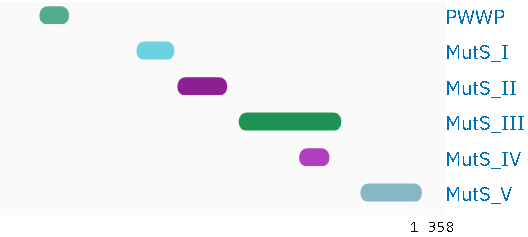
| Информация | AC | |
|---|---|---|
| Доменная архитектура 1 | MutS_I - MutS_II - MutS_III - MutS_IV - MutS_V | PF01624 - PF05188 - PF05192 - PF05190 - PF00488 |
| Белок с архитектурой 1 | DNA mismatch repair protein MutS | P23909 |
| Доменная архитектура 2 | PWWP - MutS_I - MutS_II - MutS_III - MutS_IV - MutS_V | PF00855 - PF01624 - PF05188 - PF05192 - PF05190 - PF00488 |
| Белок с архитектурой 2 | DNA mismatch repair protein | Q8C2N9 |
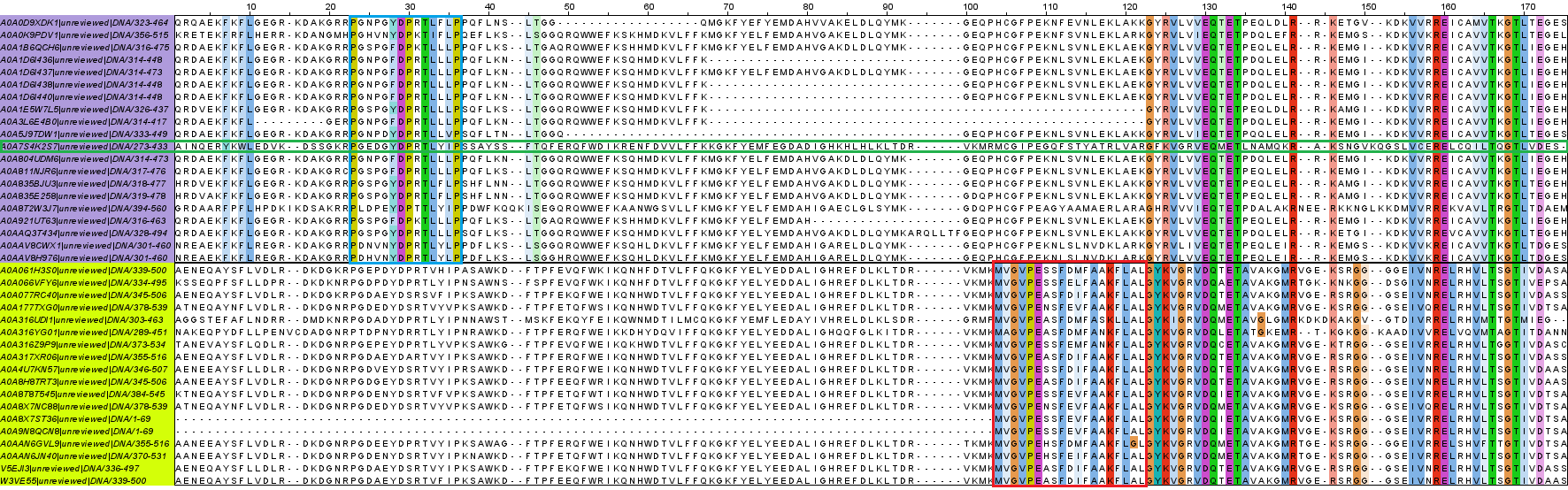
Для проведения исследования были выбраны белки с архитектурой PTM_DIR17_Tudor - MutS_I - MutS_II - MutS_III - MutS_IV - MutS_V (20 последовательностей, архитектура 1) и с архитектурой MutS_I - MutS_II - MutS_III - MutS_IV - MutS_V - zf-C2H2_3 - Acetyltransf_13 (18 последовательностей, архитектура 2) (см. рис.5).

Проект Jalview - первая (верхняя) группа соответствует белкам с архитектурой 1, вторая (нижняя) - белкам с архитектурой 2.
После проведения выравнивания был обнаружен достоверный блок, который можно выделить только у группы домена белков с архитектурой 2. Это столбцы 104-122 (см. рис.6, выделено красным). Многие из столбцов другой группы достоверно отличаются от них, например:
Столбец 104 - в 17 из 20 последовательностей домена из белков с архитектурой 1 - H, в домене белков с архитектурой 2 - M;
105 - в 18 из 20 последовательностей первой группы - C, во второй группе - V;
107 - в 17 из 20 последовательностей первой группы - F, во второй группе - V;
110 - в 16 из 20 последовательностей первой группы - K, во второй группе выделить преобладающий остаток нельзя;
111 - в 16 из 20 последовательностей первой группы - N, во второй группе - S;
115 - в 16 из 20 последовательностей первой группы - N, во второй группе - F;
119 - в 18 из 20 последовательностей первой группы - L, во второй группе - F в 19 из 20 последовательностей;
120 - в 17 из 20 последовательностей первой группы - A, во второй группе - L;
122 - в 17 из 20 последовательностей первой группы - [KR], во второй группе - L.
При окраске выравнивания по Clustal также становится заметен блок 23-36 у последовательностей первой группы (см. рис.6, выделено синим), но если присмотреться ко второй группе, то можно заметить, что если не учитывать индели на этих позициях в двух строках, то окажется, что многие из указанных столбцов характерны и для второй группы
Достоверно отличающиеся столбцы можно заметить и в других участках. Так в столбце 127 у 19 из 20 последовательностей первой группы - L, а у всех второй - G; в 128 столбце у 19 из 20 последовательностей первой группы - [VI], у второй - R; в 135 столбце у 19 из 20 последовательностей первой группы - P, у второй - A; в 125 и 130 столбцах все остатки функционально консервативны, но каждой группе соответствует свой остаток.
Основываясь на этих наблюдениях можно сделать вывод о том, что домены MutS I в своей эволюции разошлись прежде чем войти в состав белков с изученной архитектурой, на что указывает наличие в выравнивании как столбцов, характерных для обеих групп, так и столбцов, характерных лишь для одной из них.
Кроме того можно заметить, что одна из строк первой группы - A0A7S4K2S7|unreviewed|DNA, 11 строка (см. рис.6, выделено зелёным) - содержит большое число позиций (в том числе столбцы 104, 112, 118, 127), отличающихся от соответствующих позиций других последовательностей этой группы, но идентичных последовательностям второй группы. Эта последовательность может быть переходным вариантом в эволюции домена. Возможно, организм, белок которого содержит эту последовательность, таксономически более близок организмам, белки которых составляют вторую группу.