Задача: провести сборку генома de novo по данным секвенирования бактерии Buchnera aphidicola str. Tuc7.
Для выполнения поставленной задачи были выбраны короткие одноконцевые прочтения, полученные с помощью технологии Illumina. Код доступа чтений в базе данных ENA: SRR4240356. Общее число чтений: 7511529.
Данные чтения содержат последовательности адаптеров. Чтобы избавиться от них, все известные последовательности адаптеров были записаны в файл adapters.fa. Затем была применена программа Trimmomatic для одноконцевых чтений:
В результате выполнения данной операции было удалено 153091 прочтений (2.04% от всех чтений).
Затем нужно было очистить последовательности от позиций плохого качества и удалить слишком короткие прочтения. Для этого была использована следующая команда:
То есть с правых концов чтений были удалены нуклеотиды с качеством меньше 20, а также были удалены чтения, короче 32 нуклеотидов. В результате было удалено 305092 (4.15%) чтений. Если до 'чистки' плохих чтений файл с ними весил 0.72 Гб, то после неё - 0.69 Гб. Полученные послеедовательности чтений уже можно использовать для сборки генома.
Для сборки генома была выбрана программа velvet, осуществляющая алгоритм, использующий граф де Брёйна. Поэтому первым делом следует подготовить k-меры. Это было сделано с помощью следующей команды:
Применённые опции:
-fastq &mdash формат файла с чтениями;
-short &mdash были использованы короткие одноконцевые чтения;
Assem &mdash название директории с результатами выполнения команды;
31 &mdash k-меры какой длины нужно получить.
После того, как данная команда была выполнена и k-меры были подготовлены, можно запускать сам алгоритм сборки:
В результате была создана сборка, состоящая из 82 контигов и имеющая N50 равный 65554 при общей длине сборки 659837, что является неплохим показателем. Было определено три самых длинных контига:
>NODE_8_length_111962_cov_38.660198
>NODE_6_length_107488_cov_34.174030
>NODE_10_length_80939_cov_37.524174
После тэга 'length' указана длина контига, после 'cov' - покрытие (можно понимать как то, среднее значение того, сколько раз встретился каждый k-мер из контига в чтениях). Таким образом, самые длинные контиги имеют длины 111962, 107488 и 80939 (при чём это длина, указанная 'в контигах', т.е. их длины в нуклеотидах на 30 больше).
Была рассчитана медиана покрытий контигов. Она равна 16.64. Собственно, аномально высокое значение покрытия (>83,2) имеют следующие 8 контигов:
>NODE_27_length_282_cov_458.429077
>NODE_17_length_950_cov_447.494751
>NODE_14_length_934_cov_444.608124
>NODE_29_length_308_cov_436.269470
>NODE_2_length_524_cov_419.526703
>NODE_20_length_501_cov_309.155701
>NODE_55_length_42_cov_211.428574
>NODE_36_length_45_cov_166.199997
Такие контиги могут быть связаны с повторами в геноме. Аномально низкое же покрытие (<3.3) имеют 2 контига:
>NODE_74_length_31_cov_3.064516
>NODE_123_length_91_cov_2.362637
Взглянув на них, можно заметить, что они действительно отличаются от остальных, например, в них практически отсутствуют повторы T. Такие контиги не несут практически никакой полезной информации, могут быть связаны с засорением пробы и т.д.
Все полученные контиги можно посмотреть здесь.
Сравним 3 самых длинных контига с референсным геномом Buchnera aphidicola (штамм Sg) (код доступа Refseq: GCF_000007365.1). Для этого были проведены выравнивания соответствующих последовательностей с помощью megablast.
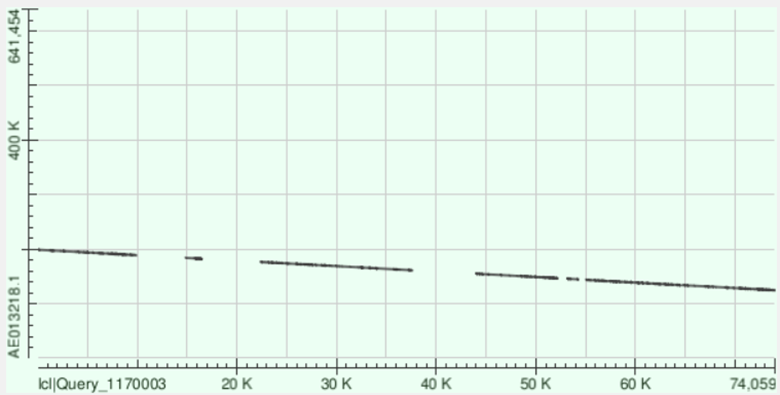
Контиг 10: выравнялся на обратную цепь хромосомы на координатах 124592 - 198923 (см. рис.1, таблица 1).
| № | % identity | alignment length | mismatches | gap opens | q. start | q. end | s. start | s. end |
|---|---|---|---|---|---|---|---|---|
| 1 | 79.574 | 11456 | 2102 | 192 | 22381 | 33728 | 176511 | 165186 |
| 2 | 78.960 | 9957 | 1823 | 217 | 73 | 9899 | 198923 | 189109 |
| 3 | 79.186 | 8259 | 1529 | 148 | 44003 | 52168 | 155000 | 146839 |
| 4 | 76.304 | 9626 | 1947 | 258 | 55035 | 64475 | 143634 | 134158 |
| 5 | 83.522 | 3708 | 550 | 44 | 33915 | 37593 | 164960 | 161285 |
| 6 | 82.980 | 3208 | 497 | 37 | 64594 | 67784 | 134086 | 130911 |
| 7 | 75.529 | 4867 | 1005 | 147 | 69297 | 74059 | 129376 | 124592 |
| 8 | 84.906 | 1378 | 177 | 24 | 67916 | 69275 | 130830 | 129466 |
| 9 | 79.380 | 1644 | 288 | 46 | 14864 | 16478 | 184028 | 182407 |
| 10 | 78.030 | 1188 | 244 | 17 | 53176 | 54353 | 145787 | 144607 |
Контиг 6: выравнялся на прямую цепь хромосомы на координатах 224649 - 329761 (см. рис.2, таблица 2).
| № | % identity | alignment length | mismatches | gap opens | q. start | q. end | s. start | s. end |
|---|---|---|---|---|---|---|---|---|
| 1 | 83.468 | 5450 | 731 | 133 | 51319 | 56687 | 275325 | 280685 |
| 2 | 78.073 | 7443 | 1447 | 139 | 77558 | 84912 | 300247 | 307592 |
| 3 | 76.223 | 8399 | 1826 | 147 | 16322 | 24649 | 240921 | 249219 |
| 4 | 79.908 | 5460 | 991 | 84 | 102 | 5506 | 224649 | 230057 |
| 5 | 77.382 | 6424 | 1216 | 179 | 57455 | 63770 | 281375 | 287669 |
| 6 | 78.549 | 5184 | 974 | 104 | 45990 | 51108 | 270046 | 275156 |
| 7 | 80.170 | 4241 | 732 | 92 | 28384 | 32565 | 252821 | 257011 |
| 8 | 78.069 | 4432 | 853 | 87 | 91727 | 96076 | 314359 | 318753 |
| 9 | 77.210 | 4660 | 954 | 85 | 11648 | 16251 | 236289 | 240896 |
| 10 | 78.229 | 3964 | 770 | 82 | 39827 | 43745 | 264126 | 268041 |
| 11 | 82.851 | 2525 | 400 | 23 | 8978 | 11491 | 233479 | 235981 |
| 12 | 83.645 | 2299 | 356 | 18 | 24681 | 26969 | 249302 | 251590 |
| 13 | 77.841 | 3493 | 667 | 85 | 67433 | 70870 | 290753 | 294193 |
| 14 | 82.632 | 2234 | 370 | 18 | 96696 | 98919 | 319264 | 321489 |
| 15 | 75.288 | 4257 | 914 | 115 | 103039 | 107231 | 325579 | 329761 |
| 16 | 78.009 | 2592 | 472 | 75 | 71691 | 74232 | 294987 | 297530 |
| 17 | 81.729 | 1828 | 292 | 30 | 5693 | 7500 | 230176 | 231981 |
| 18 | 76.698 | 2635 | 519 | 79 | 86913 | 89509 | 309620 | 312197 |
| 19 | 81.965 | 1547 | 264 | 14 | 75706 | 77242 | 298415 | 299956 |
| 20 | 80.143 | 1395 | 257 | 20 | 65795 | 67179 | 289161 | 290545 |
| 21 | 79.644 | 1125 | 221 | 8 | 63986 | 65105 | 287911 | 289032 |
| 22 | 84.628 | 592 | 81 | 5 | 74265 | 74848 | 297640 | 298229 |
| 23 | 77.904 | 792 | 155 | 17 | 33553 | 34335 | 258005 | 258785 |
Контиг 8: самый длинный из полученных. Выравнялся на прямую цепь хромосомы на координатах 458393 - 567722 (см. рис.3, таблица 3)
| № | % identity | alignment length | mismatches | gap opens | q. start | q. end | s. start | s. end |
|---|---|---|---|---|---|---|---|---|
| 1 | 81.778 | 16134 | 2643 | 218 | 87410 | 103366 | 545069 | 561082 |
| 2 | 79.468 | 12395 | 2254 | 225 | 14620 | 26908 | 472743 | 484952 |
| 3 | 87.689 | 5808 | 524 | 123 | 81581 | 87300 | 539282 | 544986 |
| 4 | 82.278 | 6557 | 1015 | 109 | 103595 | 110099 | 561261 | 567722 |
| 5 | 82.452 | 4519 | 717 | 48 | 63097 | 67589 | 521171 | 525639 |
| 6 | 78.234 | 5729 | 1090 | 117 | 75762 | 81418 | 533534 | 539177 |
| 7 | 79.337 | 4975 | 932 | 78 | 70506 | 75431 | 528532 | 533459 |
| 8 | 78.512 | 4435 | 863 | 75 | 5068 | 9461 | 463184 | 467569 |
| 9 | 77.526 | 4761 | 943 | 98 | 32 | 4732 | 458393 | 463086 |
| 10 | 78.420 | 4203 | 805 | 76 | 47478 | 51658 | 505626 | 509748 |
| 11 | 79.554 | 3546 | 580 | 102 | 57949 | 61400 | 515883 | 519377 |
| 12 | 73.441 | 6913 | 1485 | 282 | 36247 | 43004 | 494400 | 501116 |
| 13 | 75.730 | 4660 | 975 | 121 | 27011 | 31602 | 485189 | 489760 |
| 14 | 79.089 | 2415 | 447 | 49 | 9515 | 11901 | 467567 | 469951 |
| 15 | 79.275 | 2316 | 434 | 28 | 33857 | 36133 | 491916 | 494224 |
| 16 | 75.583 | 3432 | 694 | 112 | 54522 | 57893 | 512526 | 515873 |
| 17 | 81.909 | 1697 | 264 | 32 | 45773 | 47445 | 503857 | 505534 |
| 18 | 80.331 | 1088 | 195 | 15 | 51767 | 52845 | 509809 | 510886 |

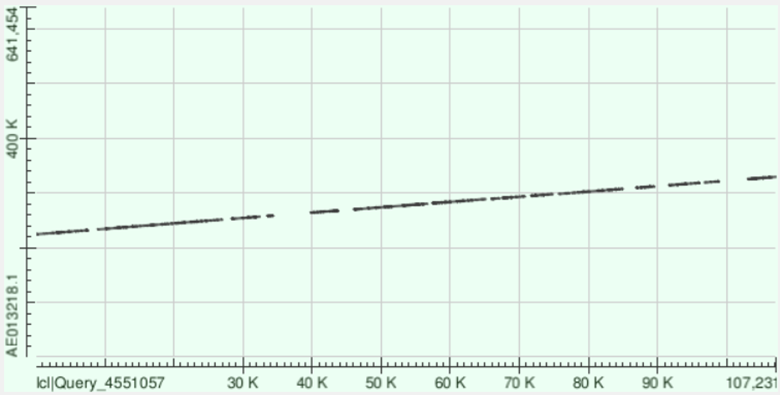
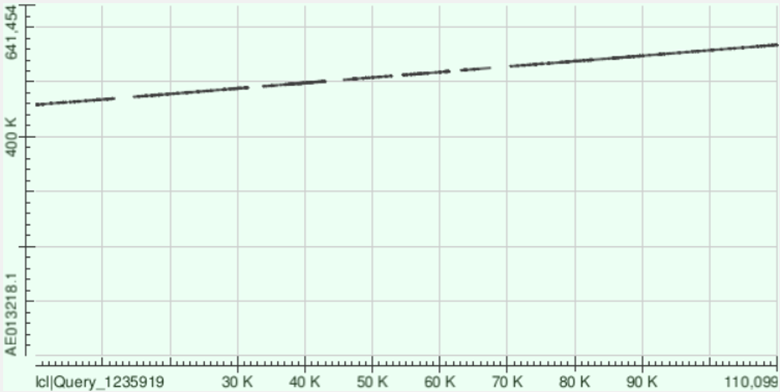
Как видим, все контиги выравниваются на хромосому B. aphidicola str. Sg довольно продолжительными участками. То есть есть длинные очень похожие между двумя последовательностями участки, а между ними - участки, между которыми megablast не находит соответствий (что объясняется тем, что megablast ищет последовательности, почти идентичные заданной). При этом нигде не наблюдается инверсий, делеций или инсерций, что ожидаемо, ведь мы сравниваем последовательности геномов двух организмов одного вида.