Практикум 10. Выравнивание последовательностей белка
Глобальное парное выравнивание гомологичных белков
Для выравнивания использовались белки с одинаковой мнемоникой, присутствующие одновременно в протеоме кишечной и сенной палочек. Выравнивание производилось с помощью программы "needle" из пакета EMBOSS. Результаты выравнивания представлены в таблице 1.
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels |
|---|---|---|---|---|---|---|---|
| ATP-dependent DNA helicase DinG | DING_ECOLI | DING_BACSU | 513.5 | 19.3% | 31.6% | 393 | 25 |
| HTH-type transcriptional repressor PurR | PURR_ECOLI | PURR_BACSU | 40.5 | 15.3% | 32.2% | 120 | 13 |
| Putative xanthine dehydrogenase molybdenum-binding subunit XdhA | XDHA_ECOLI | XDHA_BACSU | 34.0 | 5.5% | 9.8% | 696 | 14 |
Любопытно, что несмотря на гомологию белков, выравнивание показало низкий процент идентичности (<20%). Стоит отметить немалое количество пропусков во всех выравниваниях, особенно в последнем, где число "гэпов" равно 696, а процент идентичности рекордно низок (5.5%).
Локальное парное выравнивание гомологичных белков
Для получения более точных результатов было проведено локальное выравнивание тех же пар белков, с использованием программы "water" из пакета EMBOSS.
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
|---|---|---|---|---|---|---|---|---|---|
| ATP-dependent DNA helicase DinG | DING_ECOLI | DING_BACSU | 523.0 | 25.4% | 41.3% | 155 | 22 | 97.3% | 74.5% |
| HTH-type transcriptional repressor PurR | PURR_ECOLI | PURR_BACSU | 52.0 | 17.5% | 35.4% | 90 | 13 | 82.4% | 86.0% |
| Putative xanthine dehydrogenase molybdenum-binding subunit XdhA | XDHA_ECOLI | XDHA_BACSU | 43.5 | 19.0% | 30.4% | 51 | 5 | 14.5% | 47.3% |
В результате степень идентичности возросла (максимум до 25.4%), но все равно осталась маленькой для гомологичных белков. У первых двух пар количество пропусков уменьшилось примерно вдовое, в то время как у белков с мнемоникой "XDHA" этот показатель упал почти в 7 раз (с 696 до 51). Доля покрытия белка в первых двух случаях оказалась в среднем около 75-98%, в последнем - доля белков из пары весьма неравнозначна. Так, у кишечной палочки процент покрытия белка выравниванием составляет 14.5%, а у сенной - 47.3%.
Результаты третьей пары могут быть обусловлены небольшим количеством белков с такой мнемоникой и недостаточной их изученностью. Всего по запросу "mnemonic:xdha*" в базе данных Uniprot появились 3 записи, 2 из которых относятся к рассматриваемым бактериям, а последний - к другому штамму кишечной палочки (Escherichia coli O157:H7). На странице записи1 в описании указано, что классфикация фермента не была точно установлена, до сих пор неизвестно, относится ли он к дегидрогеназам или же к оксидазам. Все это дает основания предполагать, что несмотря на рецензию и положение в Swiss-Prot белки с мнемоникой "XDHA" недостаточно изучены.
Результат применения программ выравнивания к неродственным белкам
Для сравнения с гомологичными белками также были проведены аналогичные выравнивания случайной пары белков с разной мнемоникой. Результаты находятся в таблице 3 и таблице 4 ниже.
| Protein Name 1 | Protein Name 2 | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels |
|---|---|---|---|---|---|---|---|---|
| tRNA-specific adenosine deaminase | Phosphomethylpyrimidine synthase | TADA_ECOLI | THIC_BACSU | 34.0 | 5.3% | 8.9% | 527 | 10 |
| Protein Name 1 | Protein Name 2 | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
|---|---|---|---|---|---|---|---|---|---|---|
| tRNA-specific adenosine deaminase | Phosphomethylpyrimidine synthase | TADA_ECOLI | THIC_BACSU | 42.0 | 32.0% | 64.0% | 0 | 0 | 15.0% | 4.2% |
Глобальное выравнивание показало самый низкий процент идентичности (5.3%) и второй по величине показатель "гэпов", тем самым демонстрируя различия белков. Поскольку выравнивание производится формально, такие показатели можно считать на уровне погрешности.
В то же время локальное выравнивание показало хоть и до сих пор небольшие показатели схожести (32% идентичности), но для всех пар они являются рекордными (за исключением покрытия, оно достигает минимума в 4.2%). Более того, у этого выравнивания не обнаружено ни одного пропуска, а следовательно, нет и инделей. Вероятно, такое совпадение можно назвать случайным, потому что белки едва ли похожи друг на друга по функции (EC:3.5.4.33 и EC:4.1.99.17).
Множественное выравнивание белков и импорт в Jalview
Всего для мнемоники "PurR" найдено 96 белков (запрос "mnemonic:purr*"), из которых 3 записи устарели, поэтому в итоге остается 93. Для выравнивания преимущественно использовались белки из известных организмов. Их коды доступа: P0ACP7, P37551, O68446, Q9KRC1, Q7CIS2, P46456 и B4EWM9. По данным страницы Uniprot2 белок из кишечной палочки с такой мнемоникой носит имя "HTH-type transcriptional repressor PurR".
Выравнивание проводилось при помощи программы "muscle" из пакета EMBOSS при стандартных настройках. Команду и файл можно просмотреть ниже.
muscle -in purr.fasta -out purr_alignment.fastaПросмотреть fasta файл выравнивания
Обработка производилась в программе Jalview, последовательности были отсортированы по парному выравниванию (pairwise alignment) и покрашены по шаблону "% Identity". Результаты можно просмотреть в виде проекта, изображения с аннотацией или филогенетического дерева.
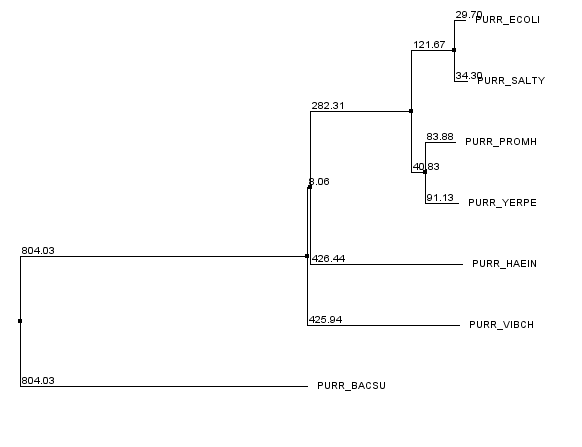
Уже по изображениям можно сделать вывод о больших расхождениях последовательности белка сенной палочки с другими белками. В остальном последовательности похожи между собой: дополнительное вычисление парных выравниваний показывает процент идентичности между остальными последовательностями не менее 50%. Поэтому имеется множество участков с выраженной структурой (например, столбцы с 131 по 142 включительно консервативны и одинаковы у всех 6 белков). В то же время процент идентичности с белком сенной палочки не превосходит 20%, что хотя и примерно соответствует результатам работы needle между потенциально гомологичными белками, но дает основание предполагать, что белок, возможно, не гомологичен остальным.
Сравнение белка Q9K4U1 с его гомологом P12758
Помимо уже упомянутых организмов - кишечной и сенной палочек, было проведено выравнивание с участием белка из холерного вибриона, его код доступа - Q9K4U13. Для более точного построения выравнивания с его гомологом P127584 использовалась программа water с опцией '-auto'.
Результаты представлены в таблице 5. Они в высокой степепени соответстуют гомологичным белкам - все показатели, кроме 1 инделя и гэпа являются максимальными для всех проведенных сравнений. Кроме того, длина покрытия стремится к 100%, а значит такое выравнивание почти повторяет глобальное, и проведение анализа с помощью needle даст те же самые результаты.
| Protein Name 1 | Protein Name 2 | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
|---|---|---|---|---|---|---|---|---|---|---|
| Uridine phosphorylase | Uridine phosphorylase | Q9K4U1_VIBCL | UDP_ECOLI | 1008.0 | 75.1% | 90.1% | 1 | 1 | 99.6% | 100.0% |
Сравнение входных параметров needle и water
Эти параметры охватывают штрафы за открытие и продолжение гэпов. По-умолчанию для программы needle они составдяют 10.0 за первый и 0.5 за каждый последующий гэпы в инделе. (результат скачивания curl происходит из-за расположения белка Q9K4U1 в TrEMBL)
needle uniprot:Q9K4U1 sw:P12758
Needleman-Wunsch global alignment of two sequences
% Total % Received % Xferd Average Speed Time Time Time Current
Dload Upload Total Spent Left Speed
100 17581 0 17581 0 0 1644 0 --:--:-- 0:00:10 --:--:-- 31394
Gap opening penalty [10.0]:
Gap extension penalty [0.5]:
Эти параметры по-умолчанию имеют те же самые значения и у water.
water uniprot:Q9K4U1 sw:P12758
Smith-Waterman local alignment of sequences
% Total % Received % Xferd Average Speed Time Time Time Current
Dload Upload Total Spent Left Speed
100 17581 100 17581 0 0 1718 0 0:00:10 0:00:10 --:--:-- 95032
Gap opening penalty [10.0]:
Gap extension penalty [0.5]:
В качестве свидетельства влияния этих параметров на поиск выравнивания, например, можно отменить афинные штрафы за гэп у одного из уже проведенных (локального, между белками с мнемоникой XDHA).
water sw:xdha_ecoli sw:xdha_bacsu Smith-Waterman local alignment of sequences Gap opening penalty [10.0]: 10 Gap extension penalty [0.5]: 10
Теперь оптимальное выравнивание изменилось, все гэпы исчезли, покрытие уменьшилось, однако проценты идентичности и схожести возросли. Результаты отображены в таблице 6.
| Protein Name 1 | Protein Name 2 | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
|---|---|---|---|---|---|---|---|---|---|---|
| Putative xanthine dehydrogenase molybdenum-binding subunit XdhA | Putative xanthine dehydrogenase molybdenum-binding subunit XdhA | XDHA_ECOLI | XDHA_BACSU | 29.0 | 21.1% | 34.2% | 0 | 0 | 5.1% | 11.5% |