Сборка de novo
Подготовка к работе
Информация о запуске (run) по ссылке.
Команда для скачивания fastq:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/008/SRR4240378/SRR4240378.fastq.gz
Команда для копирования и объединения адаптеров:
cp /mnt/scratch/NGS/adapters/* . | cat *.fa > ad_all.fa
Подготовка чтений программой trimmomatic
Проведена с помощью команды:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 -trimlog trim.log -threads 12 SRR4240378.fastq.gz adm.fastq.gz ILLUMINACLIP:ad_all.fa:2:7:7
Из вывода в stdout команды Trimmomatic можно заключить, что осталось 4338744 (98.15%) чтений:
Input Reads: 4420587 Surviving: 4338744 (98.15%) Dropped: 81843 (1.85%)
Отсечение с конца нуклотидов с качеством менее 20 и чтений длиной менее 32 произвдено командой:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 -trimlog trim2.log -threads 12 adm.fastq.gz trimed.fastq.gz ILLUMINACLIP:ad_all.fa:2:7:7 MINLEN:32 TRAILING:20
Из выввода:
Input Reads: 4338744 Surviving: 4279176 (98.63%) Dropped: 59568 (1.37%)
Разница между input reads в первом использовании и surviving во втором составляет 141411 удаленных чтений. Оценка размеров файлов произведена с помощью команды "ls":
ls -lh
Итого: SRR4240378.fastq.gz (изначальный файл) - 91 МБ, adm.fastq.gz - 89 МБ (файл без адаптеров) и trimed.fastq.gz - 87 МБ (фильтрованный).
Подготовка k-меров посредством velveth
Для k=31, коротких непарных чтений (short) и формата fastq комнада выглядит как:
velveth velveth 31 -fastq -short trimed.fastq.gz
Построение графа де-Брейна
Осуществлено командной:
velvetg velveth
Из файла Log получено количество верщин графа - 361 и N50 - 7028.
Среднее покрытие посчитано, как общее покрытие контигов, поделенное на их количество, оно составляет 467.877:
awk '{total += $6} END {print total/361}' stats.txt
467.877
Аномально большие и маленькие по длине контиги были найдены с помощью команд:
sort -k 2 -n stats.txt | tail sort -k 2 -n stats.txt | head
Самые длинные:
- Длина:16745 , покрытие:20.90
- Длина:19371 , покрытие:20.55
- Длина:36746 , покрытие:20.02
Самые короткие (несколько с длиной 1):
- Длина:1 , покрытие:148886
- Длина:1 , покрытие:606
- Длина:1 , покрытие:617
Анализ длинных контигов с хромосомой GenBank/EMBL AC — CP009253 с помощью megablast
Самые длинные контиги с номерами 8, 57, 15. Все последовательности контигов получены с помощью команды:
seqretsplit contigs.fa -auto
В результате выравниваний были получены некоторые характеристики, они представлены в таблице 1.
| Номер контига | Координаты хромосомы | Total score | %Identity | Гэпы в лучшем выравнивании | E-value |
|---|---|---|---|---|---|
| 8 | 480874-516539 | 13826 | 75.62% | 351/8617(4%) | 0 |
| 57 | 573092-587055 | 4532 | 73.43% | 461/9822(4%) | 0 |
| 15 | 144368-151796 | 4423 | 77.80% | 243/7536(3%) | 0 |
Для просмотра наложения были рассмотрены карты локального сходства:
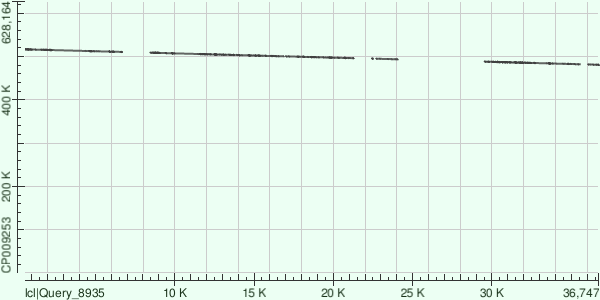
Восьмой контиг состоит из 7 участков. Расположены линейно, слегка убывают, имеются суммарные индели примерно на 8000 нуклеотидов.
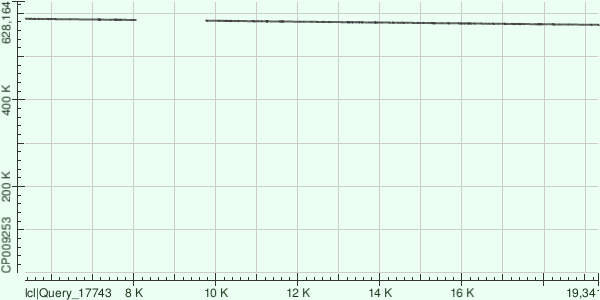
57-ой контиг состоит из 2 участков. Расположены линейно, слегка убывают, имеются суммарные индели примерно на 1500 нуклеотидов.
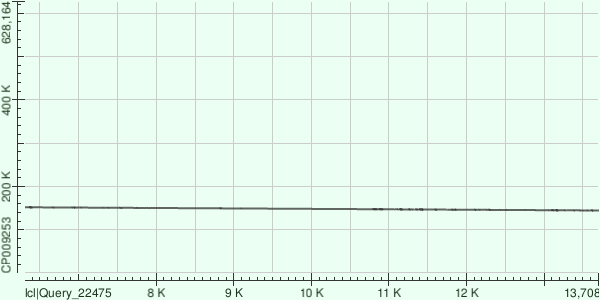
15-ой контиг состоит из 1 участка. Линейный, слегка убывает.