Филогенетические деревья
Филогенетическое дерево бактерий
Для построения дерева были выбраны 7 видов бактерий, видовые наименования которых соотнесены с компактными мнемоническими именами в таблице ниже.
| Видовое название | Мнемоническое название | ||
|---|---|---|---|
| Yersinia pestis | YERPE | ||
| Escherichia coli | ECOLI | ||
| Haemophilus influenzae | HAEIN | ||
| Pasteurella multocida | PASMU | ||
| Pseudomonas mendocina | PSEMY | ||
| Neisseria meningitidis | NEIMA | ||
| Polynucleobacter asymbioticus | POLAQ | ||
Филогенетическое дерево имеет скобочную структуру:
((((YERPE, ECOLI), (HAEIN, PASMU)), PSEMY), (NEIMA, POLAQ))
Оно представлено на рисунке 1.
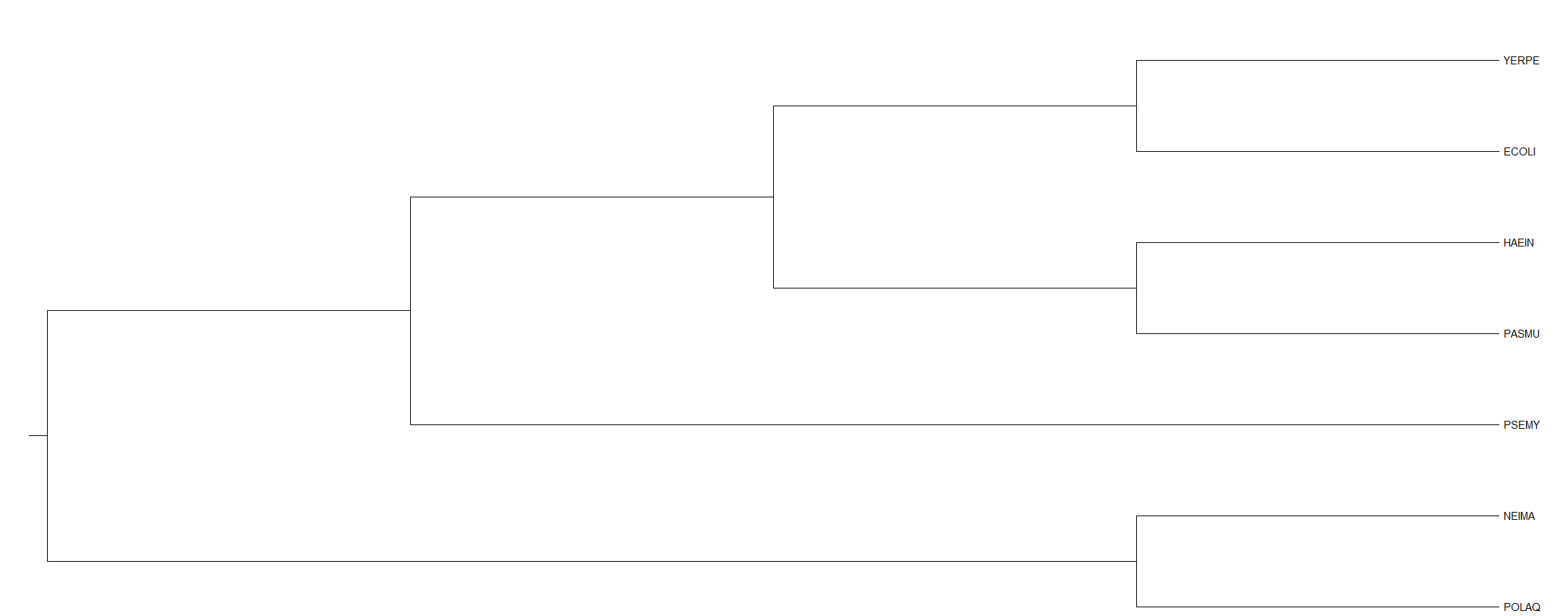
Тривиальные ветви образуют следующие разбиения:
- {YERPE, ECOLI} против {HAEIN, PASMU, PSEMY, NEIMA, POLAQ} соответствует Enterobacterales
- {HAEIN, PASMU} против {YERPE, ECOLI, PSEMY, NEIMA, POLAQ} соответствует Pasteurellaceae
- {YERPE, ECOLI, HAEIN, PASMU} против {PSEMY, NEIMA, POLAQ} соответствует Gammaproteobacteria
- {HAEIN, PASMU,YERPE, ECOLI, PSEMY} против { NEIMA, POLAQ} соответствует Betaproteobacteria
Реконструкция филогении
Таксоны, соответствующие тривиальным ветвям представлены в списке тривиальных ветвей из предыдущего задания. Их поиск осуществлялся с помощью сервиса NCBI taxonomy.
Для реконструкции дерева была выбрана энолаза из всех рассмтариваемых в первом задании видов. Ее мнемоника - ENO. Последовательности белков были скачены с сайта NCBI, помещены на kodomo и выровнен с помощью Muscle, изменив названия на мнемонику бактерий. Команда:
muscle -in list.fasta -out al.fasta
Полученнле выравнивание было открыто в MEGA X, на его основанни были построены деревья: максимального правдоподобия, максимальной экономии и UGPMA.
Укоренение, бутстрэп и дерево по нуклеотидным последоавтельностям
Дерево максимальной экономии невозможно укоренить в точку с наименьшей суммой расстояний, потому что, в этом методе не рассчитываются расстояния. Но такое дерево можно укоренить с помощью внешней группы, в данном случая была использована последовательность энолазы сенной палочки. Последоавтельность была добавлена в файл. Аналогично предыдущему задания было получено выравнивание. На данных выравнивания было построено дерево методом максимальной экономии, оно было укорененно в энолазу сенной палочки, а после из него было выделено поддерево без внешней группы.
Для семейства энолаз был проведен бутстрэп-анализ: при построение методом UGPMA дерева пункт "Test of Phylogeny" был изменен на @Bootstrap method". В результате были получены оригинальное дерево и консенусное дерево. Они полностью совпадают между собой и с изначальным деревом.
Для сравнения деревьев было дополнительно построено дерево, основанное на последовательностях 16S РНК данных бактерий. Последовательности содержатся в файле. Выровненные mucsle послдовательности содеражатся в файле. Дерево построено методом наибольшего правдоподобия и содержится в файле.