Построение профиля семейства белков и проверка его работы
Рассматриваемое семейство доменов - суперсемейство фосфорилаз из 13 практикума второго семестра. В качестве доменной архитектуры была выбрана архитектура из последовательно идущих UDP_PNP_1 и NACHT доменов. Поиск белков с данной архитектурой выполнен запросом в Uniprot, так были получены 304 записи о белках. Таблица(лист "Architecture") с расширенными столбцами была скачена, в ней же была построена гистограмма длин белков. Средняя длина составила примерно 600-1000 аминокислотных остатков. Изображение гистограммы представлено на рисунке 1.
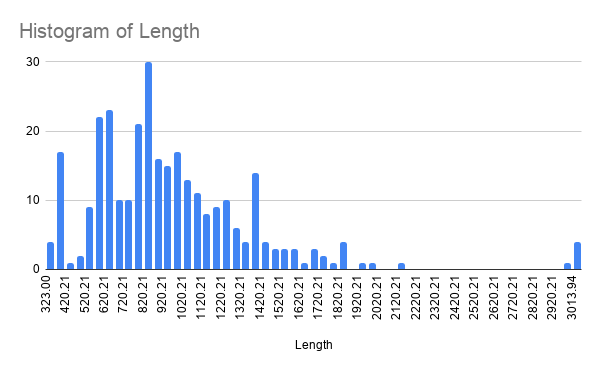
Выделение белков необходимой длины произведено с помощью базовой функции Google Sheets "IF", а также с помощью фильтра. Белки с длинами в заданном интервале происходят из следующих семейств:
43 Aspergillaceae
31 Nectriaceae
9 -
8 Glomerellaceae
7 Hypocreaceae
5 Clavicipitaceae
5 Orbiliaceae
5 Saccotheciaceae
4 Elsinoaceae
4 Phaeotrichaceae
3 Botryosphaeriaceae
3 Herpotrichiellaceae
3 Zopfiaceae
2 Pleosporaceae
2 Trichocomaceae
2 Trypetheliaceae
1 Ajellomycetaceae
1 Ascobolaceae
1 Aulographaceae
1 Cordycipitaceae
1 Cucurbitariaceae
1 Dissoconiaceae
1 Hyaloscyphaceae
1 Microcoleaceae
1 Mytilinidiaceae
1 Nitrospiraceae
1 Plexauridae
1 Pseudonocardiaceae
1 Pyriculariaceae
1 Rutstroemiaceae
1 Thermoascaceae
1 Trematosphaeriaceae
1 Xylariaceae
В основном - это грибы, часто аскомицеты. Некоторые белки без указанного семейства, полный список идентификаторов для скачивания включает 153 строки и находится по ссылке. Для скачивания последовательностей был написан и исполнен скрипт на bash. В результате были получены последовательности. Послдеовательности были выровнены muscle с параметрами по умолчанию:
muscle -in cb.fasta -out al.fasta
Множетсвенное выравнивание было открыто JalView, покрашено по Blosum62 и очищено до предполагаемого N-концевого консервативного блока примерно на 220 позиций. В итоге для построения профиля был получен файл.
HMM профиль создан с помощью команды hmm2build с опцией -g (глобальные выравнивания) и откалиброван командой hmm2calibrate:
hmm2build -g pfl.hmm alc.fasta
hmm2calibrate pfl.hmm
В результате был получен файл HMM профиля.
Последовательности всех белков с заданной доменной архитектурой были получены аналогичным способом: получением списка AC и скриптом на bash, который реализовывал команду seqret и дописывал в файл all.fasta.
Поиск осуществлен командой hmm2search с опцией --domE в 0.1, чтобы установить порог E-value в 0.1 (как указано в презентации и табюлице). Команда поиска:
hmm2search --domE 0.1 pfl.hmm all2.fasta > outs3.txt
Полученный список находок.
Поиск по белкам, содержащим домен NACHT с помощью HMM-профиля был скопирован в таблицу(Лист "Check up copy"). Для него были построен график весов, посчитаны специфичность и чувствительность, построены ROC-кривая и таблица таблица предсказания. Графики представлены ниже.
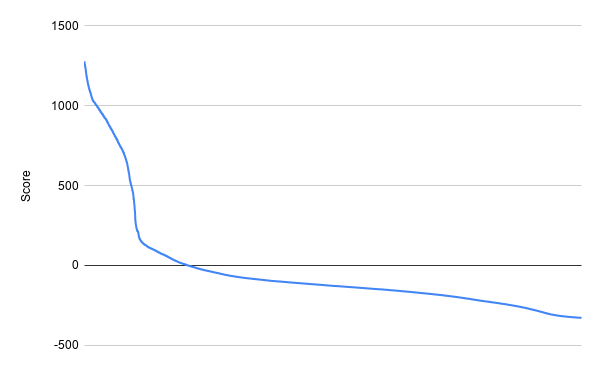
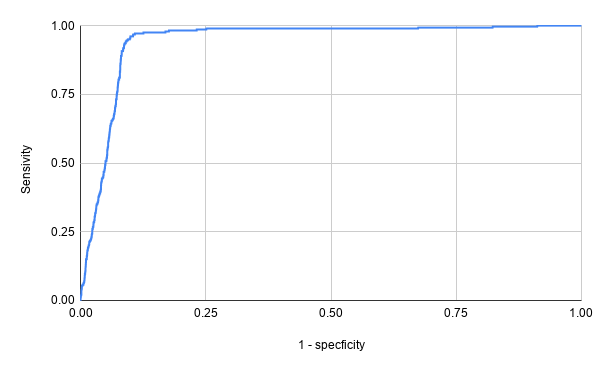
Одна из наиболее близких к правому верхнему углу кривой точек находится в 2352 строке (что примерно соответствует второму падению на графике весов). Для нее была построена таблица предсказания.
| Row 2352 | PFAM+ | PFAM- |
| HMM+ | 266 | 19494 |
| HMM- | 2082 | 14 |
Применение точного теста Фишера указало на p-value таблицы меньшее, чем 1e-5, что говорит о том, что профиль семейства относительно точен.