Часть 1. GO Enrichment Analysis
1. Анализ был проведен для биологических путей из файла list29.txt. В нем 15 ID.
2. В анализе обогащения участвовали 14 из 15 ID, вероятно, из-за того, что один содержал множественное картирование (GO Enrichment Analysis), при этом один был некартированным (Unmapped IDs).
3. Среди GO terms, удовлетворяющих условию на поправленное p-value (FDR P < 0.05) 89 находок. Их количество совпадает с количеством строк файла выдачи за вычетом первых 7 заголовочных строк.
4. Десять наиболее значимых GO terms в порядке убывания:
- liposaccharide metabolic process
- glycolipid metabolic process
- oligosaccharide metabolic process
- membrane lipid metabolic process
- carbohydrate derivative metabolic process
- carbohydrate metabolic process
- glycosphingolipid metabolic process
- glycoprotein metabolic process
- carbohydrate derivative biosynthetic process
- sphingolipid metabolic process
5. Изображения графа для пяти наиболее значимых GO terms представлено ниже.
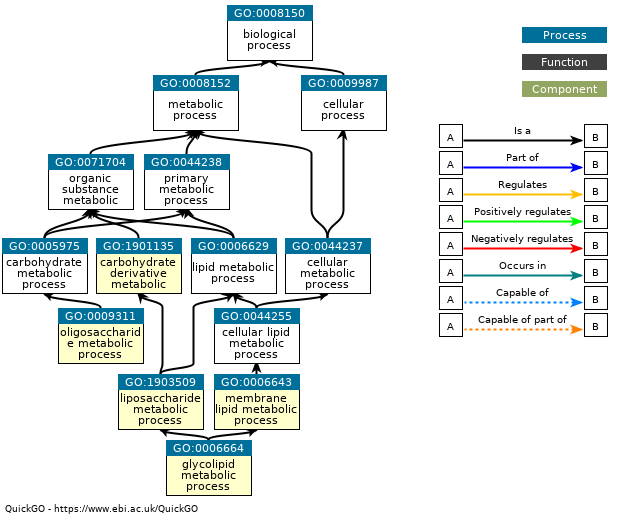
6. Граф представляет собой иерархическое дерево биологических процессов (корень), данные GO terms закрашены желтым цветом. Все вершины графа связаны отношениями включения, в частности "A is a B".
7. Ближайший предок всех 5 GO terms - метаболизм органических веществ, но это крупная категория. Поэтому были добавлены еще пять категорий. В полученном графе 7 из 10 terms являются частью метаболизма производных углеводов, еще одно близко к этому, остальные два приходятся на метаболизм липидов. В итоге ID из списка предположительно относятся к метаболизму производных углеводов.
Часть 2. String
1. Для данных ID также был пострен граф с помощью веб-сервиса String. Его изображение представлено ниже.
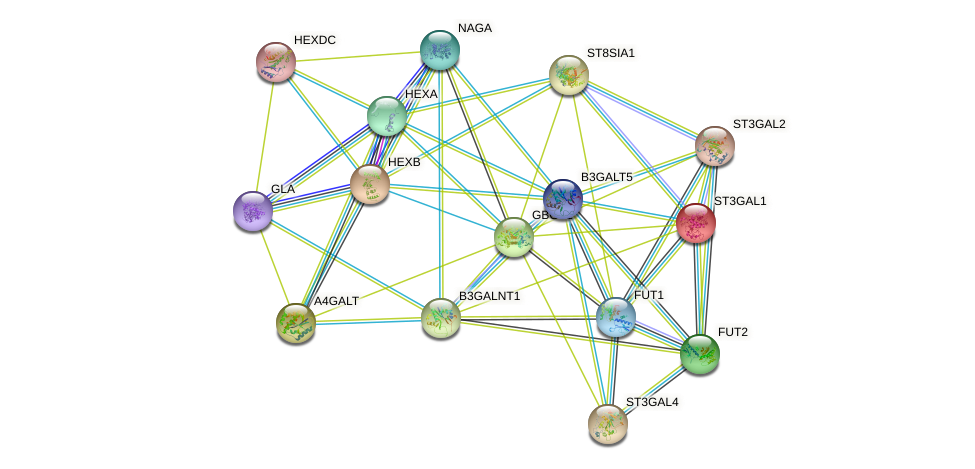
2. Так как в каждой вершине графа содержится уменьшенное изображение 3D-структуры, для всех идентификаторв существует известная или предсказанная 3D-структураю
3. В исходном графе нет только отношений соседства генов и слияния генов (gene neighboorhood, gene fusion). При двух использовании клавиши "More" появлется gene fusion и gene neighboorhood.
4. Все гены консервативны для человека, с увеличением уровня таксона консервативность уменьшается. Дерево из раздела Gene Cooccurrence представлено ниже.
5. Для человека лучше всего коэксперссируются гены FUT3 и FUT5. Для остальных организмов существует множество других пар коэкспессии генов, для мыши и рыбки данио дополнительн выделяется пара генов NAGA и GM2A. График коэкспрессии представлен ниже.
Часть 3. Human Protein Atlas
1. Анализ был проведен для ID NAGA. Его summary находится по ссылке.
2. NAGA кодирует лизосомальный фермент альфа-N-ацетилгалактозаминидазу, который отщепляет альфа-N-ацетилгалактозаминил молекулы от глюкоконъюгатов.
3. Фермент неспецифичен для конкретных регионов головного мозга (Low region specificity). Ниже представлено изображение экспрессии РНК в мозге человека.
4. Для ID предсказано внутриклеточное расположение. РНК к белку в клетке человека распределена по всей клетке, но точное местоположение белка не установлено (вся клетка закрашена серым). Изображение и легенда представлены ниже.
5. Больше всего белка найдено в поджелудочной железе, коже и специфичных для пола тканях, фермент не найден только в глазах и крови. Экспрессия РНК и белка отличаются, например, в поджелудочной железе экспрессия белка больше экспрессии РНК, а в мышечных клетках - наоборот. Изображение уровней экспрессии РНК и белка в разных тканях человека представлено ниже.
6. РНК синтезируется в примерно одинаковых объемах для всех типов тканей, но в отдельных типах клеток: моноцитах, дендритных и плацентарных, наблюдается более высокий уровень экспресии РНК. Столбчатая диаграмма экспрессии РНК представлена ниже.